(2: 中国科学院大学, 北京 100049)
(3: 华中农业大学, 武汉 430070)
(2: University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
(3: Huazhong Agricultural University, Wuhan 430070, P. R. China)
由于水体富营养化的加剧,蓝藻水华成为世界水体所面临的共同威胁[1-2].微囊藻是富营养化湖泊蓝藻水华常见优势属[3-4].微囊藻水华聚集腐烂会消耗水体中大量的氧气,恶化水质(如pH值下降、营养盐升高),引起水生生物死亡[2, 5].蓝藻水华堆积腐败过程还会产生恶臭异味,甚至导致诱发黑臭水体“湖泛”的产生[6-7].这些不仅影响环境美观,还使水质安全面临新的威胁.
对微囊藻水华腐败过程中物质循环转化进行研究能帮助我们更好地应对微囊藻水华对水体造成的威胁.在富营养湖泊中,细菌,尤其是非自养细菌,在水体的物质循环和能量转化中起到至关重要的作用[8-10].因此,了解微囊藻水华腐败过程中细菌群落组成的响应能为我们进一步了解其在湖泊物质循环中的作用提供理论基础.有研究运用原位围隔,向其中添加蓝藻,模拟蓝藻水华的发生,并研究围隔中真核微生物群落和细菌群落短期内对蓝藻腐败分解过程的响应[5, 11].Shao等[12]利用围隔实验研究了附着细菌群落对微囊藻水华降解的响应.尽管相关研究证实微囊藻水华会显著影响水体微生物的组成[13-15],但是仍然缺乏对微囊藻分解完整过程中细菌群落组成特征的研究.
太湖是我国典型的富营养化浅水湖泊,每年夏季都暴发严重的微囊藻水华[16].大量蓝藻水华在东南季风或者水生植物阻留的情况下在西部或北部岸边发生聚集腐败[16].在太湖梅梁湾设置原位围隔,通过添加大量微囊藻模拟蓝藻聚集腐败的过程,进而研究细菌群落组成对蓝藻聚集腐败的的响应和此过程中特殊细菌类群的变化.
1 材料与方法 1.1 实验设计于2013年8月21-31日在太湖梅梁湾沿岸进行原位围隔实验.实验期间天气情况为:8月21-25日有断续小雨,微风;8月26日大雨;8月27-31日晴,微风.共设6个围隔(2.0m×2.5m),位于梅梁湾沿岸,平均水深0.8m.实验前一天从梅梁湖孟湾藻水分离站收集微囊藻浆.8月21日进行围隔水体本底理化指标测定(第0d),8月22日向围隔中添加微囊藻,添加后立即进行现场指标测定和样品采集.实验共设2个处理,C组(对照组)围隔不添加藻浆,H组(实验组)添加藻浆后叶绿素a(Chl.a)浓度达378μg/L,每组设置3个平行,实验持续10d.
1.2 生物样品采集实验期间每天采样监测(其中,第5d即8月26日,因下大雨未采样),水样取自表层水以下0.5m.100~200ml水样先经5μm孔径滤膜(47mm直径,Millipore,Germany)过滤,然后过滤于0.2μm孔径滤膜上,分别收集浮游细菌与附着细菌.滤膜保存于-80℃冰箱直至分析使用.采集1L水样用1 %鲁哥试剂固定,用于浮游植物定性定量分析.
1.3 理化指标测定溶解氧(DO)、pH值、氧化还原电位(Eh)、电导率(Cond)、温度(T)、透明度(SD)、浊度(Turbidity)和总悬浮固体总量(TDS)使用多参数水质分析仪(Horiba U53,Japan)原位测定,化学指标总氮(TN)、总溶解性氮(TDN)、铵态氮(NH4+-N)、硝态氮(NO3--N)、亚硝酸盐(NO2--N)、总磷(TP)、磷酸盐(PO43--P)、总溶解性磷(TDP)、Chl.a、总有机碳(TOC)、溶解性有机碳(DOC)浓度和化学需氧量(COD)采样后送回实验室,基于标准方法[17]立刻测定.
1.4 浮游植物鉴定及计数浮游植物样品沉淀48h后,吸取上清液,浓缩至50ml保存.鉴定前,充分摇匀,取0.1ml至计数框,在10×40倍视野下进行种类鉴定和计数.微囊藻群体经超声分离成单细胞后再计数.浮游植物种类的鉴定主要参照文献[18],浮游植物生物量采用体积法进行估算,假定1mm3的浮游植物体积相当于1mg浮游植物鲜重.
1.5 细菌群落结构分析 1.5.1 DNA提取及PCR扩增方法参照细菌提取试剂盒(Omega,USA)的标准程序提取细菌的基因组DNA,之后使用带40 bp GC夹的细菌特异性引物357F(5’-CCTACGGGAGGCAGCAG-3’)和通用引物518R(5’-ATTACCGCGGCTGCTGG-3’)[19]扩增模板DNA.采用50μl PCR体系,降落PCR方法,程序如下:94℃预变性5min;94℃变性1min,65℃退火1min,然后每个循环降低1℃进行10个循环;72℃延伸1min;55℃退火进行20个循环,72℃延伸10min.用1.2 %的琼脂糖凝胶电泳检测PCR产物量及扩增特异性.
1.5.2 DGGE及克隆测序基因组DNA扩增产物通过DGGE(Dcode system,BioRad)进行变性梯度凝胶电泳(8 % (w/v)聚丙烯酰胺凝胶(丙烯酰胺:双丙烯酰胺=37.5 :1)),分析微生物群落多样性.聚丙烯酰胺的变性梯度范围为40 % ~60 %,缓冲液为1×TAE,电泳参数为:60℃,150V,7h.电泳结束后用核酸染料GelRed(用1×TAE进行1 :10000稀释)避光染色30min,然后用UV成像系统(Gene,Hongkong)成像、拍照.
对感兴趣的条带进行切胶测序.首先切胶回收DNA,以此为模板使用不带GC夹的引物进行扩增,采用纯化试剂盒(Omega,USA)对PCR产物进行纯化回收,之后连接到pMD18-T载体(Takara,Japan)质粒上.连接产物转化入感受态DH5α大肠杆菌,之后挑选阳性克隆进行测序.利用BLAST和FASTA进行序列信息比对.本研究得到的序列提交至DDBJ,序列号为LC018357~LC018398.
1.6 统计分析DGGE图谱聚类分析使用NTSYS软件(版本2.10.e).通过记下每个条带的有无(分别标记为1和0)建立二元矩阵.样本之间的相似性系数计算公式为:SD=(2NAB)/(NA+NB),其中NAB代表样本A和B的公共条带数,NA和NB分别代表样本A和B的条带数.根据相似性系数,利用非加权组平均法(UPGMA)构建系统树图(SHAN法则).
软件CANOCO 4.5(Microcomputer,Ithaca,New York,USA)用来分析细菌群落组成与环境变量的关系.当趋势对应分析(DCA)的第1长轴<2时,采用冗余分析(RDA);当第1长轴>2时,采用典范对应分析(CCA)[20-21].采用蒙特卡罗置换检验筛选与细菌群落组呈显著相关的环境变量.
环境变量的统计分析均使用SPSS软件(版本13.0),显著水平设定为0.05(P<0.05).
2 结果与分析 2.1 环境变量分析实验期间主要环境参数的变化如图 1所示.添加蓝藻后,H组围隔中DO浓度迅速降低,而第5d(大雨)之后,DO浓度逐渐回升(图 1b),与DO密切相关的Eh具有相似的变化规律,从第7d开始回升(图 1c).添加蓝藻后的1~4d,H组COD浓度迅速升高并显著高于对照组(P<0.05),第5d后COD值缓慢下降,实验后期与C组的COD值无显著差异(P>0.05)(图 1d).在1~4d,H组的pH值低于C组(图 1e),之后逐渐升高并高于C组.在添加蓝藻后的整个实验期间,H组围隔中TN和TP浓度显著高于C组围隔(P<0.05)(图 1f和g),NO3--N的平均浓度也高于C组(图 1h).然而,NH4+-N、TDN和TDP浓度,在第0~3d,H组与C组中无显著差异(P>0.05)(图 1i、j和k),之后在H组围隔中迅速升高,采样后期显著高于C组(P<0.05).H组围隔中TOC浓度在添加微囊藻后迅速升高,第1~4d明显高于C组(P<0.05),而第5d(大雨)之后,H组TOC浓度逐渐下降,直至实验结束时,且与C组值无显著差异(P>0.05)(图 1l).
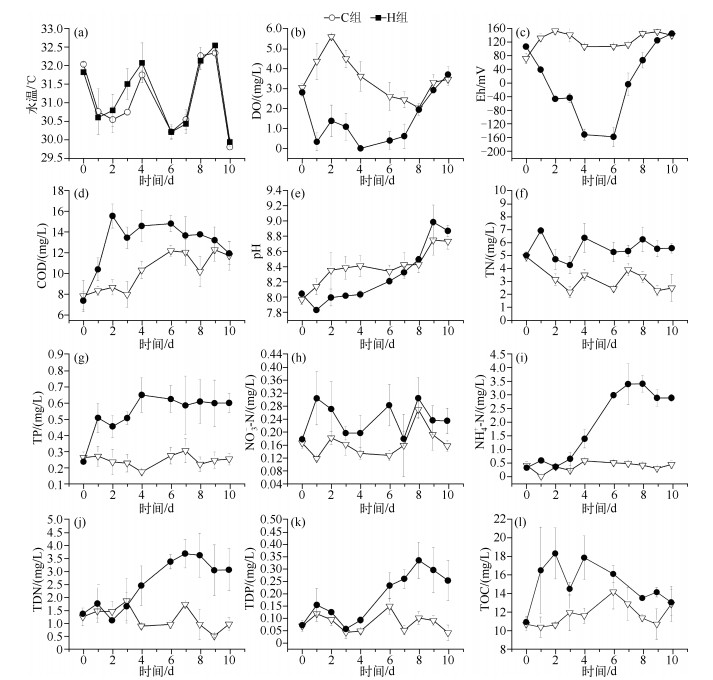
|
图 1 实验期间主要环境因子的变化 Fig.1 Temporal variations in the main environmental factors during the course of experiment |
在第1d添加微囊藻后,H组围隔浮游植物生物量达到60.84mg/L,Chl.a浓度达到378.1μg/L,其中微囊藻生物量为58.58mg/L,占浮游植物总生物量的95.7 %,H组围隔中微囊藻生物量相当于严重水华水平(图 2).H组在添加微囊藻后,1~5d内浮游植物生物量不断下降,第6d直到实验结束,H组围隔的藻类生物量与C组无显著差异(图 2a),微囊藻生物量的变化规律相似(图 2b);而H组围隔Chl.a浓度则在添加藻后的第2d就迅速下降到与对照组相近的水平(图 2c),这表明H组围隔中的微囊藻在1~4d发生快速厌氧死亡.
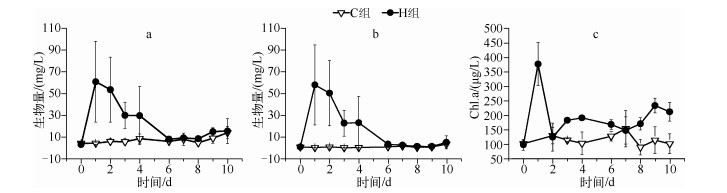
|
图 2 浮游植物生物量(a)、微囊藻生物量(b)和Chl.a浓度(c)随时间的变化 Fig.2 Temporal variations in biomass of phytoplankton (a), biomass of Microcystis spp.(b)and Chl.a concentration (c) |
DGGE图谱显示了两组围隔中浮游细菌与附着细菌群落组成的变化(图 3a、b).聚类分析结果(图 3c、d)显示,浮游细菌的群落组成可聚为两大类:其中C组所有样品与添加微囊藻前和刚添加微囊藻后的H0和H1聚为一大类,而剩下的H组样品聚为另一大类.其中H2单独聚为一类,H3和H4聚为一类,第5d之后的样品(H6~H10)聚为一类;而C组中0~6d的样品与H0和H1聚为一类,7~10d的样品聚为一类(图 3c).附着细菌的群落组成可聚为4类:H组添加微囊藻之前的样品H0与C组0~4d的样品(C0~C4)聚为一类,C组6~10 d的样品与H组7~10 d的样品聚为一类,H组中添加微囊藻后1~6 d的样品聚为一类(图 3d).
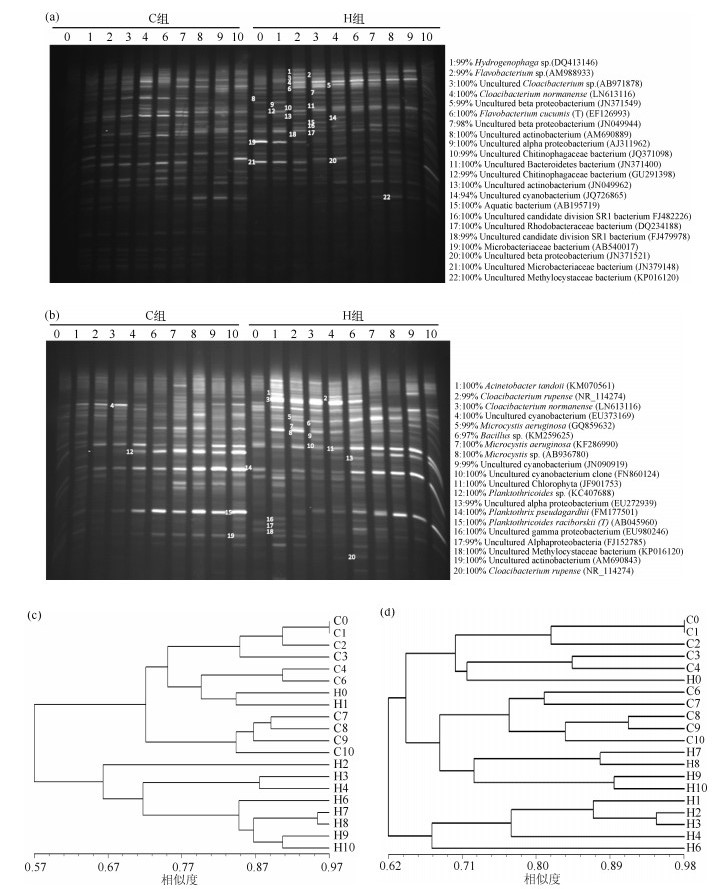
|
图 3 DGGE图谱:浮游细菌群落(a)和附着细菌群落(b);UPGMA聚类分析结果:浮游细菌群落(c)和附着细菌群落(d) Fig.3 DGGE profiles of free-living bacterial communities composition (a) and particle-attached bacterial communities composition(b); Cluster analysis of free-living bacterial community composition (c)and particle-attached bacterial community composition(d) based on DGGE profiles |
图 3a和图 3b所示编号为已进行回收测序的条带,与条带序列同源性最高的序列信息列于右侧.在浮游细菌群落中,H组出现多个隶属于黄杆菌科(Flavobacteriaceae)(拟杆菌门)的条带(条带2、3、4、6),且明显占优势地位,而这些条带未在C组中出现.特别是条带4(隶属于黄杆菌科,与培养类群Cloacibacterium nor-manense的相似度为100 %),在H组添加微囊藻后第2~10d的样品中均占显著优势.与条带4隶属于相同属的分类单元条带3出现在H1中,条带2出现在H组2~10d的样品中,隶属于黄杆菌属Flavobacterium(相似度99 %),而隶属于同一属的条带6(与培养类群Flavobacterium cucumis (T)的相似度为100 %)在H组1~2d的样品中占据优势地位.而在H组2~3d的样品中占明显优势的分类单元条带5隶属于β-变形菌门(Betaproteobacteria).而放线菌门(Actinobacteria)是C组样品中的一个重要门类,在C组样品中占明显优势,如条带8、13、21.在附着细菌群落中,所测条带序列有一半隶属于蓝藻门,由于本研究的重点关注细菌,蓝藻序列在此不做讨论.在附着细菌中,我们发现,条带3与浮游细菌群落中条带4序列相同(与Cloacibacterium normanense的相似度为100 %),且在H组第2~4d的样品中占绝对优势.
基于趋势对应分析第一长轴(DCA)<2,利用RDA模型来寻找细菌群落组成与环境因子之间的相互关系.RDA的结果(图 4a)显示, 浮游细菌的群落组成与水体Eh、pH值、TP浓度、浮游植物生物量以及NO3--N浓度呈显著相关(P<0.05),两轴共可解释浮游细菌群落组成变化的44.55 %.其中,这5个环境因子依次解释了浮游细菌群落组成变化的20.0 %、15.2 %、11.1 %、7.1 %和6.2 %.附着细菌群落组成与pH值、TP浓度、DO浓度以及浮游植物生物量呈显著相关(P<0.05),两轴共解释附着细菌群落组成变化的35 %.pH值、TP浓度、DO浓度和浮游植物生物量分别解释了附着细菌群落组成的17.9 %、14.0 %、9.7 %和7.2 % (图 4b).图 4显示,浮游细菌样品聚为3类:第1类:C组样品与H0和H1;第2类:H组加藻后2~4d的样品;第3类,H组6~10的样品.附着细菌样品聚为4类:第1类:C组0~4d样品和H0;第2类:C组6~10d的样品;第3类:H组1~6d样品;第4类,H组7~10d样品.由此可见,RDA排序模型结果与DGGE的UPGMA聚类分析结果相似(图 3c、d).
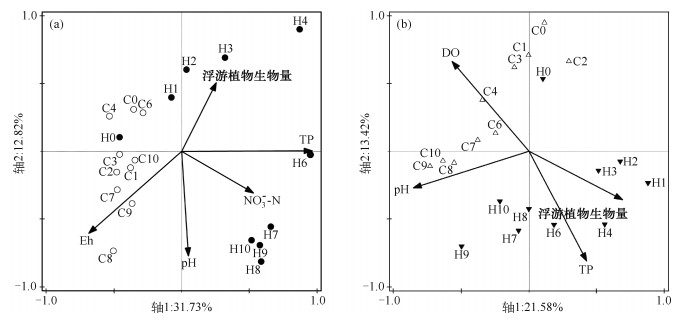
|
图 4 基于DGGE和环境变量的RDA分析结果:浮游细菌群落(a);附着细菌群落(b) Fig.4 Biplots diagram of the redundancy analysis (RDA) on free-living bacterial community (a), particle-attached bacterial community (b) (DGGE profiles of 16S rRNA genes)constrained by environmental variables |
根据围隔水体理化指标的监测结果(图 1和图 2),添加微囊藻水华后1~4d,DO浓度迅速降低,水体成为还原性的环境,而微囊藻生物量的下降,说明水体中微囊藻水华正处于厌氧分解的过程,从而导致水体中化学需氧量升高,同时水体中pH值下降,营养盐浓度升高(如TN、TP浓度).添加微囊藻水华后6~10d实验期间,在第5d大雨后,DO浓度和Eh迅速回升,水体逐渐转变为氧化性环境,但是H组围隔的NH4+-N、TDN和TDP浓度在这个阶段显著高于C组,而TOC浓度在这个阶段开始逐渐下降,说明微囊藻厌氧分解积累的有机质开始消耗,使水体中NH4+-N、TDN和TDP浓度在整个实验期间逐渐升高.而TN、TP浓度在整个实验期间都显著高于对照组.
有研究表明,当水体中出现蓝藻水华堆积时,其厌氧分解过程便占主导地位,耗竭水体中的氧气,危害其他生物生存[16].而微生物厌氧分解蓝藻的过程会产生大量有机酸和CO2,使水体pH值降低[5].蓝藻细胞内的N、P伴随着其厌氧分解大量释放到水中[22],从本研究中可看出,整个实验期间H组的TN、TP浓度都显著高于对照组,至厌氧蓝藻分解后仍持续维持较高的水平,此结果说明水体富营养化与蓝藻水华暴发之间的物质循环过程是值得关注的科学问题.
3.2 细菌群落组成对蓝藻水华分解的响应已有的研究表明,富营养化湖泊中细菌群落组成与蓝藻水华密切相关[13-15].Li等[11]利用原位围隔实验,研究了细菌群落组成对微囊藻聚集和降解的响应,报道了添加微囊藻到厌氧分解开始期间,细菌群落的短期动态响应.本研究通过DGGE技术分析了浮游细菌和附着细菌群落组成对蓝藻水华分解整个过程的响应.聚类分析结果发现,添加了微囊藻的围隔与未添加微囊藻的围隔中,浮游细菌群落组成存在显著区别,而H1与C组样品聚成一类,这可能与H1为刚添加完微囊藻后就采集样品有关,细菌群落组成对环境的响应需要时间.附着细菌群落组成在添加微囊藻后1~6d,与对照组有显著区别,这期间微囊藻水华堆积并发生厌氧分解,生物量不断降低;而之后H组(7~10d)附着细菌群落组成与对照组聚为一类,表明微囊藻水华厌氧分解结束后,附着细菌群落组成发生了恢复.然而,实验期间浮游细菌群落组成未出现类似的变化过程,这可能是因为尽管厌氧分解过程结束,但围隔水体中大部分理化因子(如TN、TP、NH4+-N浓度等)仍未恢复.由此可以看出,浮游细菌群落组成与附着细菌群落组成的影响因素存在区别.
3.3 微囊藻水华分解过程中影响细菌群落组成的重要理化因子根据已有的研究,很多环境因子,如温度[23]、pH值[24]、原生动物[25-26]、浮游植物[14, 27-28]、营养盐[29]和水生植物[30]都被证实是细菌群落组成的影响因子.RDA排序模型结果表明,在微囊藻水华分解的微环境过程中,Eh、pH值、TP浓度、浮游植物生物量以及NO3--N浓度与浮游细菌群落组成密切相关,而附着细菌群落组成则与pH值、TP浓度、DO浓度以及浮游植物生物量呈显著相关.微囊藻水华的分解使得水体变为还原性的环境(表现为低的DO浓度和Eh值),pH值降低,促进N、P营养盐的释放[22].而Eh和DO浓度是两个密切相关的理化因子,反映了水体的氧化还原状况,所以在微囊藻厌氧分解过程中水体的氧化还原状况、pH值、营养盐(N、P)浓度、浮游植物生物量与细菌群落组成密切相关.已有相关研究证实微囊藻水华会显著影响水体的BCC[13-15],尤其是其聚集与分解过程[11-12].有机质的来源及种类浓度变化能显著影响湖泊中的BCC组成及活性[31-33].在富营养化湖泊中,浮游植物是水体TOC浓度的重要来源[34].微囊藻水华的分解使水体TOC浓度升高,为水体细菌提高复杂多样的有机质来源,从而改变细菌的群落组成特征.简而言之,微囊藻水华可直接或间接地影响水体细菌群落组成.
3.4 参与微囊藻水华分解过程的重要细菌群落目前,有多个细菌类群被认为与微囊藻水华分解有关.Li等[11]通过蓝藻堆积的围隔模拟实验研究细菌的早期响应,发现微球菌亚目可能与微囊藻降解有关.Xing等[35]在室内实验中发现梭菌属类群在微囊藻的厌氧分解中起着重要作用.而在本研究中最引起关注的是隶属于黄杆菌科(拟杆菌门)的一个类群(浮游细菌条带4和附着细菌条带3,与培养类群Cloacibacteriu normanense的相似度为100 %),这个类群在微囊藻厌氧分解阶断占显著优势.通过同源性比对发现,与其相似性100 %的类群多出现在污水或厌氧污泥中,而Cloacibacterium normanense最早也是从污水中分离鉴定而来的,属于兼性厌氧菌[36].对这类细菌进一步的分离、培养及其生态生理特性的研究将有助于阐明其在微囊藻降解过程中的确切作用.
4 结论本研究通过原位围隔实验模拟微囊藻水华分解过程,研究了此过程中细菌群落组成的动态变化.结果表明,细菌群落组成随着微囊藻分解的不同阶段发生显著变化.微囊藻厌氧分解过程伴随着水体氧化还原环境的改变(DO浓度和Eh降低),pH值、藻类生物量的下降和营养盐(N、P)释放,细菌群落组成与这些理化因子密切相关.研究中发现微囊藻分解阶段的特殊细菌群落,其中隶属于黄杆菌科(拟杆菌门)的类群在微囊藻厌氧分解阶段占显著优势,其功能有待于进一步研究.
致谢: 感谢朱荣在围隔实验采样中给予的帮助.| [1] |
Lehma EM. Seasonal occurrence and toxicity of Microcystis in impoundments of the Huron River, Michigan, USA. Water Research, 2007, 41(4): 795-802. DOI:10.1016/j.watres.2006.09.030 |
| [2] |
Paerl H, Fulton RS, Moisander PH et al. Harmful freshwater algal blooms, with an emphasis on cyanobacteria. The Scientific World Journal, 2001(1): 76-113. |
| [3] |
Chen Y, Qin B, Teubner K et al. Long-term dynamics of phytoplankton assemblages:Microcystis-domination in Lake Taihu, a large shallow lake in China. Journal of Plankton Research, 2003, 25: 445-453. DOI:10.1093/plankt/25.4.445 |
| [4] |
Conley DJ, Paerl HW, Howarth RW et al. Controlling eutrophication:nitrogen and phosphorus. Science, 2009, 323: 1014-1015. DOI:10.1126/science.1167755 |
| [5] |
Chen M, Chen F, Xing P et al. Microbial eukaryotic community in response to Microcystis spp. Bloom, as assessed by an enclosure experiment in Lake Taihu, China. FEMS Microbiology Ecology, 2010, 74: 19-31. DOI:10.1111/j.1574-6941.2010.00923.x |
| [6] |
Ma Z, Niu Y, Xie P et al. Off-flavor compounds from decaying cyanobacterial blooms of Lake Taihu. Journal of Environmental Sciences, 2013, 25: 495-501. DOI:10.1016/S1001-0742(12)60101-6 |
| [7] |
Zhang XJ, Chen C, Ding JQ et al. The 2007 water crisis in Wuxi, China:analysis of the origin. Journal of Hazardous Materials, 2010, 182: 130-135. DOI:10.1016/j.jhazmat.2010.06.006 |
| [8] |
Cole JJ, Likens GE, Strayer DL. Photosynthetically produced dissolved organic carbon:an important carbon source for planktonic bacteria. Limnology and Oceanography, 1982, 27(6): 1080-1090. DOI:10.4319/lo.1982.27.6.1080 |
| [9] |
Cole JJ, Findlay S, Pace ML. Bacterial production in fresh and saltwater ecosystems:a cross-system overview. Marine Ecology Progress, 1988, 43(1/2): 1-10. |
| [10] |
Paerl H, Pinckney J. A mini-review of microbial consortia:their roles in aquatic production and biogeochemical cycling. Microbial Ecology, 1996, 31: 225-247. |
| [11] |
Li H, Xing P, Chen M et al. Short-term bacterial community composition dynamics in response to accumulation and breakdown of Microcystis blooms. Water Research, 2011, 45: 1702-1710. DOI:10.1016/j.watres.2010.11.011 |
| [12] |
Shao K, Zhang L, Wang Y et al. The responses of the taxa composition of particle-attached bacterial community to the decomposition of Microcystis blooms. Science of the Total Environment, 2014, 488/489: 236-242. DOI:10.1016/j.scitotenv.2014.04.101 |
| [13] |
Xing P, Kong F. Intra-habitat heterogeneity of environmental factors regulating bacterioplankton community composition in Lake Taihu, China. Aquatic Microbial Ecology, 2007, 48: 113-122. DOI:10.3354/ame048113 |
| [14] |
Niu Y, Shen H, Chen J et al. Phytoplankton community succession shaping bacterioplankton community composition in Lake Taihu, China. Water Research, 2011, 45: 4169-4182. DOI:10.1016/j.watres.2011.05.022 |
| [15] |
Tian C, Tan J, Wu X et al. Spatiotemporal transition of bacterioplankton diversity in a large shallow hypertrophic freshwater lake, as determined by denaturing gradient gel electrophoresis. Journal of Plankton Research, 2009, 31: 885-897. DOI:10.1093/plankt/fbp028 |
| [16] |
Qin B, Xu P, Wu Q et al. Environmental issues of lake Taihu, China. Hydrobiologia, 2007, 581: 3-14. DOI:10.1007/s10750-006-0521-5 |
| [17] |
金相灿, 屠清瑛. 湖泊富营养化调查规范. 北京: 中国环境科学出版社, 1990.
|
| [18] |
胡鸿钧, 魏印心. 中国淡水藻类-系统, 分类及生态. 北京: 科学出版社, 2006.
|
| [19] |
Muyzer G, De Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA. Applied and Environmental Microbiology, 1993, 59: 695-700. |
| [20] |
Braak Ct, Šmilauer P. CANOCO reference manual and CanoDraw for Windows user's guide:software for canonical community ordination (version 4.5). Section on Permutation Methods. Section on Permutation Methods. Microcomputer Power, Ithaca, New York, 2002. |
| [21] |
terBraak CJ, Verdonschot PF. Canonical correspondence analysis and related multivariate methods in aquatic ecology. Aquatic Sciences, 1995, 57: 255-289. DOI:10.1007/BF00877430 |
| [22] |
Ye L, Wu X, Tan X et al. Cell lysis of cyanobacteria and its implications for nutrient dynamics. International Review of Hydrobiology, 2010, 95: 235-245. DOI:10.1002/iroh.v95:3 |
| [23] |
Shade A, Kent AD, Jones SE et al. Interannual dynamics and phenology of bacterial communities in a eutrophic lake. Limnology and Oceanography, 2007, 52: 487-494. DOI:10.4319/lo.2007.52.2.0487 |
| [24] |
Yannarell AC, Triplett EW. Geographic and environmental sources of variation in lake bacterial community composition. Applied and Environmental Microbiology, 2005, 71: 227-239. DOI:10.1128/AEM.71.1.227-239.2005 |
| [25] |
Langenheder S, Jürgens K. Regulation of bacterial biomass and community structure by metazoan and protozoan predation. Limnology and Oceanography, 2001, 46: 121-134. DOI:10.4319/lo.2001.46.1.0121 |
| [26] |
Van Hannen E, Veninga M, Bloem J et al. Genetic changes in the bacterial community structure associated with protistan grazers. Archiv für Hydrobiologie, 1999, 145: 25-38. DOI:10.1127/archiv-hydrobiol/145/1999/25 |
| [27] |
Höfle MG, Haas H, Dominik K. Seasonal dynamics of bacterioplankton community structure in a eutrophic lake as determined by 5S rRNA analysis. Applied and Environmental Microbiology, 1999, 65: 3164-3174. |
| [28] |
Rooney-Varga JN, Giewat MW, Savin MC et al. Links between phytoplankton and bacterial community dynamics in a coastal marine environment. Microbial Ecology, 2005, 49: 163-175. DOI:10.1007/s00248-003-1057-0 |
| [29] |
Berdjeb L, Ghiglione JF, Domaizon I et al. A 2-year assessment of the main environmental factors driving the free-living bacterial community structure in Lake Bourget (France). Microbial Ecology, 2011, 61: 941-954. DOI:10.1007/s00248-010-9767-6 |
| [30] |
Wu QL, Zwart G, Wu J et al. Submersed macrophytes play a key role in structuring bacterioplankton community composition in the large, shallow, subtropical Taihu Lake, China. Environmental Microbiology, 2007, 9: 2765-2774. DOI:10.1111/emi.2007.9.issue-11 |
| [31] |
Crump BC, Kling GW, Bahr M et al. Bacterioplankton Community Shifts in an Arctic Lake Correlate with Seasonal Changes in Organic Matter Source. Applied and Environmental Microbiology, 2003, 69: 2253-2268. DOI:10.1128/AEM.69.4.2253-2268.2003 |
| [32] |
Pérez MT, Sommaruga R. Differential effect of algal-and soil-derived dissolved organic matter on alpine lake bacterial community composition and activity. Limnology and Oceanography, 2006, 51: 2527-2537. DOI:10.4319/lo.2006.51.6.2527 |
| [33] |
Judd KE, Crump BC, Kling GW. Variation in dissolved organic matter controls bacterial production and community composition. Ecology, 2006, 87: 2068-2079. DOI:10.1890/0012-9658(2006)87[2068:VIDOMC]2.0.CO;2 |
| [34] |
Baines SB, Pace ML. The production of dissolved organic matter by phytoplankton and its importance to bacteria:patterns across marine and freshwater systems. Limnology and Oceanography, 1991, 36: 1078-1090. DOI:10.4319/lo.1991.36.6.1078 |
| [35] |
Xing P, Guo L, Tian W et al. Novel Clostridium populations involved in the anaerobic degradation of Microcystis blooms. Isme Journal, 2011, 5(5): 792-800. DOI:10.1038/ismej.2010.176 |
| [36] |
Allen TD, Lawson PA, Collins MD et al. Cloacibacterium normanensegen. nov., sp. Nov., a novel bacterium in the family Flavobacteriaceae isolated from municipal wastewater. International Journal of Systematic and Evolutionary Microbiology, 2006, 56: 1311-1316. DOI:10.1099/ijs.0.64218-0 |
 2016, Vol. 28
2016, Vol. 28 

