(2: 临沂大学资源环境学院, 临沂 276000)
(2: College of Resource and Environment, Linyi University, Linyi 276000, P.R.China)
附着细菌是指生长在植物表面并与植物形成复杂共生关系的一类细菌[1].在湖泊生态系统中,附着细菌不仅数量众多,而且种类十分丰富.附着细菌作为湖泊生态系统中重要组成部分,对有机物的降解以及营养物质循环过程都起着重要作用[2-5].沉水植物附着细菌的数量通常可以达到105~107 cells/cm2[6].相比浮游细菌,附着细菌具有更高的初级生产力[7].在附着细菌及其宿主沉水植物的共同作用下,湖泊水体的水质情况能够得到显著改善.因此研究附着细菌对于解决湖泊生态系统的各种问题有重要意义[8-9].
沉水植物附着细菌主要包括拟杆菌(Bacteroidetes)、放线菌(Actinobacteria)、变形菌(Proteobacteria)、蓝藻细菌(Cyanobacteria)和疣微菌(Verrucomicrobia)等门类[1].优势菌群在附着细菌生物膜形成过程中发挥着重要作用,生境条件和植物本身都会对附着细菌的群落结构造成影响[10].早期对附着藻类宿主专一性的研究较多,而对沉水植物附着细菌的研究相对比较匮乏[11].何聃等[12]对苦草(Vallisneria natans)和轮叶黑藻(Hydrilla verticillata)附着细菌多样性进行研究,认为宿主植物对附着细菌有很大影响. Cai[10]对太湖不同地点马来眼子菜附着细菌群落结构特征进行研究,发现不同取样点马来眼子菜附着细菌组成具有高度的相似性,并推断其也具有一定的宿主选择. Hempel等[6]对轮藻(Chara aspera)和狐尾藻(Myriophyllum verticillatum)附着细菌的研究认为,植物体分泌出不同的多酚类物质对表面附着细菌有抑制作用,其中拟杆菌门的细菌丰度受这类物质含量的影响非常明显.本研究采集洪泽湖3种典型沉水植物各自生长旺盛期的植株,利用T-RFLP技术对其叶表面附着细菌的群落多样性进行研究,目的是为了探讨不同植物附着细菌群落结构差异及环境因子对附着细菌群落结构的影响,为深入研究富营养化湖泊植物-微生物结构功能关系提供支撑.
1 研究区概况及研究方法 1.1 研究区概况洪泽湖(33°6′~33°40′N,118°10′~118°52′E)水域面积约1597 km2,平均水深1.9 m,是我国第4大淡水湖泊,湖区水生植物比较丰富,水质较好[13-14].但受人类活动的影响,近年来部分湖区水生植被出现退化趋势[15].选择洪泽湖西部溧河洼湖湾为研究区域,该湖区水生植物种类丰富、盖度较高,但受围网围垦等因素影响,水生植物分布及水质的空间差异较大.本研究选取洪泽湖3种典型的沉水植物,菹草(Potamogeton crispus)、篦齿眼子菜(Potamogeton pectinatus)和金鱼藻(Ceratophyllum demersum)作为研究对象.
1.2 样品采集及水质测定于2014年4、5和7月分别采集菹草、篦齿眼子菜和金鱼藻各自生长旺盛期的植物样品[16].为了使取样更具代表性,每种沉水植物在不超过5 m范围内随机采集3个样品,编号分别为1、2、3.小心剪取长势比较一致的沉水植物枝叶500 g左右,立即放入无菌的聚乙烯袋中,用冰袋保温带回实验室测定附着细菌.使用HACH HQ30D便携式检测仪现场测定水体pH值、溶解氧(DO)浓度、水温(WT)等水质指标,并且按照规范采集水样[17],运回实验室测定相关水质指标.用AutoAnalyzer 3全自动水质连续流动分析仪(德国SEAL公司)测定水体总氮(TN)、总磷(TP)、氨氮(NH3-N)浓度等.叶绿素a浓度(Chl.a)用90 %的丙酮溶液提取,然后由比色法进行测定.
1.3 附着细菌样品处理在无菌室取出沉水植物样品,分别选取形态一致的茎叶组织鲜重40 g,使用震荡-超声波法洗脱表面附着物[1].洗脱液通过1.2 μm醋酸纤维混合膜将大颗粒物除去,滤液再通过0.22 μm醋酸纤维混合膜,滤膜保存在-20℃下.
1.4 样品总DNA提取按照E.Z.N.A.Water DNA试剂盒步骤提取样品基因组总DNA.使用NanoDrop 2000超微量紫外分光光度计检测DNA浓度和纯度. DNA样本于-20℃冻存.
1.5 T-RFLP分析使用通用引物对27F/1492R扩增16S rDNA部分片段[18-20].其中上游引物27F的5′末端使用FAM荧光标记. PCR扩增体系:模板DNA 2 μl,上下游引物各1 μl,2×EasyTaq® PCR Super Mix 25 μl(TransGenic Biotech),ddH2O补足50 μl. PCR扩增条件为:94℃ 5 min;94℃ 30 s,56℃ 30 s,72℃ 1 min,35个循环;72℃ 10 min. PCR产物经1 %的琼脂糖凝胶电泳检测后用DNA纯化试剂盒纯化.
用限制性内切酶MspⅠ对上述PCR产物进行酶切,反应体系为MspⅠ 0.5 μl,10×RE buffer 2 μl,BSA 0.2 μl,PCR产物20 μl,用ddH2O将反应体系补足到40 μl.混匀后在37℃酶切4 h,80℃作用20 min后停止反应,酶切产物交由上海生物工程有限公司进行限制性片段的基因扫描,由Peak Scanner Software v1.0软件分析处理获得附着细菌的T-RFLP图谱.
1.6 数据处理T-RFLP图谱采用Genemarker V2.4软件进行处理.单个T-RF (Terminal Restriction Fragment)的相对峰面积(Pi)可通过公式Pi =ni/N×100 %进行计算,其中,ni为单个T-RF的实际峰面积,N为图谱中所有峰的面积之和.本实验中Pi值仅采用片段长度在50~650 bp区间的T-RF数值进行统计计算作为各T-RF的相对丰度,相对丰度小于1 %的T-RF不予考虑.计算Shannon-Wiener多样性指数和Simpson多样性指数.通过Phylogenetic Assignment Tool数据库(https://secure.limnology.wisc.edu/trflp/)对主要T-RFs所代表的物种进行推测.
根据不同样品中T-RF的丰度大小,将物种数据进行标准化处理后,通过Canoco4.5软件进行主成分分析(PCA).为了研究群落结构与环境因子的关系,首先对物种数据进行去趋势对应分析(DCA),结果表明第一排序轴长度为1.603(小于2),因此选用基于线性模型的冗余分析(RDA).采用手动选择,找出对群落结构变化有显著影响(P<0.05)的环境因子,利用Monte Carlo permutation test检验RDA排序轴特征值的显著性.利用PRIMER软件对3种沉水植物的片段信息做一元ANOSIM统计分析,检测它们之间的相似性.
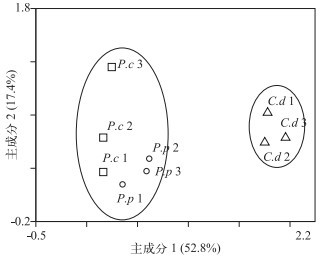
2 结果与讨论 2.1 3种沉水植物附着细菌群落结构分析不同沉水植物附着细菌群落结构之间有一定差异(图 1),结果表明,长度为91 bp的T-RF在全部样品中都被检测出,并且在其中7个样品中相对丰度超过了20 %,尤其在金鱼藻样品中,该片段的平均相对丰度达到27 %.长度为167 bp的T-RF也广泛存在于所有样品中,且在各样品中相对丰度均大于10 %,并在1个金鱼藻样品中相对丰度达到30 %.长度为143 bp的T-RF在篦齿眼子菜和菹草样品中平均相对丰度分别达到11 %和20 %,而在金鱼藻样品中相对丰度却小于1 %;89 bp的T-RF在金鱼藻样品中相对丰度平均约为13 %,而在菹草和篦齿眼子菜样品中相对丰度低于5 %;94 bp的T-RF在菹草样品中相对丰度超过15 %,而在其他样品中未被检测出;91 bp和167 bp所代表的细菌在3种沉水植物附着细菌中所占比例较高且含量相对稳定.基于样品T-RFLP数据进行主成分分析(图 2),图中点的距离越近代表样品相似性程度越高,结果表明所有样品大致可以分为2个独立的群,金鱼藻可以单独聚为一类,而篦齿眼子菜和菹草可以聚为一类,说明菹草和篦齿眼子菜的附着细菌群落组成具有一定的相似性.为进一步了解不同沉水植物附着细菌群落结构的相似性情况,利用PRIMER软件对3种植物做一元ANOSIM统计分析,R值越接近1,表明样本之间的差异越大,越倾向于分类到不同的组群内.结果显示篦齿眼子菜和金鱼藻群落组间差异最大(R=0.815,P=0.1),其次是菹草和金鱼藻群落(R=0.407,P=0.1),菹草和篦齿眼子菜群落结构差异最小(R=0.185,P=0.3).

|
图 1 洪泽湖不同沉水植物附着细菌群落结构 Fig.1 Community structure of epiphytic bacteria on different submerged macrophytes in Lake Hongze (P.c、P.p和C.d分别代表菹草、篦齿眼子菜和金鱼藻样品) (P.c, P.p, C.d represent Potamogeton crispus, Potamogeton pectinatus and Ceratophyllum demersum samples, respectively) |
|
图 2 基于T-RFLP的主成分分析 Fig.2 PCA for T-RFs of three submerged macrophytes. Ellipses show similar groups (P.c、P.p、C.d分别代表菹草、篦齿眼子菜和金鱼藻样品) (P.c, P.p, C.d represent Potamogeton crispus, Potamogeton pectinatus and Ceratophyllum demersum samples, respectively) |
通过细菌数据库进行比对,对一些优势T-RFs代表的物种进行推测.分别为Firmicutes(89 bp)、Bacteroidetes(91 bp)、γ-Proteobacteria/Actinobacteria(143 bp)、Actinobacteria/α-Proteobacteria(167 bp)等门类. Bacteroidetes在淡水湖泊生态系统中起着非常重要的作用,可以协助部分有机大分子物质分解[21]. Vander等[22]的研究表明,在以浮游藻类为主要初级生产者的浊水态湖泊中以Cyanobacteria为主,而在以高等水生植物为初级生产者的清水态湖泊中则以Bacteroidetes居多,这与洪泽湖实际情况相符.而Proteobacteria广泛分布于全球不同类型的湖泊中,数量极为丰富,这类细菌往往有显著的固氮作用.
对3种沉水植物附着细菌群落结构差异的原因进行探究.首先,菹草和篦齿眼子菜均属于眼子菜科,而金鱼藻属于金鱼藻科. 3种沉水植物的叶片形态差异极大,篦齿眼子菜的叶片为线形;菹草的叶片为线状披针形,边缘呈浅波状,有细小锯齿;金鱼藻叶片为轮生,边缘有散生的刺状细齿[16].不同的叶形为细菌提供了不同的附着基质.水体中的浮游细菌经过一段时间选择与适应,最终在植物体表形成了稳定的菌落[23].其次,在其生长过程中,植物内生细菌也有可能会通过组织缝隙、表面裂口等物理通道或随植物的代谢产物一并迁移到植物叶表,并且最终各自形成独特的细菌群落结构[1].此外,附着细菌的生长繁殖与周围水体环境密切相关,营养盐负荷和特殊的生境条件等都会影响附着细菌的种类和丰度.菹草群丛和篦齿眼子菜群丛的营养盐比较接近(表 1),很可能是引起2种植物附着细菌群落结构更为接近的主要原因.
| 表 1 3种沉水植物样品周围水体的主要理化特征 Tab.1 Chemical and physical characteristics of water surrounding submerged macrophytes |
菹草、篦齿眼子菜和金鱼藻附着细菌的T-RFs平均数目分别约为44、49和35个. 3种沉水植物附着细菌多样性指数变化与T-RFs数量变化基本一致.篦齿眼子菜附着细菌拥有较高的物种多样性,其次是菹草,金鱼藻附着细菌的多样性指数最低. Shannon-Wiener指数和Simpson指数的趋势呈现一致性, 均匀度指数则基本保持不变(表 2).
| 表 2 3种沉水植物附着细菌多样性指数 Tab.2 Diversity index of epiphytic bacteria on three submerged macrophytes |
国外报道过沉水植物与附着细菌发生相互作用时会分泌出一些有机化合物,主要是植物次生代谢物(如酚类),能够对其他光养生物或微生物产生影响[24].肖溪等[25]也对几种沉水植物的化感作用进行类似的研究,对于化感活性强的植物,大量胞外分泌物可能有针对性地抑制一种或者几种细菌的生长,使得部分细菌的种类和数量减少. 3种沉水植物中,金鱼藻有较强的化感活性,篦齿眼子菜和菹草具有较弱的化感活性[25],这可能是导致金鱼藻附着细菌多样性低于另外2种沉水植物的原因.
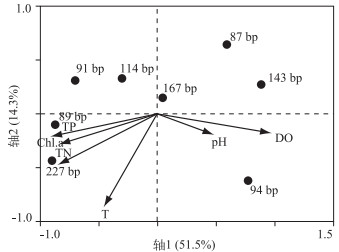
2.3 沉水植物附着细菌群落结构与环境因子相关性分析采样点总体水质情况较好(表 1),植物对水体营养盐的净化作用非常明显.菹草群丛和篦齿眼子菜群丛周围水体水化指标相差不大,均低于金鱼藻群丛.选取相对丰度超过5 %的优势T-RFs进行RDA分析. 89、227和91 bp T-RFs的丰度与TP、TN和叶绿素a浓度等呈正相关,与溶解氧浓度呈负相关;87、94和143 bp T-RF的丰度与溶解氧浓度呈正相关,而与TN、TP和叶绿素a浓度呈负相关;而167和114 bp T-RFs所代表细菌的丰度与以上4个环境因子之间的相关性比较小.此外87 bp T-RF与水温呈负相关(图 3).
|
图 3 样品T-RFs与环境因子的RDA分析 Fig.3 RDA showing different T-RFs in relation to the main environmental factors |
很多研究表明环境因子对附着细菌群落结构有一定的影响[26]. RDA分析结果表明部分附着细菌受到环境因子影响比较小,如167 bp T-RFs,它们稳定存在并成为优势菌群.另外一些菌群,如89、91、94、143和227 bp T-RFs,这类细菌根据环境变化而被检测出.而通过它们丰度变化可以了解一些湖泊生态系统健康状况,如γ-Proteobacteria数量增加代表水质变好,而Bacteroidetes的数量往往与藻类释放的有机物质降解过程密切联系[4].
3 结论1) 3种沉水植物附着细菌群落结构具有一定差异,主要表现在其片段种类及相对丰度不同.菹草样品和篦齿眼子菜样品的水化指标比较接近,但仍然存在部分T-RFs的相对丰度有较大差异,因此可以推测沉水植物自身在一定程度上对附着细菌的种类和丰度产生了影响,使其表现出一定的宿主选择性.
2) 部分限制性片段的丰度变化较大,如89、94、143和227 bp T-RFs,这些差异在一定程度上反映了不同样品的环境差异性.然而究竟是宿主植物还是水体营养盐等对它们的影响更大,仍然需要做深入研究.而如167 bp T-RFs在3种沉水植物中的相对丰度较为稳定,受上述环境因子的限制较小.这部分菌群普遍存在于不同植物叶表,它们能够适应生存环境并且成为优势种,很有可能对附着细菌的群落结构和功能有着重要的作用.
基于16S rRNA的分子生物学方法可弥补传统分离培养方法的不足,从而更全面地了解环境中微生物的群落结构.但由于T-RFLP技术定性分析具有不确定性,任意一种T-RF可能会代表一种或者多种细菌,因此需通过构建克隆文库或高通量测序来获得更多细菌物种的信息[23, 27],从而进一步解释附着微生物对于外界环境变动的响应机制.此外,附着细菌和宿主沉水植物之间的潜在关系,附着细菌是否对宿主沉水植物的衰亡过程起到作用也需要进一步调查研究[12].
| [1] |
He Dan, Ren Lijuan, Xing Peng et al. Research progress of diversity and community structure of epiphytic bacteria on submerged macrophytes. Chinese Bulletin of Life Sciences,, 2014, 26(2): 161-168. [何聃, 任丽娟, 邢鹏等. 沉水植物附着细菌群落结构及其多样性研究进展. 生命科学, 2014, 26(2): 161-168. DOI:10.13376/j.cbls/2014025] |
| [2] |
Bohn TJ, Lettenmaier DP, Sathulur K et al. Methane emissions from western Siberian wetlands: heterogeneity and sensitivity to climate change. Environmental Research Letters, 2007, 2(4). DOI:10.1088/1748-9326/2/4/045015 |
| [3] |
Zhao DY, Liu P, Fang C et al. Submerged macrophytes modify bacterial community composition in sediments in a large, shallow, freshwater lake. Canadian Journal of Microbiology, 2013, 59(4): 237-244. DOI:10.1139/cjm-2012-0554 |
| [4] |
Li Linlin, Tang Xiangming, Gao Guang et al. Influence of submerged vegetation restoration on bacterial diversity and community composition in West Lake. J Lake Sci, 2013, 25(2): 188-198. [李琳琳, 汤祥明, 高光等. 沉水植物生态修复对西湖细菌多样性及群落结构的影响. 湖泊科学, 2013, 25(2): 188-198. DOI:10.18307/2013.0203] |
| [5] |
Wu Qinglong, Xing Peng, Li Huabing et al. Impacts of regime shift between phytoplankton and macrophyte on the microbial community structure and its carbon cycling in lakes. Microbiology China, 2013, 40(1): 87-97. [吴庆龙, 邢鹏, 李化炳等. 草藻型稳态转换对湖泊微生物结构及其碳循环功能的影响. 微生物学通报, 2013, 40(01): 87-97.] |
| [6] |
Hempel M, Blume M, Blindow I et al. Epiphytic bacterial community composition on two common submerged macrophytes in brackish water and freshwater. BMC Microbiology, 2008, 8: 58. DOI:10.1186/1471-2180-8-58 |
| [7] |
Theil-Nielsen J, Sondergaard M. Production of epiphytic bacteria and bacterioplankton in three shallow lakes. Oikos, 1999, 86(2): 283-292. DOI:10.2307/3546446 |
| [8] |
Cai H, Jiang H, Krumholz LR et al. Bacterial community composition of size-fractioned aggregates within the phycosphere of cyanobacterial blooms in a eutrophic freshwater lake. Plos ONE, 2014, 9(8): 1-10. |
| [9] |
Gordon-Bradley N, Lymperopoulou DS, Williams HN. Differences in bacterial community structure on Hydrilla verticillata and Vallisneria americana in a freshwater spring. Microbes and Environments, 2014, 29(1): 67-73. DOI:10.1264/jsme2.ME13064 |
| [10] |
Cai X, Gao G, Tang X et al. The response of epiphytic microbes to habitat and growth status of Potamogeton malaianus Miq. in Lake Taihu. Journal of Basic Microbiology, 2013, 53(10): 828-837. |
| [11] |
Kahlert M, Pettersson K. The impact of substrate and lake trophy on the biomass and nutrient status of benthic algae. Hydrobiologia, 2002, 489(1/2/3): 161-169. |
| [12] |
He D, Ren LJ, Wu QL. Epiphytic bacterial communities on two common submerged macrophytes in Taihu Lake: diversity and host-specificity. Chinese Journal of Oceanology and Limnology, 2012, 30(2): 237-247. DOI:10.1007/s00343-012-1084-0 |
| [13] |
Gao Fangshu, Qian Yi, Wang Guoxiang et al. Characteristics and problems of the ecosystem in the Lake Hongze. Environmental Science & Technology, 2010, 33(5): 1-5. [高方述, 钱谊, 王国祥. 洪泽湖湿地生态系统特征及存在问题. 环境科学与技术, 2010, 33(5): 1-5.] |
| [14] |
Yu Hui, Zhang Wenbin, Lu Shaoyong et al. Spatial distribution characteristics of surface sediments nutrients in Lake Hongze and their pollution status evaluation. Environmental Science, 2010, 31(4): 961-968. [余辉, 张文斌, 卢少勇等. 洪泽湖表层底质营养盐的形态分布特征与评价. 环境科学, 2010, 31(4): 961-968.] |
| [15] |
Ren Y, Pei H, Hu W et al. Spatiotemporal distribution pattern of cyanobacteria community and its relationship with the environmental factors in Lake Hongze, China. Environmental Monitoring and Assessment, 2014, 186(10): 6919-6933. DOI:10.1007/s10661-014-3899-y |
| [16] |
Boyce Thompson Institute in Jiangsu Province. Jiangsu Flora. Nanjing: Jiangsu Science Press, 1975, 126-162. [江苏省植物研究所. 江苏植物志. 南京: 江苏科学出版社, 1975, 126-162.]
|
| [17] |
Jin Xiangcan, Tu Qingying eds. Investigation handbook of lake eutrophication . 2nd ed. Nanjing: China Environmental Science Press, 1990, 138-208. [金相灿, 屠清瑛. 湖泊富营养化调查规范. 第二版. 南京: 中国环境科学出版社, 1990, 138-208.]
|
| [18] |
Sipila TP, Keskinen AK, Akerman ML et al. High aromatic ring-cleavage diversity in birch rhizosphere: PAH treatment-specific changes of IE3 group extradiol dioxygenases and 16S rRNA bacterial communities in soil. Isme Journal, 2008, 2(9): 968-981. DOI:10.1038/ismej.2008.50 |
| [19] |
Swan BK, Ehrhardt CJ, Reifel KM et al. Archaeal and bacterial communities respond differently to environmental gradients in anoxic sediments of a California Hypersaline Lake, the Salton Sea. Applied and Environmental Microbiology, 2010, 76(3): 757-768. DOI:10.1128/AEM.02409-09 |
| [20] |
Wells GF, Park HD, Eggleston B et al. Fine-scale bacterial community dynamics and the taxa-time relationship within a full-scale activated sludge bioreactor. Water Research, 2011, 45(17): 5476-5488. DOI:10.1016/j.watres.2011.08.006 |
| [21] |
Kirchman DL. The ecology of Cytophaga-Flavobacteria in aquatic environments. FEMS Microbiology Ecology, 2002, 39(2): 91-100. |
| [22] |
Vander GK, Vande Kerckhove T, Vloemans N et al. Characterization of bacterial communities in four freshwater lakes differing in nutrient load and food web structure. FEMS Microbiology Ecology, 2005, 53(2): 205-220. DOI:10.1016/j.femsec.2004.12.006 |
| [23] |
Ren Lijuan, He Dan, Xing Peng et al. Bacterial diversity and ecological function in lake water bodies. Biodiversity Science, 2013, 21(4): 421-432. [任丽娟, 何聃, 邢鹏等. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性, 2013, 21(4): 421-432.] |
| [24] |
Bian Guiguo, Zhao Weidong, Da Lai. The research and application of the allelopathic effects of submerged plants inhibit the growth of algae. Northern Environment, 2012, 24(1): 59-64. [边归国, 赵卫东, 达来. 沉水植物化感作用抑制藻类生长的研究与应用. 北方环境, 2012, 24(1): 59-64.] |
| [25] |
Xiao Xi, Lou Liping, Li Hua et al. Algal control ability of allelopathically active submerged macrophytes: A review. Chinese Journal of Applied Ecology, 2009, 20(3): 705-712. [肖溪, 楼莉萍, 李华等. 沉水植物化感作用控藻能力评述. 应用生态学报, 2009, 20(3): 705-712.] |
| [26] |
Ji Haiting, Xie Dong, Zhou Hengjie et al. Advances in ecological research on epiphytic community of submerged macrophytes. J Lake Sci, 2013, 25(2): 163-170. [纪海婷, 谢冬, 周恒杰等. 沉水植物附植生物群落生态学研究进展. 湖泊科学, 2013, 25(2): 163-170. DOI:10.18307/2013.0201] |
| [27] |
Song Hongning, Du Binghai, Zhang Mingyan et al. Effect of environmental factors on bacterial community in Lake Dongping sediment. Acta Microbiologica Sinica, 2010, 50(8): 1065-1071. [宋洪宁, 杜秉海, 张明岩等. 环境因素对东平湖沉积物细菌群落结构的影响. 微生物学报, 2010, 50(8): 1065-1071.] |
 2016, Vol. 28
2016, Vol. 28 


