中华刺鳅(Mastacembelus sinensis)属于鲈形目(Perciformes)、刺鳅科(Mastacembelidae)、刺楸属(Mastacembelus),俗称石铲、刀鳅、钢鳅等,是广泛分布我国辽河、黄河、长江、钱塘江、珠江等水系的中小型淡水鱼类[1].改革开放以来,由于人为活动和江河环境污染,特别是农作物秸秆向水体中抛置,导致水体污染严重,中华刺鳅野生资受到严重破坏.目前,中华刺鳅资源量不断下降.
cyt b基因作为常用线粒体DNA分子标记,具有较强的中间解析和鉴别能力,广泛应用于动物的群体遗传结构、物种鉴别和系统进化的研究中[2-5].近年来,cyt b基因也越来越多地应用于鱼类遗传多样性研究.赵凯等[6]分析了黄河上游和柴达木盆地托索湖的裸裂尻鱼cyt b基因序列变异,初步认为黄河和柴达木水系托索湖的裸裂尻鱼未显著分化,两种群表现出较低水平的遗传多样性.阎雪岚等[7]采用cyt b序列分析了采自新疆克孜河3个群体的塔里木裂腹鱼及1个斑重唇鱼群体的种群遗传多样性和遗传结构,结果显示克孜河的塔里木裂腹鱼和斑重唇鱼均处于很低的遗传多样性水平,物种维持力较弱.赵峰等[8]采用cyt b基因片段为遗传标记,对黄海南部和东海的6个银鲳群体遗传结构进行了分析,结果表明6个群体均呈现出高单倍型多样性和低核苷酸多样性的特点,群体间具有高度的基因交流,是一个随机交配群体.田辉伍等[9]则利用cyt b基因研究了长江上游的长江干流、岷江、赤水河和嘉陵江的8个群体长薄鳅的遗传结构,表明长薄鳅种群内的变异大于种群间的变异,种群基因交流十分频繁.目前,有关刺鳅的研究很少,仅有的几篇报道主要集中于基础生物学[10-15]、病害研究[16]以及分类地位讨论[17-22].尤其是其分类地位一直存在争议,存在刺鳅(Mastacembelus aculeatus)和中华光盖刺鳅(Pararhynchobdlla sinensis)等多种学名,倪勇与伍汉霖先生[1]根据其形态特征比对,最终确定了其分类地位,学名为中华刺鳅(Mastacembelus sinensis).本研究对采自江苏省5个湖泊中华刺鳅种群cyt b片段进行了测序与比较分析,研究5个湖泊中华刺鳅群体间的遗传多样性和遗传结构,以深入了解和掌握5个湖泊中华刺鳅种群当前的种质资源现状,为其今后的保护与开发提供基础分子生物学依据.
1 材料和方法 1.1 研究材料实验用中华刺鳅于2015年5-7月采集自江苏省苏中和苏北地区的5个湖泊水域,分别为江苏淮安的白马湖(B)、高邮湖(G)、洪泽湖(H)、骆马湖(L)以及江苏泰州的溱潼湖(Q),剪取尾鳍,无水乙醇固定后,-20℃冷冻保存备用.
1.2 DNA提取基因组DNA采用试剂盒提取(Sangon Ezup Column Animal Genomic DNA Purification Kit).总DNA经检测后稀释至100 ng/μl,-20℃冷冻保存备用.
1.3 PCR扩增及测序参照GenBank数据库中大刺鳅(Mastacembelus armatus)的线粒体全序列(登入号:KJ184553),设计引物扩增cyt b基因,引物序列为F:5’-ATCCCTCCTGGGACTCTGTC-3’;R:5’-CGTGTGGAGGATTGGGACAA-3’,引物由上海生工生物公司合成. PCR反应体系为50 μl:模板DNA 50 ng;2×PCR Mix 25 μl(包含Taq酶2.5U,dNTPs 10 μmol,MgCl2 0.1 mmol),上、下游引物(10 μmol/L)各1 μl,其余体积用水补足.反应程序为:94℃预变性2 min,94℃变性45 s,62℃退火1 min,72℃延伸1 min,经35个循环后再72℃延伸10 min. PCR产物经割胶纯化后,直接送上海生工生物公司利用正反引物进行双向测序.
1.4 数据分析根据测序峰图对双向测序序列进行人工拼接,之后用BioEdit 7.0.5[23]软件比对获得同源序列,MEGA 6.0软件[24]分析碱基的组成,并构建系统进化树. DnaSP软件[25]统计变异位点类型与数目,计算单倍型种类、平均核苷酸差异数、单倍型多样性指数和核苷酸多态性等,用Arlequin3.01软件[26]中的Kimura 2-Parameters方法构建5个种群间的遗传距离,分子生物学方差分析(AMOVA)方法计算遗传分化指数(Fst)及遗传变异组成.
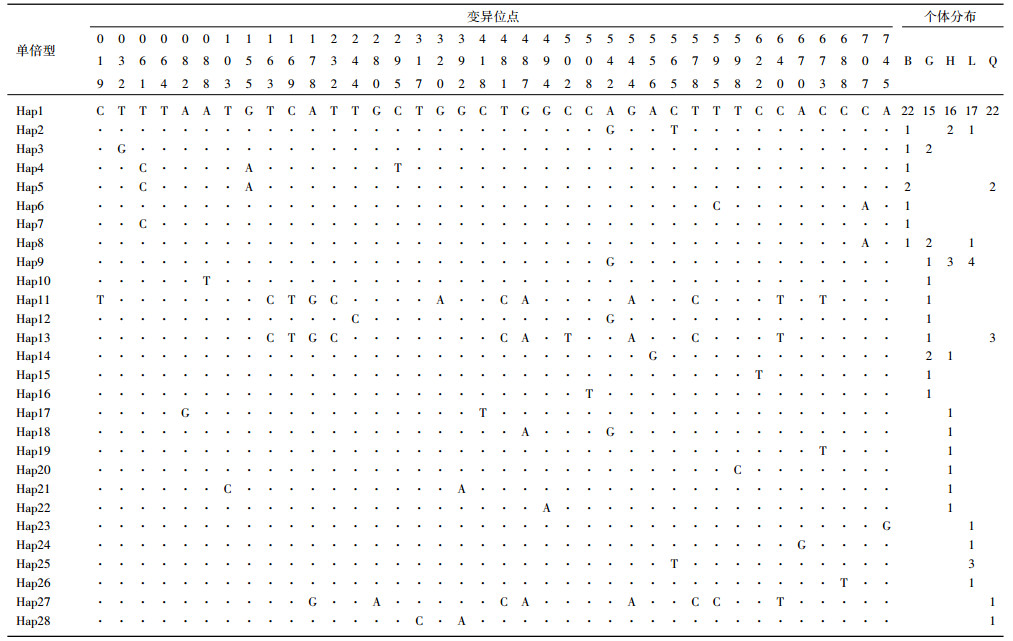
2 结果 2.1 序列分析5个湖泊中华刺鳅种群144个个体的cyt b基因,经拼接比对后,获得长度为767 bp的同源序列. MEGA 6软件分析表明,T、C、A、G碱基组成平均为29.2%、30.1%、26.1%和14.6%,A+T含量为55.3%,略高于G+C含量.序列共发现多态性位点38个,占核苷酸总数的4.95%,其中包含18个单态突变位点和20个简约信息位点,未检测到碱基缺失. 144个个体中共检出28个单倍型序列,所有单倍型序列已提交至GeneBank (登入号:KT448786~KT448813),种群间共享单倍型共8个,其中单倍型Hap1 5个群体间都有分布且数量也大,可能是原始单倍型(表 1). 5个种群的单倍型平均多样性指数为0.58751,平均核苷酸差异数为1.63947,平均核苷酸多样性指数为0.00214(表 2). 5个湖泊的中华刺鳅群体中,高邮湖群体的单倍型多样性指数最高,溱潼湖群体的核苷酸多样性指数最高,而骆马湖群体的单倍型多样性指数和核苷酸多样性指数均为最低.
| 表 1 中华刺鳅线粒体cyt b基因序列28种单倍型在5个种群中的分布 Tab.1 Distribution of 28 haplotypes in spiny eel populations from five lakes |
| 表 2 5个湖泊的中华刺鳅种群的单倍型及遗传多样性参数 Tab.2 Haplotypes and genetic diversity of spiny eel populations from five lakes |
将测序获得的144个个体的cyt b基因序列输入Arlequin软件,使用Kimura 2-Parameters方法构建5个湖泊种群间的相对遗传距离和Fst (表 3). 5个种群的平均遗传距离为0.00216,高邮湖种群与溱潼湖种群间的遗传距离最远为0.00338,遗传距离最近的是白马湖和骆马湖种群之间为0.00116.而5个湖泊刺鳅种群内部遗传距离从大到小依次为:溱潼湖(0.00368)>高邮湖(0.00311)>洪泽湖(0.00148)>白马湖(0.00118)>骆马湖(0.00107). 5个种群的平均Fst为0.03556,5个湖泊的中华刺鳅种群间存在一定的遗传分化,但是种群间的Fst值均较低,种群间Fst显著性检验有近一半(P > 0.05),表明5个湖泊中华刺鳅种群间存在较大的基因流,遗传分化差异较低(表 4).
| 表 3 5个湖泊的中华刺鳅种群间K 2-P遗传距离和遗传分化系数 Tab.3 Average K 2-P distance and fixation indice between spiny eel populations from five lakes |
| 表 4 5个湖泊中华刺鳅种群间遗传差异的AMOVA分析 Tab.4 Analysis of AMOVA of spiny eel populations from five lakes |
AMOVA法估算5个群体间遗传变异结构和来源的结果表明,种群间的变异占总变异的3.56%,而种群内的变异对总变异的贡献率达到96.44% (表 4),种群内未出现遗传分化.另外,用DnaSP软件分析单倍型数据所得遗传分化系数(Gst)为0.01092,基因流(Nm)为22.65;而144个个体的Gst为0.05601,Nm为4.21,也表明种群间的基因交流很频繁.
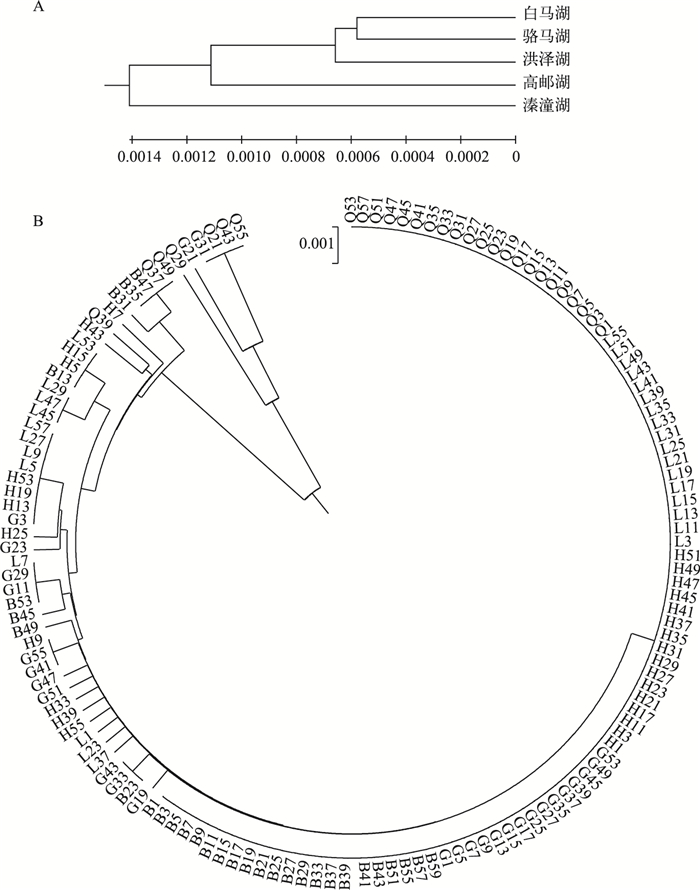
2.3 系统进化分析将144个中华刺鳅个体的cyt b基因序列输入MEGA6软件,对5个湖泊群体进行系统进化分析,得到UPGMA系统进化树(图 1A). 5个中华刺鳅群体没有明显聚类,形成了单系类群,京杭大运河水系连接的白马湖、骆马湖、洪泽湖和高邮湖群体首先聚类,最后与溱潼湖聚合.而144个个体的构建的UPGMA系统进化树则表明,5个群体的个体都没有独立聚类,相互散布在不同的地理群体中(图 1B).
|
图 1 5个湖泊中华刺鳅群体间(A)及个体间(B)的UPGMA系统进化树 Fig.1 Dendrogram of spiny eel populations (A) and 144 individuals (B) from five lakes by UPGMA method based genetic distance |
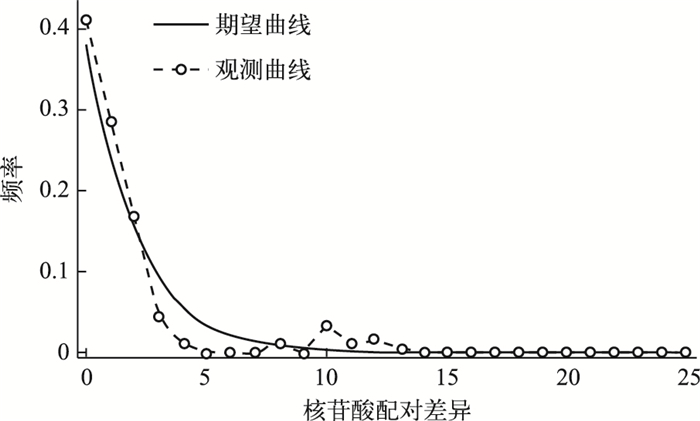
用Fu’Fs检验方法检验5个湖泊中华刺鳅群体的中性突变,结果表明种群的整体Fs=-23.887(P < 0.05),显著偏离中性突变,表明5个中华刺鳅群体整体上有一定的扩张趋势,但白马湖、骆马湖、高邮湖和洪泽湖群体的Fs值都小于0,而溱潼湖群体的Fs值大于0.所有个体的线粒体cyt b基因序列进行碱基歧点分布分析,个体间碱基歧点分布的观测曲线与期望曲线大体一致,中间只有一个较低的凸起(图 2),表明5个湖泊的中华刺鳅历史上种群相对比较稳定,经历过一个比较小的种群扩张事件.
|
图 2 5个湖泊的中华刺鳅群体线粒体cyt b基因错配碱基分布 Fig.2 The mismatch distribution of the Mitochondrial DNA cyt b of spiny eel populations from five lakes |
本研究获得了5个湖泊中华刺鳅群体的线粒体cyt b基因片段,序列碱基组分析表明,中华刺鳅线粒体cyt b基因的A+T含量为55.3%,略高于G+C平均含量,而G含量显著低于其他碱基,表现出反G偏倚,与其他动物线粒体DNA的研究结果一致[27].
遗传多样性是一个物种长期生存和进化的前提,物种的遗传多样性越高,该物种的进化潜力也就越大,对环境变化的适应能力也就越强[5].本研究获得5个湖泊144尾中华刺鳅cyt b基因767 bp的序列,共获得基因单倍型28个,变异位点38个,单倍型多样性和核苷酸多样性分别为0.58751和0.00214,说明江苏省5个湖泊的中华刺鳅单倍型多样性和核苷酸多样性都很低,远低于黄颡鱼[4] (0.945、0.00419)和福建近海竹荚鱼[28] (0.937、0.336)等经济鱼类的cyt b基因单倍型多样性和核苷酸多样性,甚至低于野生濒危物种鱇浪白鱼[5] (0.996、0.00434) cyt b基因的单倍型多样性和核苷酸多样性,仅与濒危物种青海湖裸鲤[29](0.783、0.00205)、黄河裸体裂尻[6](0.700、0.0026)和长薄鳅[9] (0.60852、0.00089) cyt b基因的单倍型多样性和核苷酸多样性相当,高于怒江角鱼[30](0.579、0.00070) cyt b基因单倍型多样性和核苷酸多样性.如此低的遗传多样性,可能意味着中华刺鳅种群曾经历过“瓶颈效应”的打击[31],说明当前该物种的进化潜力比较低,对环境变化的适应能力很弱,存在着较大的灭绝风险.
3.2 种群遗传结构AMOVA分析表明,种群内的遗传变异显著超过各种群间的遗传变异,占全部遗传变异的96.44%,这表明中华刺鳅群体间差异不显著,群体间未出现遗传分化.
Fst值可以反映群体之间的遗传分化程度,当Fst值介于0~0.05时表示分化较弱.本文中5个湖泊中华刺鳅群体的Fst值为0.0356,说明5个中华刺鳅种群间存遗传分化较弱,不存在显著的系统地理分化. Shaklee等[32]根据已发表的文献认为鱼类在种群、种和属三级水平上的遗传距离D值分别为0.05、0.30和0.9.中华刺鳅5个湖泊群体间遗传距离为0.00216,说明中华刺鳅群体间的遗传分化极低,远没有达到种群的分化水平.另外,与其他鱼类群体间的遗传距离相比较(0.035~0.019)[33],中华刺鳅群体间遗传距离(0.00116~0.00338)可以看出,中华刺鳅种群无论是群体间还是群体内部序列差异都极小,说明其遗传多样性水平极低. Wright提出种群Nm > 4时,群体就是一个随机的单位[34].而本研究中单倍型间的Nm高达22.65,个体间的Nm为4.21,表明5个群体间基因流动的相当强烈,也进一步说明中华刺鳅种群间未出现分化,应作为一个整体进行资源保护.系统进化分析表明5个湖泊的中华刺鳅群体没有明显聚类,形成了单系类群.以上结果都表明中华刺鳅5个湖泊群体在cyt b水平上未出现群体遗传分化. Grant等依据单倍型多样度h和核苷酸多样度π推测了鱼类群体的4种进化情景[35],本研究中5个湖泊的中华刺鳅种群遗传多样性低,遗传没有分化,接近于低h、低π情形,可能因为湖泊间地理距离近,水系连通,使得基因交流的可能性很大;同时,也可能存在建立者效应,即5个湖泊的中华刺鳅群体都源于单一建立者;此外,江河等水体环境污染和人为活动导致栖息地减少,致使中华刺鳅群体数量急剧下降,而群体变小后,瓶颈效应和遗传漂变可能在某种程度上导致其遗传多样性的降低,损失的遗传变异也会增多.
遗传多样性是生命进化的基础,遗传多样性的缺失会威胁到种群的生存.本研究以cyt b基因为工具,研究了5个湖泊中华刺鳅群体间的遗传多样性和遗传结构,以深入了解和掌握当前中华刺鳅种质资源现状,为其今后的保护与开发提供基础分子生物学依据. 5个湖泊的中华刺鳅种群处于较低的遗传多样性水平,且未形成遗传分化,物种维持力较弱.本研究结果表明当前5个湖泊的中华刺鳅资源现状不容乐观,亟待加强保护.种群一旦遭受破坏,就很难以复,这个宝贵物种也将面临灭绝的危险.同时,应尽快对其他水域中华刺鳅遗传多样性现状进行研究,全面了解该物种种质资源现状.
| [1] |
Ni Yong, Wu Hanlin. Fishes of Jiangsu Province. Beijing: China Agriculture Press, 2006, 740-742. [倪勇, 伍汉霖. 江苏鱼类志. 北京: 中国农业出版社, 2006, 740-742.]
|
| [2] |
Brown WM.Evolution of animal mitochondrial DNA.In:Nei M, Koehn RK eds.Evolution of genes and proteins.Sunderland MA:Sinauer, 1983:62-88.
|
| [3] |
Saccone C, Pesole G, Sbisa E. The main regulatory region of mammalian mitochondrial DNA:structure-function model and evolutionary pattern. J Mol Evol, 1991, 33(1): 83-91. DOI:10.1007/BF02100199 |
| [4] |
Zhong Liqiang, Liu Pengpeng, Pan Jianlin et al. Genetic variation analysis of yellow catfish (Pelteobagrus fulvidraco) from five lakes in the middle and lower reaches of the Yangtze River based on Mitochondrial DNA cyt b. J Lake Sci, 2013, 25(2): 302-308. [钟立强, 刘朋朋, 潘建林等. 长江中下游5个湖泊黄颡鱼种群线粒体细胞色素b基因的遗传变异分析. 湖泊科学, 2013, 25(2): 302-308. DOI:10.18307/2013.0219] |
| [5] |
Liu Hongyan, Xiong Fei, Yang Dong et al. Mitochondrial cytochrome b gene sequence diversity in wild and cultured populations of Anabarilius grahami. J Huazhong Agric Univ, 2011, 30(1): 94-98. [刘红艳, 熊飞, 杨东等. 基于细胞色素b基因的鱇浪白鱼野生群体和养殖群体遗传多样性分析. 华中农业大学学报, 2011, 30(1): 94-98.] |
| [6] |
Zhao Kai, Yang Gongshe, Li Junbing et al. Phylogenetic structure of Schizopygopsis pylzovi populations from Mitochondrial cytochrome b gene sequence variations. Acta Hydrob Sinica, 2006, 30(2): 129-133. [赵凯, 杨公社, 李俊兵等. 黄河裸裂尻鱼群体遗传结构和cyt b序列变异. 水生生物学报, 2006, 30(2): 129-133.] |
| [7] |
Yan Xuelan, Yang Jinquan, Tang Wenqiao et al. Genetic diversity of two Cyprinid fishes:Schizothorax biddulphi and Diptychus maculates in Kezilesu River, Xinjiang Uygur Autonomous Region based on Cyt b gene sequences variation. Chinese J Zool, 2009, 44(5): 8-13. [阎雪岚, 杨金权, 唐文乔等. 基于线粒体Cyt b基因序列变异的克孜河塔里木裂腹鱼和斑重唇鱼遗传多样性. 动物学杂志, 2009, 44(5): 8-13.] |
| [8] |
Zhao Feng, Zhuang Ping, Zhang Longzhen et al. Population genetic structure of Pampus argenteus in the south Yellow Sea and East China Sea based on the mitochondrial Cyt b sequence. Acta Hydrob Sinica, 2011, 35(5): 745-752. [赵峰, 庄平, 章龙珍等. 基于线粒体Cytb基因的黄海南部和东海银鲳群体遗传结构分析. 水生生物学报, 2011, 35(5): 745-752.] |
| [9] |
Tian Huiwu, Duan Xinbin, Wang Dengqiang et al. Sequence variability of Cytochrome b and genetic structure of Leptobotia elongata in the upper Yangtze River. Freshw Fish, 2013, 43(6): 13-18. [田辉伍, 段辛斌, 汪登强等. 长江上游长薄鳅Cytb基因的序列变异与遗传结构分析. 淡水渔业, 2013, 43(6): 13-18.] |
| [10] |
Wu Lv. Cellular composition of the islets in Mastacembelus. Science, 1950, 32(3): 91. [伍律. 刺鳅胰岛之细胞组成. 科学, 1950, 32(3): 91.] |
| [11] |
Wu Lv. The pancreas and the pancreatic islets of a teleost, Mastacembelus aculeatus. Acta Hydrob Sinica, 1956, 2(2): 272-278. [伍律. 刺鳅的胰脏和胰岛. 水生生物学集刊, 1956, 2(2): 272-278.] |
| [12] |
Zhou Rongjia, Yu Qixing, Cheng Hanhua et al. PCR amplification of SRY box gene in Monopterus albus and Mastacembelus aculeatus. Chinese Sci Bull, 1996, 41(7): 640-642. [周荣家, 余其兴, 程汉华等. PCR扩增黄鳝和刺鳅SRY盒基因. 科学通报, 1996, 41(7): 640-642.] |
| [13] |
Liu Jiangdong, Huang Xiao, Yu Qixing et al. The cytogenetic evidences for confirming the existence of sex chromosomes in Mastacembelus aculeatus(Osteichthyes, Perciformes). J Wuhan Univ (Nat Sci Ed), 1999, 45(2): 185-190. [刘江东, 黄晓, 余其兴等. 刺鳅性染色体的细胞遗传学确定证据. 武汉大学学报:自然科学版, 1999, 45(2): 185-190.] |
| [14] |
Chen Ji, Zhao Gang, Zang Yating et al. The construction of X chromosome library of spiny eel (Mastacembelus aculeatus). Acta Hydrob Sinica, 2009, 33(4): 571-576. [陈戟, 赵刚, 臧亚婷等. 刺鳅X染色体DNA文库的构建. 水生生物学报, 2009, 33(4): 571-576.] |
| [15] |
Wu Yuanan, Liang Zhiqiang, Li Chuanwu et al. Analysis and evaluation of nutritional components in the muscle of two kinds of Mastacembelus. Acta Nutrimenta Sinica, 2010, 32(5): 499-502. [伍远安, 梁志强, 李传武等. 两种刺鳅肌肉营养成分分析及评价. 营养学报, 2010, 32(5): 499-502.] |
| [16] |
Han Jiugao. Infection and control of Ichthyophthiriasis spp.on Mastacembelus aculeatus. J Anhui Agri Sci, 2007, 35(16): 4826-4834. [韩九皋. 刺鳅感染小瓜虫病及防治研究. 安徽农业科学, 2007, 35(16): 4826-4834. DOI:10.3969/j.issn.0517-6611.2007.16.060] |
| [17] |
Travers RA. A review of the Mastacembeloidei, a suborder of synbranchiform teleost fishes.Part Ⅱ:hylogenetic analysis. Bull Br Mus Nat Hist Zool, 1984, 47(2): 83-150. |
| [18] |
Kottelat M. Notes on the taxonomy and distribution of some western Indonesian freshwater fishes, with diagnoses of a new genus and six new species (Pisces:Cyprinidae, Belonidae, and Chaudhuriidae). Ichthyol Explor Fres, 1991, 2(3): 273-287. |
| [19] |
Johnson GD, Patterson C. Percomorph phylogeny:A survey of Acanthomorphs and a new proposal. Bull Mar Sci, 1993, 52(1): 554-626. |
| [20] |
Kottelat M, Lim KP. Diagnosis of two new genera and three new species of earthworm eels from the Malay Peninsula Borneo (Teleostei:Chaudhuriidae). Ichthyol Explor Fres, 1994, 5(2): 181-190. |
| [21] |
Britz R. Ontogeny of ethmoidal region and hyopalatine arch in Macrognathus pancalus(Percomorpha, Mastacembeloidei), with critical remarks on mastacembeloid inter-and intrarelationships. Am Mus Novit, 1996, 3181: 1-18. |
| [22] |
Vreven EJ. Mastacembelidae (Teleostei; Synbranchiformes) subfamily division and African generic division:An evaluation. J Nat Hist, 2005, 39(4): 351-370. DOI:10.1080/0022293042000195975 |
| [23] |
Hall TA. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl Acids Symp Ser, 1999, 41: 95-98. |
| [24] |
Tamura K, Stecher G, Peterson D et al. MEGA6:Molecular evolutionary genetics analysis version 6.0. Mol Biol Evol, 2013, 30(4): 2725-2729. |
| [25] |
Rozas J, Anchez-DelBarrio JC, Esseguer XM et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods. Bioinformatics, 2003, 19(18): 2496-2497. DOI:10.1093/bioinformatics/btg359 |
| [26] |
Excoffier L, Laval G, Schneider S.Arlequin ver 3.01:An integrated software package for population genetics data analysis.Berne:Computational and Molecular Population Genetics Laboratory (CMPG), Switzerland:University of Berne, 2006.
|
| [27] |
Wolstenholme DR. Animal mitochondrial DNA:Structure and evolution in:Mitochondrial genomes. San Diego: Academic Press, 1992, 173-372.
|
| [28] |
Niu Sufang, Su Yongquan, Wang Jun et al. Genetic polymorphism of mitochondrial control region and cyt b in Trachurus japonicus from Fujian coastal waters. J Fish Sci China, 2011, 18(1): 66-74. [牛素芳, 苏永全, 王军等. 福建近海竹荚鱼线粒体DNA控制区和细胞色素b遗传多态性. 中国水产科学, 2011, 18(1): 66-74.] |
| [29] |
Zhao Kai, He Shunping, Peng Zuogang et al. Mitochondrial diversity and population structure of Gymnocypris przewalskii przewalskii (Teleostei:Cyprinidae). J Qinghai Univ (Nat Sci), 2006, 24(4): 1-4. [赵凯, 何舜平, 彭作刚等. 青海湖裸鲤的种群结构和线粒体DNA变异. 青海大学学报, 2006, 24(4): 1-4.] |
| [30] |
Zhang Dongya, Chen Yong, Liu Shaoping et al. Population genetic structure of the endangered species Epalzeorhynchus bicornis in the Nujiang River. Freshw Fish, 2009, 39(2): 3-7. [张东亚, 陈勇, 刘绍平等. 怒江濒危鱼类角鱼种群遗传结构研究. 淡水渔业, 2009, 39(2): 3-7.] |
| [31] |
Billington N, Hebert PD. Mitochondrial DNA diversity in fishes and its implications for introductions. Canadian Journal of Fisheries and Aquatic Sciences, 1991, 48. |
| [32] |
Shaklee JB, Tamaru CS, Waples RS. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins. Pac Sci, 1982, 36(2): 141-157. |
| [33] |
Chen Di.Analysis of genetic structure in mitochondrial cytochrome b of Coilia ectenes in Yangtze River[Dissertation].Guangzhou:Jinan University, 2006(in Chinese with English abstract). [陈迪. 长江刀鲚遗传多样性的细胞色素b基因序列分析[学位论文]. 广州: 暨南大学, 2006. http://cdmd.cnki.com.cn/article/cdmd-10559-2007044716.htm ]
|
| [34] |
Lü Baozhong, Zhong Yang, Gao Liping et al. Molecular evolution and phylogenetics. Beijing: High Education Press, 2002. [吕宝忠, 钟扬, 高莉萍等. 分子进化与系统发育. 北京: 高等教育出版社, 2002.]
|
| [35] |
Grant WS, Bowen BW. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation. Journal of Heredity, 1998, 89: 415-426. DOI:10.1093/jhered/89.5.415 |
 2016, Vol. 28
2016, Vol. 28