(2: 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008)
(3: 环境保护部南京环境科学研究所南京国环科技股份有限公司, 南京 210042)
(2: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
(3: Nanjing Guohuan Science and Technology Co., Ltd of Nanjing Institute of Environmental Sciences, MEP, Nanjing 210042, P. R. China)
磷是生命体必需的营养元素,也是水生态系统中最重要的生源要素之一.在水生态系统中,一般能为生物直接利用的只有溶解性反应磷(soluble reactive phosphorus, SRP),因此磷也是水生态系统维持初级生产力的重要因子[1-2].水生态系统中溶解性反应磷的浓度通常较低,在一些寡营养的海洋水体中,溶解性反应磷的浓度甚至仅处于纳摩尔级别[3-4].为了维持自身的生长与繁殖,水生微生物也进化出了不同的生存策略.对于“严格寡营养性微生物(Strict oligotrophs)”来说,其可通过降低自身对于磷营养的需求来适应低磷环境,如寡营养海洋中的原绿球藻(Prochlorococcus)和远洋杆菌(Pelagibacter)[5-6].而“机会营养性微生物(Opportuni-trophs)”则有多种策略应对低磷环境[7],最典型的就是“磷饥饿”响应策略.水生生物在“磷饥饿”条件下,可通过分泌碱性磷酸酶,酶解水体或自身细胞中的磷酸脂类有机磷,进而获得可直接利用的溶解性反应磷[8-9].碱性磷酸酶能够促进水体有机磷的矿化,对于维持水生态系统中的初级生产力等具有重要的生态学作用[10].
细菌是水生态系统中微食物网的重要组成部分[11],也是有机磷的重要矿化者[12].现有的研究已获得了海洋生态系统中细菌碱性磷酸酶的种类、亚细胞分布、活性和功能基因的表达及相关因素等方面的知识[13-22],但对淡水生态系统,尤其是湖泊中细菌碱性磷酸酶的研究还多处在关注其活性及其与环境因素关系的层面[23-34],缺乏对细菌碱性磷酸酶更为深入的认识.
我国是一个多湖泊国家,全国1 km2面积以上的湖泊有2759个,其中有651个位于长江中下游地区,且大多数为浅水富营养化湖泊[35].由于气候和人为因素的影响,浅水富营养化湖泊常爆发严重的蓝藻水华,加上浅水湖泊本身具有的小幅高频的水动力扰动和差异显著的生态系统结构等生态特征,使得浅水富营养化湖泊的物质循环和能量流通过程显著区别于海洋和深水湖泊.而且,浅水富营养化湖泊生物的生长与繁殖也常常受到磷营养的限制.因此,研究细菌碱性磷酸酶编码基因对于揭示水体中有机磷矿化的微生物驱动机制具有重要意义.
本文通过综述国内外有关细菌碱性磷酸酶及其编码基因的研究进展,阐述了细菌碱性磷酸酶在湖泊有机磷矿化过程中的地位,总结了细菌碱性磷酸酶编码基因的特性、功能以及研究方法,分析了湖泊细菌碱性磷酸酶的微生物学机制与研究前景,为我国湖泊富营养化机理的研究工作提供参考.
1 水体碱性磷酸酶的来源天然水生态系统中的碱性磷酸酶来源复杂,水体中的细菌、浮游植物、浮游动物、底栖生物和大型水生植物都可以分泌碱性磷酸酶.目前学者认同的观点是水生态系统中碱性磷酸酶主要来源于浮游藻类和细菌[36],而且在不同的天然水生态系统中,这2种来源的碱性磷酸酶对水体总碱性磷酸酶活性的贡献也有差异,通常浮游藻类产生的碱性磷酸酶活性要高于细菌产生的碱性磷酸酶活性,这与部分细菌附着于浮游藻类颗粒上增加了浮游藻类碱性磷酸酶活性有一定的关系[37].同时,在物种亲缘关系上,浮游植物中的大部分藻类还是属于原核生物,与异养细菌在基因水平上有很好的遗传发育关系,例如微囊藻(Microcystis)和超微藻(picoplankton).更为广泛的定义认为细菌应包括异养细菌和原核藻类[38].
水体中的碱性磷酸酶主要分布于细菌或藻类细胞的胞内和胞外[39].胞内碱性磷酸酶主要分布于细胞内膜、周质和细胞质.胞外碱性磷酸酶包括附着于细胞外膜的碱性磷酸酶和水体溶解态碱性磷酸酶.很多种细菌和浮游藻类分泌的胞外碱性磷酸酶既能附着在细胞表面,又能以溶解态存在于水体中[40].细菌是水生态系统中胞外碱性磷酸酶的主要产生者[41],其胞外碱性磷酸酶活性通常对湖泊水体的碱性磷酸酶活性有相当比例的贡献[42],在水生态系统的有机磷矿化过程中起着重要作用.
2 细菌碱性磷酸酶及其编码基因 2.1 细菌碱性磷酸酶的种类细菌碱性磷酸酶的种类多样,根据碱性磷酸酶蛋白序列的相似度和底物的不同,可主要分为3种类型:PhoA、PhoX和PhoD[13].
PhoA是研究最早的一种磷酸单脂酶,最初发现可由大肠杆菌(Escherichia coli)表达合成. PhoA是一种二聚体蛋白,其活性部位有用于磷酸催化的2个Zn2+和1个Mg2+组成的辅基团.这种碱性磷酸酶蛋白可通过细菌胞膜上的‘Sec’蛋白通道分泌到胞外或细胞周质中[43].
碱性磷酸酶PhoX和PhoD是近年来的研究热点,其编码基因最早分别发现于霍乱弧菌(Vibrio cholerae)[44]和枯草杆菌(Bucillus subtilis)的基因组[45].与碱性磷酸酶PhoA不同的是,这2种碱性磷酸酶均为单体蛋白,且碱性磷酸酶PhoX的活性部位有2个Fe3+与3个Ca2+作为辅基[46],2种磷酸酶的分泌通道均是双精氨酸(Twin arginine)蛋白通道[47-49].同时,PhoX和PhoD的底物不仅有磷酸单脂类,还包括磷酸二脂类化合物.因此,这2种酶既是磷酸单脂酶,也是磷酸二脂酶[13].
研究表明,3种细菌碱性磷酸酶功能区域的蛋白序列相似性较低(图 1),同种碱性磷酸酶的蛋白序列之间也有一定分异[50].目前,普遍认为在海洋和土壤生态系统中,碱性磷酸酶PhoD基因序列的多样性和丰度要高于其他2种酶的基因序列[13, 20, 50].
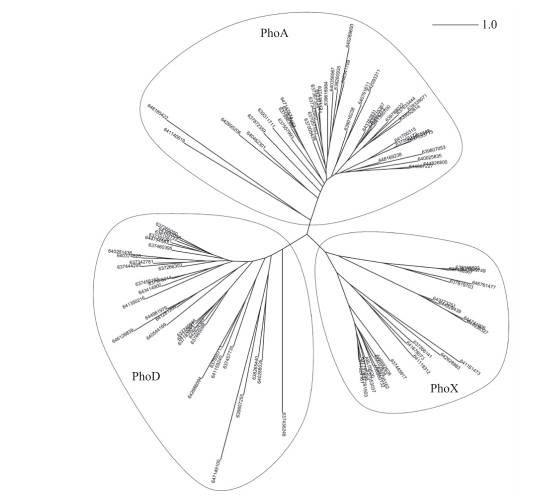
|
图 1 碱性磷酸酶PhoA、PhoX和PhoD的最大似然系统发育树[50] Fig.1 The maximum likelihood phylogenetic tree of alkaline phosphatases PhoA, PhoX and PhoD[50] |
除以上3种主要的细菌碱性磷酸酶,细菌还可能合成多种其他碱性磷酸酶,如PhoZ[51]、PhoV[52]等,不过目前仍缺乏针对这些潜在基因的表达与蛋白功能鉴定的研究.
2.2 细菌碱性磷酸酶的亚细胞分布与功能途径细菌碱性磷酸酶的亚细胞分布特征对于认识细菌碱性磷酸酶的功能有重要意义,通过生物信息学手段可有效推算碱性磷酸酶蛋白的亚细胞分布.最初,针对革兰氏阴性细菌的研究认为,碱性磷酸酶只分布于细胞周质(Periplasmic)[53-55].随后的研究发现,细菌碱性磷酸酶还分布于细胞表面与胞外环境中[56-57]. Luo等[13]通过对全球海洋调查(Global Ocean Sampling, GOS)基因组数据库中细菌碱性磷酸酶编码基因序列的生物信息学推算发现(图 2),在3733个细菌碱性磷酸酶基因序列中,有1518个(40.7 %)序列来自于细胞质内,641个(17.7 %)来自于细胞周质,1100个(29.5 %)来自于胞外环境,439个(11.8 %)来自于细胞外膜,还有35个(11.8 %)来自于细胞内膜.有超过70 %的细菌碱性磷酸酶分布于细胞内部.对于3种主要的碱性磷酸酶,超过40 %的PhoD和PhoA均分布于细胞质内,超过50 %的PhoX序列则分布于细胞外部环境.细菌碱性磷酸酶的这些亚细胞分布特征可能暗示PhoA、PhoX和PhoD有着不同的有机磷矿化途径.
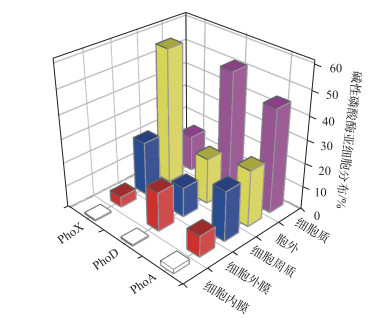
|
图 2 基于GOS细菌基因组数据库推算的细菌碱性磷酸酶的亚细胞分布(改自文献[13]) Fig.2 Subcellular localization distributions of alkaline phosphatases recovered from the GOS metagenomic database(modified from reference[13]) |
根据Luo等[13]的研究,White[15]将细菌碱性磷酸酶利用溶解态有机磷的途径归纳为3种(图 3):(1)小分子磷脂类有机磷经细胞周质内的磷脂运输系统(uptake of glycerol phosphate system,ugp)传输至细胞质内,由分布于细胞质内的碱性磷酸酶(主要是PhoA和PhoD)分解;(2)进入细胞周质的小分子有机磷脂类化合物由间质内的碱性磷酸酶分解;(3)细菌产生胞外碱性磷酸酶(主要是PhoX)利用胞外环境中的有机磷,产生的部分无机态正磷酸盐再由细菌吸收利用.其中,第一种途径对于细菌吸收磷营养可能最为重要.
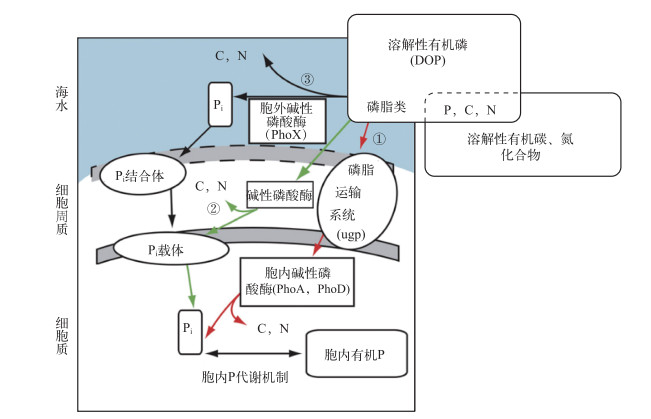
|
图 3 海洋细菌利用溶解性有机磷(DOP)的3种主要途径(改自文献[15]) Fig.3 Three predominant pathways of using dissolved organophosphate(DOP) by marine bacteria(modified from reference[15]) |
生态学中,常将调控生物地球化学循环的某些关键酶的表达基因作为标记物,采用分子生物学的手段,研究具有这些功能基因的微生物的多样性、群落结构、丰度以及相应的功能表达.例如,氨单加氧酶编码基因(amoA)作为分子标记物,被广泛用于研究环境中的氨氧化细菌和古菌群落[58-59].同样,将碱性磷酸酶编码基因作为标记物也为更好地认识碱性磷酸酶功能细菌及其功能特性提供了便利[13-14].
Sakuari等[60]首次采用变性梯度凝胶电泳法(denaturing gradient gel electrophoresis, DGGE),研究有机和化学肥料影响下土壤中碱性磷酸酶编码基因的多样性,并发现与洛蒂根瘤菌(Mesorbizobium loti)和荧光假单胞菌(Pseudomonas fluorescens)的碱性磷酸酶编码基因相似度较高的基因序列. Sebastian等[14]通过聚合酶链式反应(ploymerase chain reaction, PCR)与基因克隆方法,结合分析全球海洋调查基因组数据库发现,寡营养海洋水体中碱性磷酸酶PhoX编码基因phoX在细菌基因组中分布的广泛性要明显高于碱性磷酸酶PhoA,碱性磷酸酶编码基因phoX普遍分布于不同门类的细菌中,主要包括变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)和蓝细菌门(Cyanobacteria)等(图 4).但Luo等[13]运用生物信息学手段对全球海洋调查基因组数据库中phoA、phoX、phoD共3733条碱性磷酸酶基因的研究却发现,3种碱性磷酸酶编码基因均分布于不同门类的细菌中,其中以变形菌门占多数,phoA基因在拟杆菌门和绿菌门(Chloroflexi)中的比例要高于其他2类基因,而phoX基因在蓝细菌门中的比例也要高于phoA和phoD基因.
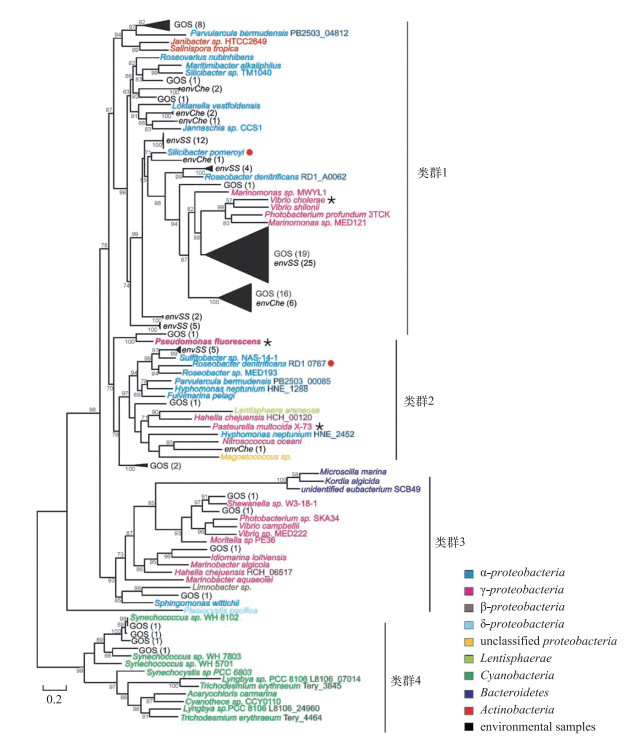
|
图 4 海洋细菌phoX基因对应磷酸酶蛋白的最大似然进化树(改自文献[14]) Fig.4 The maximum likelihood phylogenetic tree for putative PhoX protein of marine bacterial phoX(modified from reference [14]) |
目前,采用phoX基因研究碱性磷酸酶PhoX及其功能细菌的研究较多. Zaheer等[61]通过对GenBank中的phoX基因对应的蛋白序列的系统发育分析发现,phoX基因的蛋白序列可分为2种类型(PhoX-Ⅰ和PhoX-Ⅱ),其中PhoX-Ⅰ类型主要包括部分α变形菌门(α-proteobacteria)和γ变形菌门(γ-proteobacteria). Zaheer等[61]还证实同种细菌的phoX基因在基因组中存在水平迁移现象,例如,洛蒂根瘤菌MAFF303099 (Mesorbizobium loti strain MAFF303099)基因组中就有2种不同的phoX基因.还有不少学者也发现了一些蓝细菌携有新的phoX基因[18-19, 22, 62-63],并借此研究phoX基因的表达与影响因素.
对海洋和土壤生态系统的细菌基因组数据库的研究表明,碱性磷酸酶编码基因phoD的丰度要高于phoA和phoX基因[13, 20, 50].近年来,细菌phoD基因也开始成为学者研究的热点. Kageyama等[64]从嗜盐隐杆藻(Aphanothece halophytica)的基因组中成功克隆了一种phoD基因,并发现这种phoD基因的表达受盐度胁迫的影响要高于磷营养的限制. Luo等[20]对北太平洋亚热带环流的基因组数据库分析发现,透光层和深海水体中phoD基因的丰度要明显高于phoA基因.也有不少学者通过设计phoD基因探针,从土壤中获取phoD基因的信息,并以此研究施肥等因素对细菌碱性磷酸酶编码基因及其细菌的影响[50, 60, 65].随着学者对蓝藻水华问题的关注,一些研究也发现了水华优势蓝细菌种中phoD基因的存在,最典型的就是鱼腥藻(Anabaena sp.)[66].
研究表明,部分产碱性磷酸酶细菌的基因组中可能有两种甚至多种碱性磷酸酶编码基因.例如,海洋细菌物种unidentified Eubacterium SCB49和副溶血弧菌(Vibrio parahaemolyticus)的基因组中就同时含有phoA和phoX两种基因[14],而Mesorbizobium loti strain MAFF303099的基因组中有2种不同的phoX基因[61].相反,基因组中仅有1个碱性磷酸酶编码基因的细菌物种常用作研究基因表达与影响因素的模式生物,如玫瑰杆菌(Silicibacter pomeroyi)和鲁杰氏菌(Ruegeria pomeroyi)2种细菌,其基因组中只有1种phoX功能基因[14, 21].
2.4 碱性磷酸酶编码基因表达的环境因素 2.4.1 正磷酸盐浓度作为细菌可直接吸收利用的磷源和碱性磷酸酶的酶促产物,正磷酸盐浓度的高低会影响碱性磷酸酶编码基因的表达. Wanner[43]总结正磷酸盐浓度对大肠杆菌phoA基因表达影响的机理.当大肠杆菌处于磷限制状态时,其会进行甘油磷酸盐吸收基因和碱性磷酸酶PhoA基因的表达,同时也会开启一种高亲和性磷酸盐传输蛋白的编码基因pst的表达,直接吸收环境中低浓度的正磷酸盐,或者将环境中的有机磷吸收至细胞内由PhoA酶解,部分有机磷甚至可直接用于合成细胞膜磷脂分子.在磷酸盐过剩时,大肠杆菌只需合成一种低亲和性磷酸盐传输蛋白Pit,直接吸收环境中的磷酸盐.
Sebastian等[14, 21]通过对3种细菌, 玫瑰杆菌、反硝化玫瑰杆菌(Roseobacter denitrificans)和Ruegeria pomeroyi基因组中phoX基因表达的研究发现,这3种细菌的碱性磷酸酶PhoX基因的表达对磷限制有明显的响应,过剩的正磷酸盐会显著抑制phoX基因的表达.同样苜蓿中华根瘤菌(Sinorhizobium meliloti)的phoX基因表达对正磷酸盐浓度的响应也是如此[61].此外,还有不少学者关注蓝细菌的碱性磷酸酶编码基因对正磷酸盐浓度的响应,其结论都表明正磷酸盐的浓度是细菌碱性磷酸酶编码基因表达的调控因素[18, 62-63, 66-70].
2.4.2 有机磷类型作为碱性磷酸酶的底物,不同种细菌的碱性磷酸酶编码基因的表达对不同类型有机磷的响应有所不同. Wu等[48]在巴氏杆菌Pasteurella multocida strain X-73的基因组中克隆出了一种phoX基因,其表达的碱性磷酸酶可分解不同类型的有机磷底物,其中亲和性最好的有机磷底物是焦磷酸钠和三磷酸腺苷. Sebastian等[21]发现不同类型的有机磷底物对Ruegeria pomeroyi DSS-3的phoX基因的表达抑制程度不同,亲和性越好的有机磷底物越能抑制phoX基因的表达,如葡萄糖六磷酸和腺苷酸,这与亲和性好的有机磷在碱性磷酸酶催化下分解效率高有关.而Harke等[63]的研究则表明,铜绿微囊藻(Microcystis aeruginosa)phoX基因的表达会受到高浓度有机磷的抑制,但不同类型有机磷对其表达的抑制效果没有显著差异,而且这种现象普遍存在于铜绿微囊藻不同的菌株中.
2.4.3 金属离子金属离子对碱性磷酸酶基因表达的影响与金属离子的种类、浓度和碱性磷酸酶基因的类型都有关.由于Zn2+与Mg2+是碱性磷酸酶PhoA的辅基,一定浓度的Zn2+与Mg2+的存在会促进细菌phoA基因的表达. Luo等[68]通过对鱼腥藻Anabaena sp. PCC7120 phoA基因表达的研究发现,1 mmol/L的Mg2+和Co2+可使PhoA的活性分别增强1.6和1.4倍,但同样浓度的Zn2+对PhoA的活性却有一定程度的抑制,这可能与phoA基因的表达对Zn2+浓度的需求较低有关.考虑到在添加乙二胺四乙酸(ethylene diamine tetraacetic acid, EDTA)螯合所有游离态金属离子后,碱性磷酸酶PhoA的活性已无法检测,可以认为金属离子是碱性磷酸酶编码基因表达的重要条件,其种类和浓度是影响碱性磷酸酶编码基因phoA表达的重要因素.
对于碱性磷酸酶PhoX而言,其基因的表达受金属离子的影响又有所不同. Wu等[48]的研究发现,在向处于磷饥饿状态的巴氏杆菌Pasteurella multocida strain X-73培养基中加入EDTA螯合金属离子后,细菌碱性磷酸酶PhoX的活性被明显抑制.但分别加入一定浓度的不同二价金属离子后,Ca2+和Co2+能重新促进phoX基因的表达,而同样浓度的Zn2+和Mg2+对PhoX活性恢复的影响有限[48]. Zaheer等[61]对根瘤菌Sinorhizobium meliloti中phoX基因表达的研究也证实Ca2+较其他二价金属离子能更好地促进PhoX的活性.
与碱性磷酸酶PhoX相似,Ca2+也是碱性磷酸酶PhoD的辅基,而Ca2+同样能很好地激发PhoD的活性[64].此外,还有一些学者也发现Fe2+和Na+等金属离子均能对细菌碱性磷酸酶编码基因的表达产生影响[14, 69].
2.4.4 pH酸碱度是细菌碱性磷酸酶活性的重要影响因子. Luo等[68]发现鱼腥藻Anabaena sp. PCC7120的phoA基因表达的最适pH值在10.5~11.5,pH值超过12.0后,已检测不出PhoA活性. Zaheer等[61]研究也证实根瘤菌Sinorhizobium meliloti的PhoX蛋白的最适pH值范围也在10~11之间.还有一些研究也将此pH值范围作为研究碱性磷酸酶编码基因表达的背景条件[18, 62, 66, 68, 71].
2.4.5 盐度细菌碱性磷酸酶编码基因的表达不仅受磷、金属离子、酸碱度等的影响,盐度可能也是重要的影响因子之一. Ishibashi等[71]在喜盐菌Halomonas sp. 593的基因组中克隆了一种碱性磷酸酶编码基因(haalp),其表达的碱性磷酸酶在低于0.3 mol/L Na+浓度的盐度环境中没有活性.同样,Kageyama等[64]发现耐盐蓝细菌Aphanothece halophytica的phoD基因的表达不仅受磷限制,也受盐度的胁迫.
2.4.6 其他环境因素此外,温度,碳、氮源等因素对纯种细菌碱性磷酸酶编码基因的表达也均有不同程度的影响[14, 63, 68].
3 产碱性磷酸酶细菌种群的研究方法解磷细菌是指能够利用环境中非生物可直接利用磷的功能细菌,包括无机磷分解细菌和有机磷分解细菌.从解磷功能上看,产碱性磷酸酶细菌属于有机磷分解细菌的范畴.迄今为止,已有大量关于海洋[72-73]、土壤[74-78]和淡水生态系统[79-83]中有机磷分解细菌的研究,其中就包括产碱性磷酸酶细菌,这些研究采用的方法主要分为传统的功能鉴定法和功能基因分子标记法.
3.1 传统的功能鉴定法传统的功能鉴定法主要是筛选环境中具有某种功能的微生物,包括样品的采集、选择性培养基分离培养和筛选,平板培养、活菌计数,最大可能计数法,纯培养细菌的特定功能与生理生化特征鉴定,16S rDNA基因系统发育分析等.这些方法以功能选择性培养基为基础,辅以系统发育分析和培养基计数手段,研究环境中具有某种功能的微生物的数量、多样性、功能活性以及生理生化特征,是经典的微生物生态学研究手段.
目前,国内外有不少学者通过传统的功能鉴定法来研究生态系统中的产碱性磷酸酶细菌. Barik等[84]利用酚酞磷酸盐琼脂(PPA)培养基对淡水养鱼塘中的产碱性磷酸酶细菌进行分离培养,并通过产碱性磷酸酶细菌菌落的形态、颜色、革兰氏染色反应、过氧化氢酶反应等对其进行种属鉴定,发现芽孢杆菌是主要的产碱性磷酸酶细菌. Bañeras等[85]采用无磷矿物培养基(MM-P),从西班牙的2个富硫湖泊中分离了四株产碱性磷酸酶的光合硫细菌,并研究了不同浓度的溶解性反应磷对其碱性磷酸酶活性的影响. Zhou等[82]利用有机磷培养基分离了6株有机磷分解细菌,通过16S rDNA基因测序,对6株细菌的系统发育关系进行了分析,并结合平板计数法和碱性磷酸酶活性,研究了这6株碱性产碱性磷酸酶细菌在太湖沉积物中的丰度及其与碱性磷酸酶活性的关系.周纯等[83]还利用传统方法分离的产碱性磷酸酶细菌,研究了其碱性磷酸酶活性对蓝藻碎屑的响应.
以上利用传统的功能鉴定法将细菌选择性分离培养与碱性磷酸磷酸酶活性结合,有助于人们认识环境中碱性磷酸酶功能细菌的种类与功能特性.然而,这种传统的方法在很大程度上受培养基成分、培养时间和菌株分离方法等的影响.加之自然环境中仅有0.001 % ~3 %的微生物可以被分离培养[86],而且多数微生物也缺乏可用于鉴定的形态特征.因此,这种传统的功能鉴定法无法准确、全面地获取自然环境中碱性磷酸酶功能细菌的多样性和生态功能等信息,更无法了解环境因子是如何影响生境中产碱性磷酸酶细菌的群落结构和功能活性.
3.2 功能基因分子标记法随着分子生物学技术的飞速发展,利用功能酶的表达基因作为分子标记物研究功能微生物的多样性、群落结构和功能活性已经在生态学中得到了广泛的应用.最初,功能基因分子标记法是建立在细菌纯培养的基础上,通过分子克隆手段将具有某种功能的细菌基因组中的某种功能基因克隆,再利用基因重组和表达,进而验证功能基因的表达与所表达的功能酶的活性特性[43-44, 48, 64, 71].这种途径也是研究纯种产碱性磷酸酶细菌及其编码基因的最常用手段.从1980s起,就有不少研究关注细菌碱性磷酸酶编码基因phoA的功能表达机制[43, 93-94],大肠杆菌是最典型的研究对象.随后,又有研究发现了其他纯种细菌基因组中的碱性磷酸酶编码基因,最重要的如枯草杆菌(Bucillus subtilis)的phoD基因[45]以及霍乱弧菌(Vibrio cholerae)的phoX基因[44],这些研究的发现为不断认识细菌碱性碱性磷酸酶及其编码基因提供了新的思路.同时,由于自然环境中细菌碱性磷酸酶的种类多,目前的研究可能还没有全面认识到细菌碱性磷酸酶及其编码基因,以纯培养细菌为基础的功能基因分子标记法还可能为我们发现更多新颖的碱性磷酸酶基因,完善细菌利用有机磷的机理研究,因此这种方法方兴未艾.
Sakurai等[60]首次针对部分碱性磷酸酶编码基因设计了寡核苷酸探针,利用PCR-DGGE指纹图谱的手段研究了施肥土壤中碱性磷酸酶编码基因的多样性与系统发育关系,证明了碱性磷酸酶编码基因可以作为分子标记物研究环境中多样的产碱性磷酸酶细菌. Sebastian等[14]也根据细菌碱性磷酸酶编码基因phoX的序列,设计了兼并性核苷酸引物,同样从寡营养的海洋中直接扩增出了phoX基因,并通过系统发育分析发现这些基因与不同门类的纯种细菌phoX基因具有相似性.而后,Chhabra等[65]也利用Sakurai等[60]设计的引物研究了大麦根系碱性磷酸酶功能细菌的群落结构.鉴于碱性磷酸酶编码基因phoD基因在土壤生态系统中可能占有较大的比例,Tan等[50]设计了针对土壤phoD基因的寡核苷酸探针,利用DGGE和高通量测序的手段,研究了施肥对于土壤碱性磷酸酶功能细菌的群落结构和多样性的影响.在湖泊细菌碱性磷酸酶基因型多样性方面,Dai等[95-96]借助上述phoX基因序列引物研究了太湖水体细菌碱性磷酸酶编码基因phoX的基因型及其相关的环境因素,首次发现了太湖水生态系统中丰富而新颖的细菌碱性磷酸酶基因,从生物学角度证明了细菌碱性磷酸酶在太湖有机磷矿化过程中的重要作用.同样,Valdespino-Castillo等[97]也通过细菌碱性磷酸酶编码基因引物研究了墨西哥Alchichica湖的产碱性磷酸酶细菌种群.这些研究通过设计碱性磷酸酶编码基因的寡核苷酸探针,为全面研究环境中碱性磷酸酶功能细菌提供了新的途径.
碱性磷酸酶的编码基因多样,环境细菌的碱性磷酸酶编码基因序列间的保守性通常又较差,因此很难设计通用的针对所有细菌碱性磷酸酶编码基因的寡核苷酸探针,也就无法准确、全面地认识环境中的产碱性磷酸酶细菌及其功能.与DNA指纹图谱技术相比,分析已有的细菌宏基因组和宏转录组数据库能够更为全面的认识环境中的碱性磷酸酶及其编码基因.
近年来,随着环境宏基因组学研究的深入,人们已经积累了一些生态系统中的宏基因组和宏转录组数据库,例如全球海洋调查基因组数据库(Global Ocean Sampling, GOS)[13-14, 98]、美国国家生物信息中心数据库中non-redundant蛋白数据库以及寡营养海洋(North Pacific Subtropical Gyre, NPSG)宏基因组和宏转录组数据库[99].学者们借助这些数据库,通过生物信息学分析手段,研究了海洋和土壤生态系统中碱性磷酸酶的种类,碱性磷酸酶编码基因的多样性、相对比例、系统发育关系、功能表达以及与环境因素之间的关联. Sebastian与Ammerman[14]通过对NPSG宏基因组数据库和GOS宏基因组数据库的研究发现,海洋细菌碱性磷酸编码基因phoX的丰度要显著高于phoA基因,而且phoX在细菌不同门类中的分布要比phoA更为广泛.同时,通过对海岸带宏转录组数据库[98]研究推测,在富营养化的水体中碱性磷酸酶PhoA较PhoX分布更广.随后,Luo等[13, 20]通过对全球海洋调查的基因组数据研究更为清晰地认识了海洋中3种碱性磷酸酶的相对比例、亚细胞分布和系统发育关系.这些研究都为全面认识不同环境中产碱性磷酸酶细菌提供了新的途径.
4 研究展望在寡营养的海洋生态系统中,处于“磷饥饿”状态的细菌通过碱性磷酸酶利用水体中的有机磷,维持海洋生态系统的生产力和物质循环.因此,碱性磷酸酶及其功能细菌在海洋生态系统中得到了广泛的关注.现有的研究通过海洋宏基因组数据库,对海洋生态系统中细菌碱性磷酸酶及其编码基因开展深入研究,从微生物学角度阐释了海洋生态系统有机磷矿化与循环的机制.实际上,湖泊生态系统中也普遍存在磷限制,细菌碱性磷酸酶在湖泊有机磷矿化中的作用已经得到了证实[26, 42, 80].然而,目前对于湖泊生态系统中碱性磷酸酶功能细菌的研究方法还主要依靠传统的功能鉴定法,加之缺乏针对淡水生态系统细菌的宏基因组数据库,使得人们对于湖泊生态系统中产碱性磷酸酶细菌及其编码基因的认识远滞后于海洋生态系统.因此,急需构建针对典型湖泊生态系统的细菌宏基因组数据库,运用生物信息学技术,分析湖泊生态系统中细菌碱性磷酸酶的种类及其对应的功能细菌特征,不仅可对比富营养化淡水与寡营养海洋生态系统细菌碱性磷酸酶的差异,还可为深入研究淡水生态系统细菌碱性磷酸酶及编码基因奠定基础.
目前,也有零星的关于湖泊生态系统中细菌碱性磷酸酶编码基因的研究报道[63, 95-97],但还是缺乏全面认识湖泊生态系统中产碱性磷酸酶细菌的多样性、丰度及与主要环境因子间关系的研究.对于一些关键的科学问题,如湖泊生态系统中有哪些主要的产碱性磷酸酶细菌与编码基因?其与海洋和土壤生态系统中碱性磷酸酶编码基因的理论有何异同?影响湖泊中产碱性磷酸酶细菌及其编码基因分布的主导环境因素又有哪些?同时,在我国长江中下游地区,绝大多数的湖泊为浅水湖泊,而这些浅水湖泊大多面临富营养化问题[100].蓝藻水华频发是富营养化浅水湖泊所面临的最严重的生态问题.产碱性磷酸酶细菌与蓝藻水华的频繁爆发之间是否有联系?有着何种联系?对上述问题的解答是揭示富营养化浅水湖泊中产碱性磷酸酶细菌及其编码基因的分布、生态功能以及与湖泊环境因素之间关系的关键所在,目前,在富营养化湖泊生态系统中,此类问题的研究还相对缺乏.
| [1] |
Mills MM, Ridame C, Davey M et al. Iron and phosphorus co-limit nitrogen fixation in the eastern tropical North Atlantic. Nature, 2004, 429(6989): 292-294. DOI:10.1038/nature02550 |
| [2] |
Thingstad T, Krom M, Mantoura R et al. Nature of phosphorus limitation in the ultraoligotrophic eastern Mediterranean. Science, 2005, 309(5737): 1068-1071. DOI:10.1126/science.1112632 |
| [3] |
Krom M, Thingstad T, Brenner S et al. Summary and overview of the CYCLOPS P addition Lagrangian experiment in the Eastern Mediterranean. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2005, 52(22): 3090-3108. |
| [4] |
Pulido-Villena E, Rérolle V, Guieu C. Transient fertilizing effect of dust in P-deficient LNLC surface ocean. Geophysical Research Letters, 2010, 37(1). |
| [5] |
Giovannoni SJ, Tripp HJ, Givan S et al. Genome streamlining in a cosmopolitan oceanic bacterium. Science, 2005, 309(5738): 1242-1245. DOI:10.1126/science.1114057 |
| [6] |
Van Mooy BA, Rocap G, Fredricks HF et al. Sulfolipids dramatically decrease phosphorus demand by picocyanobacteria in oligotrophic marine environments. Proceedings of the National Academy of Sciences, 2006, 103(23): 8607-8612. DOI:10.1073/pnas.0600540103 |
| [7] |
Polz MF, Hunt DE, Preheim SP et al. Patterns and mechanisms of genetic and phenotypic differentiation in marine microbes. Philosophical Transactions of the Royal Society B:Biological Sciences, 2006, 361(1475): 2009-2021. DOI:10.1098/rstb.2006.1928 |
| [8] |
Ammerman JW. Role of ecto-phosphohydrolases in phosphorus regeneration in estuarine and coastal ecosystems. In:Chróst RJ, ed. Microbial Enzymes in Aquatic Environments. New York:Springer, 1991:165-186.
|
| [9] |
Siuda W, Chrost R. Utilization of selected dissolved organic phosphorus compounds by bacteria in lake water under non-limiting orthophosphate conditions. Polish Journal of Environmental Studies, 2001, 10(6): 475-484. |
| [10] |
Shan Y, Mckelvie ID, Hart BT. Determination of alkaline phosphatase-hydrolyzable phosphorus in natural water systems by enzymatic flow injection. Limnology and Oceanography, 1994, 39(8): 1993-2000. DOI:10.4319/lo.1994.39.8.1993 |
| [11] |
Michaels AF, Silver MW. Primary production, sinking fluxes and the microbial food web. Deep Sea Research Part A. Oceanographic Research Papers, 1988, 35(4): 473-490. |
| [12] |
Chróst RJ, Overbeck J. Kinetics of alkaline phosphatase activity and phosphorus availability for phytoplankton and bacterioplankton in lake plußsee(North German Eutrophic Lake). Microbial Ecology, 1987, 13(3): 229-248. DOI:10.1007/BF02025000 |
| [13] |
Luo H, Benner R, Long R et al. Subcellular localization of marine bacterial alkaline phosphatases. Proceedings of the National Academy of Sciences, 2009, 106(50): 212-219. |
| [14] |
Sebastian M, Ammerman J. The alkaline phosphatase PhoX is more widely distributed in marine bacteria than the classical PhoA. The ISME Journal, 2009, 3(5): 563-572. DOI:10.1038/ismej.2009.10 |
| [15] |
White A. New insights into bacterial acquisition of phosphorus in the surface ocean. Proceedings of the National Academy of Sciences, 2009, 106(50): 210-213. |
| [16] |
Duhamel S, Dyhrman ST, Karla DM. Alkaline phosphatase activity and regulation in the North Pacific Subtropical Gyre. Limnology and Oceanography, 2010, 55(3): 1414-1425. DOI:10.4319/lo.2010.55.3.1414 |
| [17] |
Ivancic I, Fuks D, Radic T et al. Phytoplankton and bacterial alkaline phosphatase activity in the northern Adriatic Sea. Marine Environmental Research, 2010, 69(2): 85-94. DOI:10.1016/j.marenvres.2009.08.004 |
| [18] |
Kathuria S, Martiny AC. Prevalence of a calcium-based alkaline phosphatase associated with the marine cyanobacterium Prochlorococcus and other ocean bacteria. Environmental Microbiology, 2011, 13(1): 74-83. DOI:10.1111/emi.2011.13.issue-1 |
| [19] |
Luo C, Konstantinidis KT. Phosphorus-related gene content is similar in Prochlorococcus populations from the North Pacific and North Atlantic Oceans. Proceedings of the National Academy of Sciences, 2011, 108(16): 64-66. DOI:10.1073/pnas.1102448108 |
| [20] |
Luo H, Zhang H, Long RA et al. Depth distributions of alkaline phosphatase and phosphonate utilization genes in the North Pacific Subtropical Gyre. Aquatic Microbial Ecology, 2011, 62(1): 61-69. DOI:10.3354/ame01458 |
| [21] |
Sebastian M, Ammerman J. Role of the phosphatase PhoX in the phosphorus metabolism of the marine bacterium Ruegeria pomeroyi DSS-3. Environmental Microbiology Reports, 2011, 3(5): 535-542. DOI:10.1111/emi4.2011.3.issue-5 |
| [22] |
Kutovaya OA. Using molecular probes to detect cyanobacterial communities and phosphorus utilization in the Great Lakes. Ohio:Bowling Green State University, 2012. |
| [23] |
Zhou Yiyong, Li Jianjiu, Zhang Min et al. Distribution of kinetic parameters of alkaline phosphatase in sediments of shallow lakes. J Lake Sci, 2001, 13(3): 261-266. [周易勇, 李建秋, 张敏等. 浅水湖泊中沉积物碱性磷酸酶动力学参数的分布. 湖泊科学, 2001, 13(3): 261-266. DOI:10.18307/20010310.] |
| [24] |
Gao Guang, Qin Boqiang, Zhu Guangwei et al. Seasonal variation of alkaline phosphatase activity in Meiliang Bay, Lake Taihu. J Lake Sci, 2004, 16(3): 245-251. [高光, 秦伯强, 朱广伟等. 太湖梅梁湾中碱性磷酸酶的活性及其与藻类生长的关系. 湖泊科学, 2004, 16(3): 245-251. DOI:10.18307/2004.0309.] |
| [25] |
Gao G, Zhu G, Qin B et al. Alkaline phosphatase activity and the phosphorus mineralization rate of Lake Taihu. Science in China Series D:Earth Sciences, 2006, 49(I): 176-185. |
| [26] |
Song C, Cao X, Li J et al. Contributions of phosphatase and microbial activity to internal phosphorus loading and their relation to lake eutrophication. Science in China Series D:Earth Sciences, 2006, 49(I): 102-113. |
| [27] |
Xia Zhuoying, Chen Fang, Song Chunlei et al. A study on distribution and roles of alkaline phosphatase in sediments of some lakes in the middle and lower reaches of the Yantze River. Acta Hydrobiologia Sinica, 2007, 31(1): 9-17. [夏卓英, 陈芳, 宋春雷等. 长江中下游部分湖泊沉积物碱性磷酸酶分布及其作用研究. 水生生物学报, 2007, 31(1): 9-17.] |
| [28] |
Zhou Y, Song C, Cao X et al. Phosphorus fractions and alkaline phosphatase activity in sediments of a large eutrophic Chinese lake(Lake Taihu). Hydrobiologia, 2008, 599(1): 119-125. DOI:10.1007/s10750-007-9185-z |
| [29] |
Deng JJ, Huang XF, Hu JW et al. Distribution of several microorganisms and activity of alkaline phosphatase in sediments from Baihua Lake. Asia-Pacific Journal of Chemical Engineering, 2009, 4(5): 711-716. DOI:10.1002/apj.323 |
| [30] |
Lu Na, Hu Weiping, Deng Jiancai et al. Spatial distribution characteristics and ecological significance of alkaline phosphatase in water column of Taihu Lake. Environmental Science, 2009, 30(10): 2897-2903. [路娜, 胡维平, 邓建才等. 太湖水体中碱性磷酸酶的空间分布及生态意义. 环境科学, 2009, 30(10): 2897-2903.] |
| [31] |
Manna S, Som A, Samanta S. Water alkaline phosphatase activity andphosphorus availability during summer ininland water bodies. Indian Journal of Fisheries, 2011, 53(2): 167-173. |
| [32] |
Mamatha S, Gobika A, Janani P. Phosphate solubilizing bacteria and alkaline phosphatase activity in coastal waters off Trivandrum. Journal of Costal Environment, 2012, 3(1): 89-100. |
| [33] |
Xia ZY, Zhou YY, Chen F et al. Stratification of alkaline phosphatase in sediments of two urban lakes and its effect on phosphorus cycle. Acta Ecologica Sinica, 2012, 32(3): 138-143. DOI:10.1016/j.chnaes.2012.02.006 |
| [34] |
Zhao G, Du J, Jia Y et al. The importance of bacteria in promoting algal growth in eutrophic lakes with limited available phosphorus. Ecological Engineering, 2012, 42: 107-111. DOI:10.1016/j.ecoleng.2012.02.007 |
| [35] |
Wang Sumin, Dou Hongshen. Chinese Lakes. Beijing: Science Press, 1998. [王苏民, 窦鸿身. 中国湖泊志. 北京: 科学出版社, 1998.]
|
| [36] |
Zhou Yiyong, Fu Yongqing. Phosphatases in natural water:origin, characteristics and ecological significance. J Lake Sci, 1999, 11(3): 274-282. [周易勇, 付永清. 水体磷酸酶:来源, 特征及其生态学意义. 湖泊科学, 1999, 11(3): 274-282. DOI:10.18307/1999.0313.] |
| [37] |
Song Chunlei. The role of dissolved phosphatase and microbial activity in the process of lake eutrophication. Wuhan:Institute of Hydrobiology, Chinese Academy of Sciences, 2005. [宋春雷. 溶解态磷酸酶与微生物活性在湖泊富营养化过程中的作用. 武汉: 中国科学院水生生物研究所, 2005.] |
| [38] |
Xu J. Microbial ecology in the age of genomics and metagenomics:concepts, tools, and recent advances. Molecular Ecology, 2006, 15(7): 1713-1731. DOI:10.1111/j.1365-294X.2006.02882.x |
| [39] |
Li H, Veldhuis MJ, Post AF. Alkaline phosphatase activities among planktonic communities in the northern Red Sea. Marine Ecology Progress Series, 1998, 173: 107-115. DOI:10.3354/meps173107 |
| [40] |
Chróst RJ. Environmental control of the synthesis and activity of aquatic microbial ectoenzymes.In:Chróst RJ ed. Microbial Enzymes in Aquatic Environments. New York:Springer, 1991:29-59.
|
| [41] |
Zaccone R, Caruso G, Cali C. Heterotrophic bacteria in the northern Adriatic Sea:seasonal changes and ectoenzyme profile. Marine Environmental Research, 2002, 54(1): 1-19. DOI:10.1016/S0141-1136(02)00089-2 |
| [42] |
Labry C, Delmas D, Herbland A. Phytoplankton and bacterial alkaline phosphatase activities in relation to phosphate and DOP availability within the Gironde plume waters(Bay of Biscay). Journal of Experimental Marine Biology and Ecology, 2005, 318(2): 213-225. DOI:10.1016/j.jembe.2004.12.017 |
| [43] |
Wanner B. Gene regulation by phosphate in enteric bacteria. Journal of Cellular Biochemistry, 2004, 51(1): 47-54. |
| [44] |
Majumdar A, Ghatak A, Ghosh RK. Identification of the gene for the monomeric alkaline phosphatase of Vibrio cholerae serogroup O1 strain. Gene, 2005, 344: 251-258. DOI:10.1016/j.gene.2004.11.005 |
| [45] |
Eder S, Shi L, Jensen K et al. A Bacillus subtilis secreted phosphodiesterase/alkaline phosphatase is the product of a Pho regulon gene, phoD. Microbiology, 1996, 142(8): 2041-2047. DOI:10.1099/13500872-142-8-2041 |
| [46] |
Yong SC, Roversi P, Lillington J et al. A complex iron-calcium cofactor catalyzing phosphotransfer chemistry. Science, 2014, 345(6201): 1170-1173. DOI:10.1126/science.1254237 |
| [47] |
Monds RD, Newell PD, Gross RH et al. Phosphate-dependent modulation of c-di-GMP levels regulates Pseudomonas fluorescens Pf0-1 biofilm formation by controlling secretion of the adhesin LapA. Molecular Microbiology, 2007, 63(3): 656-679. |
| [48] |
Wu JR, Shien JH, Shien HK et al. Cloning of the gene and characterization of the enzymatic properties of the monomeric alkaline phosphatase(PhoX) from Pasteurella multocida strain X-73. FEMS Microbiology Letters, 2007, 267(1): 113-120. DOI:10.1111/fml.2007.267.issue-1 |
| [49] |
Van Mourik A, Bleumink-Pluym NM, Van Dijk L et al. Functional analysis of a Campylobacter jejuni alkaline phosphatase secreted via the Tat export machinery. Microbiology, 2008, 154(2): 584-592. DOI:10.1099/mic.0.2007/012120-0 |
| [50] |
Tan H, Barret M, Mooij MJ et al. Long-term phosphorus fertilisation increased the diversity of the total bacterial community and the phoD phosphorus mineraliser group in pasture soils. Biology and Fertility of Soils, 2012, 49(6): 661-672. |
| [51] |
Chaffin DO, Rubens CE. Blue/white screening of recombinant plasmids in Gram-positive bacteria by interruption of alkaline phosphatase gene(phoZ) expression. Gene, 1998, 219(1/2): 91-99. |
| [52] |
Wagner KU, Masepohl B, Pistorius EK. The cyanobacterium Synechococcus sp. strain PCC 7942 contains a second alkaline phosphatase encoded by phoV. Microbiology, 1995, 141(12): 3049-3058. DOI:10.1099/13500872-141-12-3049 |
| [53] |
Thompson LM, Macleod RA. Biochemical localization of alkaline phosphatase in the cell wall of a marine pseudomonad. Journal of Bacteriology, 1974, 117(2): 819-825. |
| [54] |
Doonan B, Jensen T. Ultrastructural localization of alkaline phosphatase in the blue-green bacterium Plectonema boryanum. Journal of Bacteriology, 1977, 132(3): 967-973. |
| [55] |
Hassan H, Pratt D. Biochemical and physiological properties of alkaline phosphatases in five isolates of marine bacteria. Journal of Bacteriology, 1977, 129(3): 1607-1612. |
| [56] |
Poole K, Hancock RE. Secretion of alkaline phosphatase and phsopholipase C in Pseudomonas aeruginosa is specific and does not involve an increase in outer membrane permeability. FEMS Microbiology Letters, 1983, 16(1): 25-29. DOI:10.1111/fml.1983.16.issue-1 |
| [57] |
Von Tigerstrom R. Production of two phosphatases by Lysobacter enzymogenes and purification and characterization of the extracellular enzyme. Applied and Environmental Microbiology, 1984, 47(4): 693-698. |
| [58] |
Hermann M, Saunders AM, Schramm A. Effect of lake trophic status and rooted macrophytes on community composition and abundance of ammonia-oxidizing prokaryotes in freshwater sediments. Applied and Environmental Microbiology, 2009, 75(10): 3127-3136. DOI:10.1128/AEM.02806-08 |
| [59] |
Wu Y, Xiang Y, Wang J et al. Heterogeneity of archaeal and bacterial ammonia-oxidizing communities in Lake Taihu, China. Environmental Microbiology Reports, 2010, 2(4): 569-576. DOI:10.1111/emi4.2010.2.issue-4 |
| [60] |
Sakurai M, Wasaki J, Tomizawa Y et al. Analysis of bacterial communities on alkaline phosphatase genes in soil supplied with organic matter. Soil science and Plant Nutrition, 2008, 54(1): 62-71. DOI:10.1111/j.1747-0765.2007.00210.x |
| [61] |
Zaheer R, Morton R, Proudfoot M et al. Genetic and biochemical properties of an alkaline phosphatase PhoX family protein found in many bacteria. Environmental Microbiology, 2009, 11(6): 1572-1587. DOI:10.1111/emi.2009.11.issue-6 |
| [62] |
Orchard ED, Webb EA, Dyhrman ST. Molecular analysis of the phosphorus starvation response in Trichodesmium spp. Environmental Microbiology, 2009, 11(9): 2400-2411. DOI:10.1111/emi.2009.11.issue-9 |
| [63] |
Harke MJ, Berry DL, Ammerman JW et al. Molecular response of the bloom-forming cyanobacterium, Microcystis aeruginosa, to phosphorus limitation. Microbial Ecology, 2012, 63(1): 188-198. DOI:10.1007/s00248-011-9894-8 |
| [64] |
Kageyama H, Tripathi K, Rai AK et al. An alkaline phosphatase/phosphodiesterase, PhoD, induced by salt stress and secreted out of the cells of Aphanothece halophytica, a halotolerant cyanobacterium. Applied and Environmental Microbiology, 2011, 77(15): 5178-5183. DOI:10.1128/AEM.00667-11 |
| [65] |
Chhabra S, Brazil D, Morrissey J et al. Fertilization management affects the alkaline phosphatase bacterial community in barley rhizosphere soil. Biology and Fertility of Soils, 2012, 49(1): 31-39. |
| [66] |
Liu Z, Wu C. Response of alkaline phosphatases in the cyanobacterium Anabaena sp. FACHB 709 to inorganic phosphate starvation. Current Microbiology, 2012, 64(6): 524-529. DOI:10.1007/s00284-012-0101-z |
| [67] |
Adams M, Gomez-Garcia M, Grossman A et al. Phosphorus deprivation responses and phosphonate utilization in a thermophilic Synechococcus sp. from microbial mats. Journal of Bacteriology, 2008, 190(24): 8171-8184. DOI:10.1128/JB.01011-08 |
| [68] |
Luo M, Guo YC, Deng JY et al. Characterization of a monomeric heat-labile classical alkaline phosphatase from Anabaena sp. PCC7120. Biochemistry, 2010, 75(5): 655-664. |
| [69] |
Muñoz-Martín MÁ, Mateo P, Leganés F et al. Novel cyanobacterial bioreporters of phosphorus bioavailability based on alkaline phosphatase and phosphate transporter genes of Anabaena sp. PCC 7120. Analytical and Bioanalytical Chemistry, 2011, 400(10): 3573-3584. DOI:10.1007/s00216-011-5017-0 |
| [70] |
Asencio AD, Morte A, García-Carmona F et al. Partial purification and characterization of a calcium-dependent alkaline phosphatase from the cyanobacterium Arthrospira platensis. Journal of Phycology, 2012, 48(2): 347-354. DOI:10.1111/jpy.2012.48.issue-2 |
| [71] |
Ishibashi M, Odak K, Arakawa T et al. Cloning, expression, purification and activation by Na ion of halophilic alkaline phosphatase from moderate halophile Halomonas sp. 593. Protein Expression and Purification, 2011, 76(1): 97-102. DOI:10.1016/j.pep.2010.09.013 |
| [72] |
Venkateswaran K, Natarajan R. Seasonal distribution of inorganic phosphate solubilising bacteria and phosphatase producing bacteria in Porto Novo waters. Indian Journal of Marine Sciences, 1983, 12: 213-217. |
| [73] |
Seshadri S, Ignacimuthu S, Lakshminarsimhan C. Variations in hetetrophic and phosphate solubilizing bacteria from Chennai, southeast coast of India. Indian Journal of Marine Sciences, 2002, 31(1): 69-72. |
| [74] |
Rodríguez H, Fraga R. Phosphate solubilizing bacteria and their role in plant growth promotion. Biotechnology Advances, 1999, 17(4): 319-339. |
| [75] |
Dubey S. Associative effect of nitrogen-fixing and phosphate-solubilizing bacteria in rainfed soybean(Glycine max) grown in Vertisols. Indian Journal of Agricultural Science, 2001, 71(7): 476-479. |
| [76] |
Zhao Xiaorong, Lin Qimei, Sun Yanxin et al. Phosphobacteria distribution in rhizophere and nonrhizophere soil of corn. Chinese Journal of Ecology, 2001, 20(6): 62-64. [赵小蓉, 林启美, 孙焱鑫等. 玉米根际与非根际解磷细菌的分布特点. 生态学杂志, 2001, 20(6): 62-64.] |
| [77] |
Hao Jing, Hong Jianping, Liu Bing et al. Isolation, screening and combination of highly-effective phosphorus solubilizing bacterial strains in calcareous soil. Chinese Journal of Applied & Environmental Biology, 2006, 12(3): 404-408. [郝晶, 洪坚平, 刘冰等. 石灰性土壤中高效解磷细菌菌株的分离, 筛选及组合. 应用与环境生物学报, 2006, 12(3): 404-408.] |
| [78] |
Yu X, Liu X, Zhu TH et al. Isolation and characterization of phosphate-solubilizing bacteria from walnut and their effect on growth and phosphorus mobilization. Biology and Fertility of Soils, 2011, 47(4): 437-446. DOI:10.1007/s00374-011-0548-2 |
| [79] |
Wu GF, Zhou XP. Characterization of phosphorus-releasing bacteria in a small eutrophic shallow lake, Eastern China. Water Research, 2005, 39(19): 4623-4632. DOI:10.1016/j.watres.2005.08.036 |
| [80] |
Song Wei, Yuan Lina, Xiao Lin et al. ALPase activity and the distribution of phosphate solubilizing bacteria and the relationship between them in sediments of Lake Taihu. Chinese Journal of Environmental Science, 2007, 28(10): 2355-2360. [宋炜, 袁丽娜, 肖琳等. 太湖沉积物中解磷细菌分布及其与碱性磷酸酶活性的关系. 环境科学, 2007, 28(10): 2355-2360. DOI:10.3321/j.issn:0250-3301.2007.10.035] |
| [81] |
Feng Sheng, Qin Boqiang, Gao Guang. The relationships between phosphorus-transmuting bacteria and phosphorus forms inLake Taihu. J Lake Sci, 2008, 20(4): 428-436. [冯胜, 秦伯强, 高光. 太湖磷转化细菌与水体磷形态关系. 湖泊科学, 2008, 20(4): 428-436. DOI:10.18307/2008.0404.] |
| [82] |
Zhou C, Song C, Huang D et al. Isolation and characterization of organic phosphorus-mineralizing bacteria in sediment of a chinese large shallow eutrophic lake(Lake Taihu). Geomicrobiology Journal, 2011, 28(8): 660-666. DOI:10.1080/01490451.2010.511438 |
| [83] |
Zhou Chun, Song Chunlei, Cao Xiuyun et al. Responses of extracellular alkaline phosphatase activity in different organic phosphorus mineralizing bacteria strains isolated from Lake Taihu to the cyanobacterium detritus. Acta Hydrobiologia Sinica, 2012, 36(1): 119-125. [周纯, 宋春雷, 曹秀云等. 太湖不同解有机磷菌株胞外碱性磷酸酶活性对蓝藻碎屑的响应. 水生生物学报, 2012, 36(1): 119-125.] |
| [84] |
Barik S, Purushothaman C, Mohanty A. Phosphatase activity with reference to bacteria and phosphorus in tropical freshwater aquaculture pond systems. Aquaculture Research, 2001, 32(10): 819-832. DOI:10.1046/j.1355-557x.2001.00619.x |
| [85] |
Bañeras L, Ros-Ponsatí M, Cristina XP et al. Phosphorus deficiency and kinetics of alkaline phosphatase in isolates and natural populations of phototrophic sulphur bacteria. FEMS Microbiology Ecology, 2010, 73(2): 243-253. |
| [86] |
Amann RI, Ludwig W, Schleifer KH. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiological Reviews, 1995, 59(1): 143-169. |
| [87] |
Morris JJ, Kirkegaard R, Szul MJ et al. Facilitation of robust growth of Prochlorococcus colonies and dilute liquid cultures by "helper" heterotrophic bacteria. Applied and Environmental Microbiology, 2008, 74(14): 4530-4534. DOI:10.1128/AEM.02479-07 |
| [88] |
Nichols D, Lewis K, Orjala J et al. Short peptide induces an "uncultivable" microorganism to grow in vitro. Applied and Environmental Microbiology, 2008, 74(15): 4889-4897. DOI:10.1128/AEM.00393-08 |
| [89] |
Franklin RB, Garland JL, Bolster CH et al. Impact of dilution on microbial community structure and functional potential:comparison of numerical simulations and batch culture experiments. Applied and Environmental Microbiology, 2001, 67(2): 702-712. DOI:10.1128/AEM.67.2.702-712.2001 |
| [90] |
Kaeberlein T, Lewis K, Epstein SS. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127-1129. DOI:10.1126/science.1070633 |
| [91] |
Stevenson BS, Eichorst SA, Wertz JT et al. New strategies for cultivation and detection of previously uncultured microbes. Applied and Environmental Microbiology, 2004, 70(8): 4748-4755. DOI:10.1128/AEM.70.8.4748-4755.2004 |
| [92] |
Mehta S, Nautiyal CS. An efficient method for qualitative screening of phosphate-solubilizing bacteria. Current Microbiology, 2001, 43(1): 51-56. DOI:10.1007/s002840010259 |
| [93] |
Kim EE, Wyckoff HW. Reaction mechanism of alkaline phosphatase based on crystal structures:Two-metal ion catalysis. Journal of Molecular Biology, 1991, 218(2): 449-464. DOI:10.1016/0022-2836(91)90724-K |
| [94] |
Coleman JE. Structure and mechanism of alkaline phosphatase. Annual Review of Biophysics and Biomolecular Structure, 1992, 21(1): 441-483. DOI:10.1146/annurev.bb.21.060192.002301 |
| [95] |
Dai JY, Chen D, Gao G et al. Recovery of novel alkaline phosphatase-encoding genes(phoX) from eutrophic Lake Taihu. Canadian Journal of Microbiology, 2014, 60: 167-171. DOI:10.1139/cjm-2013-0755 |
| [96] |
Dai JY, Chen D, Wu SQ et al. Comparative analysis of alkaline phosphatase-encoding genes(phoX) in two contrasting zones of Lake Taihu. Canadian Journal of Microbiology, 2015, 61: 227-236. DOI:10.1139/cjm-2014-0446 |
| [97] |
Valdespino-Castillo PM, Alcántara-Hernández RJ, Alcocer J et al. Alkaline phosphatases in microbialites and bacterioplankton from Alchichica soda lake, Mexico. FEMS Microbiology Ecology, 2014, 90: 504-519. |
| [98] |
Gilbert JA, Field D, Huang Y et al. Detection of large numbers of novel sequences in the metatranscriptomes of complex marine microbial communities. PLoS One, 2008, 3(8): e3042. DOI:10.1371/journal.pone.0003042 |
| [99] |
Frias-Lopez J, Shi Y, Tyson GW et al. Microbial community gene expression in ocean surface waters. Proceedings of the National Academy of Sciences, 2008, 105(10): 3805-3810. DOI:10.1073/pnas.0708897105 |
| [100] |
Qin Boqiang. Approaches to mechanisms and control of eutrophication of shallow lakes in the middle and lower reaches of the Yangze River. J Lake Sci, 2002, 14(3): 193-202. [秦伯强. 长江中下游浅水湖泊富营养化发生机制与控制途径初探. 湖泊科学, 2002, 14(3): 193-202. DOI:10.18307/2002.0301.[] |
 2016, Vol. 28
2016, Vol. 28 

