(2: 环境保护部南京环境科学研究所, 南京 210042)
(3: 方城县水利局, 南阳 473200)
(2: Nanjing Institute of Environmental Sciences, Ministry of Environmental Protection, Nanjing 210042, P. R. China)
(3: Fangcheng County Water Conservancy Bureau, Nanyang 473200, P. R. China)
湖泊沉积物微生物通过同化、异化等代谢过程来影响湖泊生态系统中的沉积物物质循环、污染物释放等生物地球化学循环过程,是水生态系统的重要组成部分[1-3].因此,全面了解湖泊生态系统中沉积物微生物群落组成、分布特征及其在生态系统中的功能和作用,对于管理和维护湖泊生态环境具有深远的意义[2, 4].高通量测序技术(High throughput sequencing)能高通量得到特定的DNA片段,全面地展示生物群落的组成结构[5],目前国内外研究者已采用该方法研究沉积物微生物群落组成、分布特征及其影响因素等[6-10].研究表明沉积物中的环境因子,包括深度、有机质、总氮、总磷含量和pH值等均能不同程度影响沉积物细菌的群落组成[2, 6-7].黄媛等[9]采用高通量测序技术对杭州西溪湿地4种不同植被下沉积物的细菌多样性进行了研究. Liu等[7]研究表明,pH值是影响钱塘江沉积物细菌群落的最主要因素. Song等[11]对东平湖沉积物细菌群落分析发现,总磷是影响细菌群落的主要因素.之前高通量测序数据主要关注于微生物群落结构(α和β多样性),无法开展其功能研究. PICRUSt(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)是一款菌群功能和代谢的预测软件,可以通过16S rRNA基因序列预测对应的细菌和古菌的代谢功能谱[12].目前,PICRUSt功能预测分析在沉积物微生物研究中应用较少[13-14],其与高通量测序技术结合分析,将为探明沉积物微生物群落组成和功能提供重要帮助.
丹江口水库作为南水北调中线工程核心水源区,其水质状况直接关系到受水区居民的饮水安全.鉴于湖泊沉积物微生物在湖泊生态系统中的重要作用,开展丹江口水库沉积物微生物研究具有重要意义,目前有关丹江口库区沉积物细菌群落和功能研究鲜见报道.本研究选取丹江库区5个典型生态点位,采用高通量测序技术对表层沉积物细菌群落多样性进行分析,研究其群落组成和分布特征,结合PICRUSt分析预测表层沉积物细菌功能,以期探明丹江口库区表层沉积物细菌群落组成和功能,为丹江口水库水环境保护提供参考依据.
1 材料与方法 1.1 样品采集与理化指标测定根据丹江口水库(32°36′~33°48′N,110°59′~111°49′E)的地理位置特征和人类活动影响程度,选取属于河南省南阳市的丹江库区库心(样品编号为K)、渠首(Q)、黑鸡嘴(H)、宋岗(S)和台子山(T)5个点位为采样点,采样点具体信息见表 1.于2017年5月初采用抓斗式采泥器采集沉积物样品,将收集的表层(0~5 cm)沉积物置于预先灭菌容器中存于冰盒,运回实验室用于提取细菌总DNA,剩余沉积物用于理化指标测定.所采沉积物样品带回实验室于阴凉处室温风干,参照《土壤农业化学分析方法》[15]测定沉积物pH值、有机质总量(SOM)、总氮(TN)、总磷(TP)、总钾(TK)、铵态氮(NH4+-N)和硝态氮(NO3--N)含量等理化指标,每个样品设置3个重复.
| 表 1 采样点分布 Tab.1 Location of the sample sites |
表层沉积物细菌总DNA采用美国MP公司FastDNA© Spin Kit for Soil试剂盒提取,提取步骤参照说明书,每个样品设置3个重复.将提取得到的沉积物总DNA通过微量紫外分光光度计(NanoDrop© ND-1000,Wilmington,DE,USA)测定DNA浓度和纯度.
1.3 高通量测序采用通用引物(338F/806R)对细菌16S rRNA基因的V3~V4区进行PCR扩增,扩增体系为5×FastPfu Buffer 4 μl、2.5 mmol/L dNTPs 2 μl、Forward Primer(5 μmol/L) 0.8 μl、Reverse Primer(5 μmol/L) 0.8 μl、FastPfu Polymerase 0.4 μl、DNA模板10 ng,补充ddH2O至20 μl. PCR扩增的反应体系为: 94℃,5 min;30×(94℃,30 s;54℃,30 s;72℃ 45 s);72℃ 10 min[16-17].修饰后的通用引物含有不同的Tag标签用以区分不同样品,利用上海美吉生物医药科技有限公司的MiSeq PE300测序仪(Illumina Inc,San Diego,CA,USA)完成序列测定.
1.4 数据分析 1.4.1 高通量数据分析高通量数据的生物信息学分析采用QIIME(quantitative insights into microbial ecology)进行,根据序列的相似度,将序列归为多个OTU(operational taxonomic unit),OTU产出后,统计各个样品含有OTU情况及每个OTU中含有序列的数目,得到每个OTU的分类学信息[18].选取相似度在97 %条件下的OTU生成预期的稀释曲线[19].利用PCoA、Correlation聚类法分别进行数据处理、细菌群落分布、主成分分析和聚类分析.采用LEfSe在线工具寻找组与组之间有统计学差异的生物标志物[20].除趋势对应分析(DCA)和冗余分析(RDA)采用CANOCO软件(版本4.5)进行分析[21].
1.4.2 PICRUSt功能预测分析表层沉积物细菌功能和代谢途径预测采用PICRUSt软件进行分析,利用QIIME获得的colsed OTU Table与COG数据库进行比对,获得不同的数据库功能预测信息,具体分析步骤基于在线分析平台(http://picrust.github.io/picrust/)[12].
2 结果与分析 2.1 表层沉积物理化性质丹江口水库沉积物中pH值、有机质、总磷和总钾含量分布规律与采样点位空间分布基本保持一致,即地理位置相近的库心与渠首含量较为接近,低于其他3个点位,宋岗和黑鸡嘴样品理化性质居中,台子山样品中pH值、有机质和总磷含量最高,总钾含量位居第二(表 2).长期监测表明,丹江流域存在农业面源污染,其中总氮明显超标[22-24].丹江口水库表层沉积物总氮含量在0.95~1.73 g/kg之间,平均为1.20 g/kg.沉积物中TN含量空间差异性较大,其中台子山样品最高,含量达到1.76 g/kg,黑鸡嘴样品最低,总氮含量为0.95 g/kg.同时可交换态氮(铵态氮和硝态氮)含量测定结果表明黑鸡嘴样品最高,分别为68.13和63.60 mg/kg.其次为台子山样品,铵态氮和硝态氮含量分别为62.57和46.27 mg/kg.
| 表 2 各采样点沉积物主要理化参数* Tab.2 The main physical and chemical properties of each sediment sample |
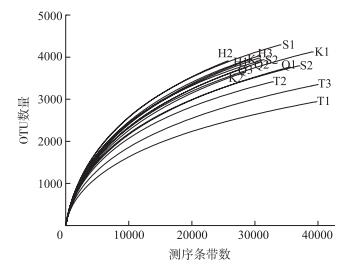
稀释性曲线可以反映样品的取样深度,被用来评价测序量是否足以覆盖所有类群. 5个点位沉积物细菌稀释性曲线如图 1所示,随测序数据量的增加物种丰富度呈现前期增加,在测序条数达到20000条以上时物种数基本趋向平稳.结果表明本次实验测序数据量合理,真实环境中细菌群落结构的置信度较高,能够代表物种的丰富度.
|
图 1 沉积物细菌稀释性曲线 Fig.1 Rarefaction curve of sediments bacterial community |
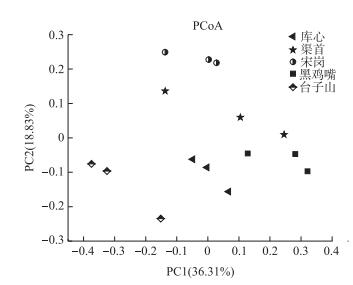
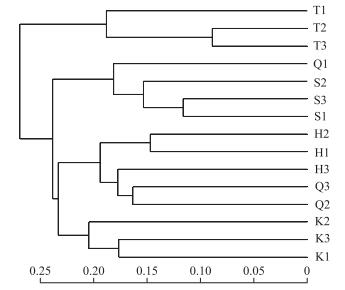
利用Qiime软件对丹江口库区5个点位表层沉积物细菌群落进行PCoA分析(OTUs水平). PCoA分析图中,两轴对排序结果的解释率分别为36.31 %和18.83 %,黑鸡嘴与渠首样品聚集于图右侧,库心样品分布于图中下部,宋岗样品分布于图上部,台子山样品分布于图左下部(图 2).基于unweighted unifrac的UPGMA方法对沉积物细菌群落构成的相似性进行聚类分析,其结果与PCoA分析相似(图 3).在0.204相似性水平上,5个点位表层沉积物细菌组成可以分为4大组,渠首和黑鸡嘴样品较为相近,聚为1组,库心和宋岗样品各自聚为1组.台子山样品在聚类图与其他样品距离最远,表明与其他样品细菌群落差异最大.
|
图 2 沉积物细菌多样性的主坐标分析 Fig.2 PCoA analysis of sediments bacterial community diversity |
|
图 3 不同样品UPGMA聚类图 Fig.3 UPGMA clusters of different bacterial communities |
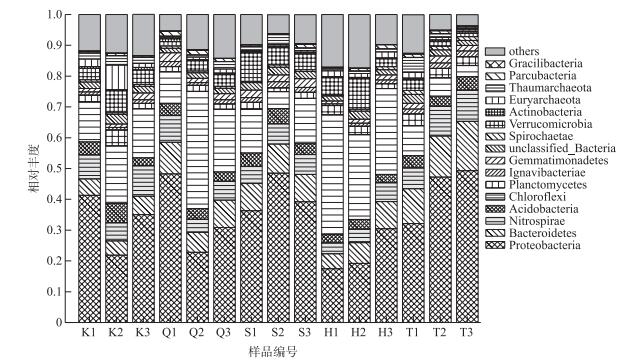
高通量测序结果表明,5个点位样品表层沉积物细菌主要由33个门组成,包括变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、硝化螺旋菌门(Nitrospirae)、酸杆菌门(Acidobacteria)、浮霉菌门(Planctomycetes)和Omnitrophica等(图 4).属于变形菌门、拟杆菌门、硝化螺旋菌门、绿弯菌门、酸杆菌门、浮霉菌门和疣微菌门的序列总和占全部序列的74.38 % ~80.91 %,这些细菌为优势种群(图 4).其中,变形菌门是库心、渠首和宋岗和台子山样品中含量最高的门, 占比分别为32.78 %、33.98 %、41.34 %和42.89 %,同时为黑鸡嘴样品含量第二高的门, 占比为22.37 %.绿弯菌门为黑鸡嘴样品含量最高的门(占比为31.47 %),同时为库心、渠首和宋岗样品含量第二高的门,分别占总群落的15.87 %、22.90 %和11.49 %.
|
图 4 不同样品门水平上物种相对丰度分布 Fig.4 Distribution of relative abundance of different bacterial community structures at phylum levels of different treatments in sediments |
为了进一步确定各样品沉积物细菌种属的丰富度差异,采用在线统计工具LEfSe来寻找宏基因组生物标志物[20].通过LEfSe工具计算出不同样品中的差异菌属,所有菌在门、纲、目、科和属水平的差异信息用饼形图表示[25-26].最内圈表示门水平的差异,依次往外的圈表示纲、目、科和属,不同颜色扇形面积表示在此范围内的菌均有差异(图 5).在门的水平上,库心样品中TM6门和黑鸡嘴样品中2个门(绿弯菌门和FCPU426门)、台子山样品中拟杆菌门、衣原体门、Hydrogenedentes和LCP-89的细菌存在显著差异.在属的水平上,库心样品中变形菌门的Pseudomonas的细菌存在显著差异; 渠首样品酸杆菌门的Geothrix,拟杆菌门的Lacibacter和Haliscomenobacter,变形菌门的Denitratisoma、Desulfatirhabdium、Desulfobacula、Phaselicystis和Oleiphilus等13个属的细菌存在显著差异; 宋岗样品中拟杆菌门的Fluviicola和Phaeodactylibacter,芽单胞菌门的Gemmatirosa,变形菌门的Phenylobacterium、Devosia、Bdellovibrio、Anaeromyxobacter、Haliangium、Polyangium、Sorangium和Pseudohongiella等27个属的细菌存在显著差异; 黑鸡嘴样品中绿弯菌门的Bellilinea,变形菌门的Acidovorax和Thioploca等6个属的细菌存在显著差异; 台子山样品中酸杆菌门GOUTB8、Rhodococcus,拟杆菌门的Actibacter、Dinghuibacter和Ferruginibacter,厚壁菌门的Syntrophomonas,变形菌门的Thiobacillus、Methylotenera、Candidatus Nitrotoga、Nitrosomonas、Syntrophus和Sulfuricurvum等30个属的细菌存在显著差异.
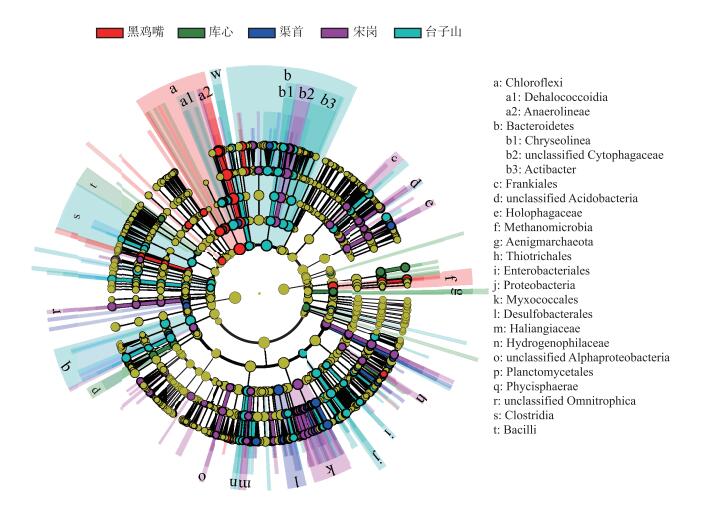
|
图 5 不同样品差异细菌分布 Fig.5 Taxon with statistical differences among different bacterial communities |
利用Canoco 4.5软件对表层沉积物细菌门分类水平群落丰度进行除趋势对应分析(Detrended Correspondence Analysis,DCA),结果显示4个轴中的特征值最大为1.186,小于3.0,选择线性模型中冗余分析(RDA)用于后续分析. RDA结果如图 6所示,所分析的环境因子中总氮、总磷、总钾和pH值等理化指标均与第一排序轴呈负相关,其中pH值、总磷和有机质含量的相关性系数分别为-0.511、-0.534和-0.525,达到显著负相关(P < 0.05). pH值与第二排序轴呈正相关,相关系数为0.0092,其他理化指标与第二排序轴均呈负相关,其中铵态氮相关性系数为-0.606,达到显著差异水平(P < 0.05).
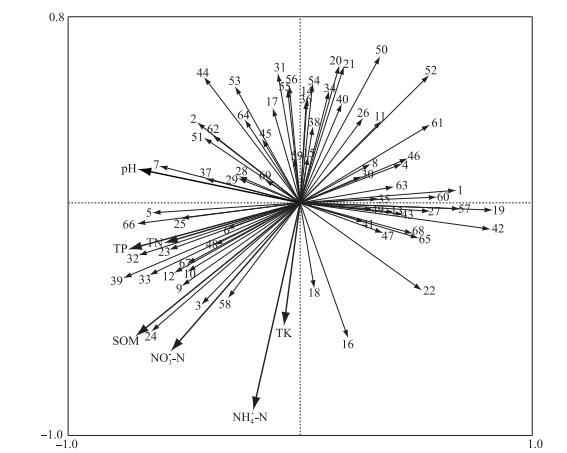
|
图 6 沉积物细菌门分类群落(数字箭头)与环境因子的RDA分析 Fig.6 RDA analysis of bacterial community species (phylum) in sediments and environmental factors |
为进一步明确表层沉积物细菌群落与环境因子的关系,将不同样品细菌属分类单元与环境因子进行Spearman相关分析(rho),结果表明(图 7),变形菌门的Arenimonas(rho=0.535,P < 0.05),酸杆菌门的Bryobacter(rho=0.655,P < 0.01),拟杆菌门的Chryseolinea(rho=0.622,P < 0.05)与pH值均呈显著正相关; 变形菌门的Candidatus Nitrotoga(rho=0.611,P < 0.05)、Desulfatirhabdium (rho=-0.698,P < 0.01)、Sva0081 sediment group (rho=-0.556,P < 0.05)、Sulfuricurvum(rho=0.530,P < 0.05)和Syntrophus(rho=0.796,P < 0.01)与有机质含量均呈显著相关; 变形菌门的Candidatus Nitrotoga(rho=-0.644,P < 0.05)与总钾含量呈显著负相关; 变形菌门的Candidatus Nitrotoga(rho=0.666,P < 0.01)和Syntrophus(rho=0.546,P < 0.05)与总磷含量均呈显著正相关; 变形菌门的Sulfuricurvum(rho=0.661,P < 0.01)、拟杆菌门的Dinghuibacter(rho=0.644,P < 0.01)、疣微菌门的Opitutus (rho=0.672,P < 0.01)、绿弯菌门的Anaerolinea(rho=-0.600,P < 0.05)和硝化螺旋菌门的Nitrospira(rho=0.600,P < 0.05)与总氮含量均呈显著相关; 酸杆菌门的Thermoanaerobaculum(rho=-0.742,P < 0.01),拟杆菌门的Dinghuibacter(rho=-0.567,P < 0.05)、Terrimonas(rho=-0.551,P < 0.05),变形菌门的Anaeromyxobacter(rho=-0.687,P < 0.01)、Desulfatirhabdium(rho=-0.884,P < 0.01)、Haliangium(rho=-0.664,P < 0.01)和Sva0081 sediment group(rho=-0.622,P < 0.05)以及疣微菌门的Opitutus(rho=-0.650,P < 0.01)与硝态氮含量均呈显著相关; 变形菌门的Candidatus Nitrotoga (rho=0.644,P < 0.01)、Desulfatirhabdium (rho=-0.524,P < 0.05)、Sva0081 sediment group (rho=-0.611,P < 0.05)、BD1-7 clade(rho=-0.644,P < 0.01)、Geobacter(rho=0.525,P < 0.05)和Rubrivivax(rho=-0.530,P < 0.05)与铵态氮含量均呈显著相关.
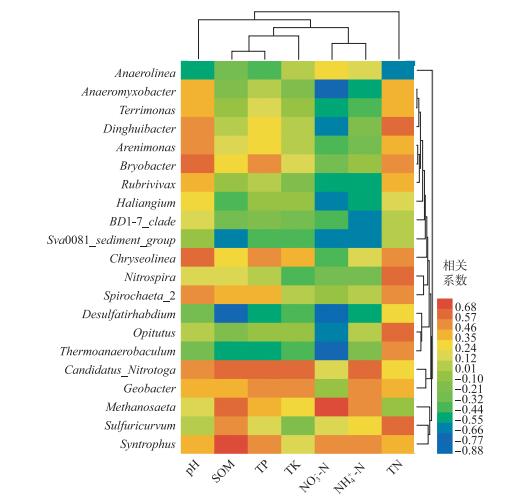
|
图 7 沉积物细菌属分类群落与环境因子的Spearman相关系数热图 Fig.7 Heatmap of Spearman's coorelation coefficients between bacterial community species (genus) in sediments and environmental factors |
为了获得不同生态点位表层沉积物细菌的功能,本研究采用PICRUSt软件进行菌群预测分析.基于Clusters of Orthologs Groups (COG)数据库预测的结果表明,共计25个基因功能家族中核结构功能基因家族(Nuclear structure)缺失,其他24个受到不同生态点位的影响(图 8).其中能量产生和转换(Energy production and conversion)、信号转导机制(Signal transduction mechanisms)、氨基酸运输和代谢(Amino acid transport and metabolism)、细胞壁/细胞膜/膜结构的生物合成(Cell wall/membrane/envelope biogenesis)、翻译、核糖体结构和生物合成(Translation,ribosomal structure and biogenesis)、复制、重建和修复(Replication,recombination and repair)、无机离子转运与代谢(Inorganic ion transport and metabolism)以及碳水化合物的运输和代谢(Carbohydrate transport and metabolism)八大类功能基因家族为主要功能基因家族,占比之和为52.98 % ~53.54 %.未能很好注释的基本预测功能家族(General function prediction only)和未知功能基因家族(Function unknown)占比分别为7.85 % ~8.40 %和8.94 % ~9.05 %.不同生态点位沉积物细菌群落功能预测比较表明,除RNA加工和修饰(RNA processing and modification)、染色质结构与动力学(Chromatin structure and dynamics)、细胞外结构(Extracellular structures)和细胞骨架(Cytoskeleton)之外,基因功能家族预测基因拷贝数表现为台子山>宋岗>库心>渠首>黑鸡嘴(图 8).其中,台子山样品中除RNA加工和修饰(RNA processing and modification)、碳水化合物的运输和代谢(Carbohydrate transport and metabolism)、脂质运输和代谢(Lipid transport and metabolism)、次生代谢产物的合成、转运和代谢(Secondary metabolites biosynthesis,transport and catabolism)和细胞外结构(Extracellular structures)之外,预测基因拷贝数均显著高于渠首和黑鸡嘴样品. RNA加工和修饰(RNA processing and modification)功能家族预测基因拷贝数为库心>宋岗>台子山>渠首>黑鸡嘴,染色质结构与动力学功能家族预测基因拷贝数为台子山>库心>宋岗>渠首>黑鸡嘴,细胞外结构功能家族预测基因拷贝数为台子山>宋岗>渠首>库心>黑鸡嘴,细胞骨架功能家族预测基因拷贝数为宋岗>台子山>库心>渠首>黑鸡嘴.
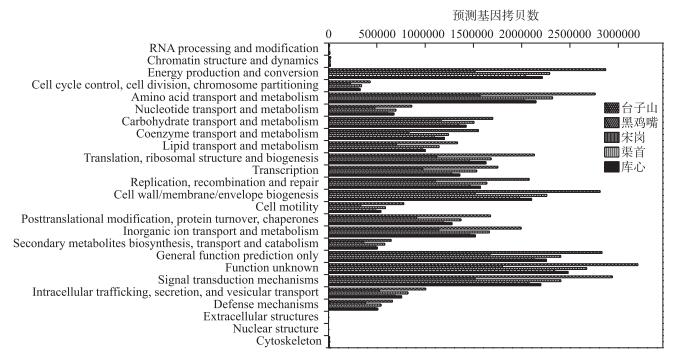
|
图 8 丹江口库区沉积物细菌PICRUSt功能预测 Fig.8 PICRUSt metabolic predictions of the bacterial communities in Danjiangkou Reservoir sediment |
湖泊微生物是水生态系统的重要组成部分,在管理和维护湖泊生态环境具有深远的意义[2, 4].本课题组曾采用高通量测序对丹江口库区浮游细菌群落进行研究[25-26],但关于丹江口库区表层沉积物细菌群落研究鲜见报道.为了解丹江口库区表层沉积物细菌群落组成,本实验采用Illumina公司MiSeq测序平台对丹江库区表层沉积物细菌群落组成进行研究,发现其主要由变形菌门、绿弯菌门、拟杆菌门、疣微菌门和硝化螺旋菌门等33个门,Fluviicola、Phaeodactylibacter、Phenylobacterium、Devosia和Bdellovibrio等318个属的细菌组成,表现出群落组成的丰富性.丹江口沉积物细菌群落组成与之前研究的洞庭湖[27]、龙景湖[28]、鄱阳湖[10]、太湖[29]和滇池[30]等淡水湖泊以及长江[31]、北运河[3]等河流及其海洋[2, 6-7, 13, 32]等水体的沉积物细菌组成类群相似,这些沉积物细菌大都隶属于变形菌门、绿弯菌门、放线菌门和厚壁菌门等,研究表明这些沉积物细菌在有机污染物降解、碳循环和水生生态系统生物地球化学循环过程中起到重要作用[28, 33-34].
3.2 丹江口库区表层沉积物细菌群落与环境因子的关系长期监测表明,丹江口库区上游河流存在农业面源污染,其中总氮明显超标[22-24].本研究采用美国环境保护署(EPA)制定的沉积物总氮、总磷污染评价标准对丹江口库区表层沉积物污染状况进行评价[35].丹江口水库表层沉积物总氮含量在0.95~1.73 g/kg之间,平均为1.20 g/kg,参照美国EPA中沉积物总氮污染评价标准,除黑鸡嘴样品外,其他4个样品均为中度污染.该结果与赵丽等[23]关于丹江口库区表层沉积物2015年测定结果基本一致.根据美国EPA制定的沉积物总磷污染的评价标准,宋岗、黑鸡嘴和台子山样品总磷含量介于0.52~0.60 g/kg之间,为中度污染,库心和渠首样品总磷含量低于0.45 g/kg,为轻度污染.
研究表明,沉积物中的环境因子,包括深度、有机质、总氮、总磷含量和pH值等均能不同程度地影响沉积物细菌的群落组成[2, 6-7]. Liu等[7]研究表明,pH值是影响钱塘江沉积物细菌群落的最主要因素. Song等[11]对东平湖沉积物细菌群落分析发现,总磷是影响细菌群落的主要因素.为了解环境因子对丹江口库区表层沉积物细菌群落的影响,本实验采用冗余分析(RDA)对沉积物细菌群落和理化指标进行分析,结果表明pH值、总磷、有机质和铵态氮能显著影响沉积物细菌群落组成,该结果与Liu等[7]、Song等[11]的研究结果类似.为进一步明确表层沉积物细菌群落与环境因子的关系,将不同样品细菌属分类单元与环境因子进行Spearman相关分析.结果发现与环境因子显著相关的细菌中变形菌门为主要组成(占47.62 %),其中Desulfatirhabdium、Sva0081 sediment group与有机质、铵态氮和硝态氮含量均呈显著负相关,Anaeromyxobacter、Haliangium与硝态氮含量均呈显著负相关,Rubrivivax与铵态氮含量呈显著负相关,Candidatus Nitrotoga、Geobacter与铵态氮含量均呈显著正相关.研究表明Desulfatirhabdium是一类硫酸盐还原细菌,在沉积物硫酸盐还原中起到作用[36],Candidatus Nitrotoga是亚硝酸氧化细菌,在亚硝酸盐降解过程中起到重要作用[37],Geobacter是异化Fe(Ⅲ)还原微生物,能广泛分布在不同沉积物中,能以Fe(Ⅲ)作为电子受体获能生长[38],这些沉积物细菌在生物地球化学循环中起到重要作用.另外,变形菌门的Sulfuricurvum、疣微菌门的Opitutus、拟杆菌门的Dinghuibacter与总氮含量均呈显著正相关,变形菌门的Syntrophus与总磷含量呈显著正相关.
3.3 PICRUSt功能预测分析之前关于沉积物细菌群落的高通量测序分析主要关注于微生物群落结构(α和β多样性),研究沉积物微生物群落组成、分布特征及其影响因素等[6-10],对其功能研究开展较少.基于高通量测序的PICRUSt功能预测分析,在沉积物微生物研究中已经开始应用. Cleary等[13]对印度尼西亚的Spermonde群岛周边海水、沉积物等细菌群落采用高通量测序进行研究,并通过PICRUSt预测其功能,发现不同样品细菌群落功能上具有差异. Verma等[14]对深海沉积物细菌群落进行研究,PICRUSt预测分析表明,深海沉积物细菌功能主要涉及代谢、环境信息处理和细胞进程等重要功能.为了获得丹江口库区不同生态点位表层沉积物细菌的功能,本研究也采用PICRUSt软件进行菌群预测分析.结果表明,丹江口库区表层沉积物细菌主要涉及能量产生和转换、信号转导机制、氨基酸运输和代谢等24个基因功能家族,表现出功能上的丰富性.其中通过PCoA和UPGMA分析细菌群落与其他样品差异较大的台子山样品,PICRUSt预测也与其他样品有较大差异,22个基因功能家族预测基因拷贝数高于其他样品.与台子山样品类似,群落上与其他样品有差异的宋岗样品,预测基因拷贝数整体趋势上高于库心、渠首和黑鸡嘴样品.基因功能家族预测基因拷贝数表现为台子山>宋岗>库心>渠首>黑鸡嘴. PICRUSt功能预测分析在丹江口沉积物微生物研究中应用,并与高通量测序技术结合分析,将为探明丹江口沉积物微生物群落组成和功能提供重要帮助.
4 结论针对丹江口库区沉积物细菌群落和功能研究鲜见报道的现状,采用高通量测序技术对表层沉积物细菌群落组成进行分析,并采用PICRUSt软件预测其功能.结果表明:
1) 丹江库区表层沉积物细菌具有丰富的群落组成,主要由变形菌门、绿弯菌门、拟杆菌门、疣微菌门、硝化螺旋菌门和酸杆菌门等33个门,Fluviicola、Phaeodactylibacter、Phenylobacterium、Devosia、Bdellovibrio和Anaeromyxobacter等318个属的细菌组成.
2) 沉积物理化指标中pH值、总磷、有机质和铵态氮含量能显著影响沉积物细菌群落组成.细菌属分类单元与环境因子Spearman相关分析表明,变形菌门中11个属的细菌与环境因子显著相关,占属分类单元上与环境因子显著相关细菌的47.62 %.
3) 丹江口库区表层沉积物细菌功能上具有丰富性,主要涉及能量产生和转换、信号转导机制、氨基酸运输和代谢等24个基因功能家族.基因功能家族预测基因拷贝数表现为台子山>宋岗>库心>渠首>黑鸡嘴.
| [1] |
Haller L, Tonolla M, Zopfi J et al. Composition of bacterial and archaeal communities in freshwater sediments with different contamination levels (Lake Geneva, Switzerland). Water Research, 2011, 45(3): 1213-1228. DOI:10.1016/j.watres.2010.11.018 |
| [2] |
Acosta-González A, Marqués S. Bacterial diversity in oil-polluted marine coastal sediments. Current Opinion in Biotechnology, 2016, 38: 24-32. DOI:10.1016/j.copbio.2015.12.010 |
| [3] |
Bao LL, Chen YJ, Wang XY. Diversity and abundance of ammonia-oxidizing prokaryotes in surface sediments in Beiyun River. China Environmental Science, 2015, 35(1): 179-189. [鲍林林, 陈永娟, 王晓燕. 北运河沉积物中氨氧化微生物的群落特征. 中国环境科学, 2015, 35(1): 179-189.] |
| [4] |
Ren LJ, He D, Xing P et al. Bacterial diversity and ecological function in lake water bodies. Biodiversity Science, 2013, 21(4): 421-432. [任丽娟, 何聃, 邢鹏等. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性, 2013, 21(4): 421-432.] |
| [5] |
van Dijk EL, Auger H, Jaszczyszyn Y et al. Ten years of next-generation sequencing technology. Trends in Genetics, 2014, 30(9): 418-426. DOI:10.1016/j.tig.2014.07.001 |
| [6] |
Mahmoudi N, Robeson MS, Castro HF et al. Microbial community composition and diversity in Caspian Sea sediments. FEMS Microbiology Ecology, 2015, 91(1): 1-11. |
| [7] |
Liu S, Ren HX, Shen LD et al. pH levels drive bacterial community structure in sediments of the Qiantang River as determined by 454 pyrosequencing. Frontiers in Microbiology, 2015, 6: 285. |
| [8] |
Lu XM, Lu PZ. Characterization of bacterial communities in sediments receiving various wastewater effluents with high-throughput sequencing analysis. Microbial Ecology, 2014, 67(3): 612-623. DOI:10.1007/s00248-014-0370-0 |
| [9] |
Huang Y, Fang X, Chu WK et al. Bacterial diversity and community structure in sediments of Xixi wetland, Hangzhou. Oceanologia et Limnologia Sinica, 2015, 46(5): 1202-1209. [黄媛, 方序, 褚文珂等. 杭州西溪湿地沉积物细菌的群落结构和多样性. 海洋与湖沼, 2015, 46(5): 1202-1209.] |
| [10] |
Wang P, Chen B, Zhang H. High throughput sequencing analysis of bacterial communities in soils of a typical Poyang Lake wetland. Acta Ecologica Sinica, 2017, 37(5): 1650-1658. [王鹏, 陈波, 张华. 基于高通量测序的鄱阳湖典型湿地土壤细菌群落特征分析. 生态学报, 2017, 37(5): 1650-1658.] |
| [11] |
Song H, Li Z, Du B et al. Bacterial communities in sediments of the shallow Lake Dongping in China. Journal of Applied Microbiology, 2012, 112(1): 79-89.
|
| [12] |
Langille MGI, Zaneveld J, Caporaso JG et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [13] |
Cleary DFR, de Voogd NJ, Polónia ARM et al. Composition and predictive functional analysis of bacterial communities in seawater, sediment and sponges in the Spermonde Archipelago, Indonesia. Microbial Ecology, 2015, 70(4): 889-903. DOI:10.1007/s00248-015-0632-5 |
| [14] |
Verma P, Raghavan RV, Jeon CO et al. Complex bacterial communities in the deep-sea sediments of the Bay of Bengal and volcanic Barren Island in the Andaman Sea. Marine Genomics, 2017, 31: 33-41. DOI:10.1016/j.margen.2016.08.003 |
| [15] |
Bao SD. Methods of soil agricultural chemical analysis. Beijing: China Agricultural Science and Technology Press, 2000. [鲍士旦. 土壤农业化学分析方法. 北京: 中国农业出版社, 2000.]
|
| [16] |
Chen ZJ, Zheng Y, Ding CY et al. Integrated metagenomics and molecular ecological network analysis of bacterial community composition during the phytoremediation of cadmium-contaminated soils by bioenergy crops. Ecotoxicology and Environmental Safety, 2017, 145: 111-118. DOI:10.1016/j.ecoenv.2017.07.019 |
| [17] |
Zheng Y, Li YY, Ding CY et al. Effects of bioenergy cropping on rhizosphere bacteria networks structure in Cd contaminated soil. Acta Scientiae Circumstantiae, 2016, 36(7): 2605-2612. [郑远, 李玉英, 丁传雨等. 能源植物修复镉污染土壤对根际细菌网络结构的影响. 环境科学学报, 2016, 36(7): 2605-2612.] |
| [18] |
Caporaso JG, Kuczynski J, Stombaugh J et al. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [19] |
Schloss PD, Westcott SL, Ryabin T et al. Introducing mothur:Open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [20] |
Segata N, Izard J, Waldron L et al. Metagenomic biomarker discovery and explanation. Genome Biology, 2011, 12(6): 1-18. |
| [21] |
Ter Braak CJ, Smilauer P. CANOCO reference manual and CanoDraw for Windows user's guide:software for canonical community ordination (version 4.5). Ithaca: Microcomputer Power, 2002.
|
| [22] |
Xu GC, Li ZB, Li P et al. Spatial distribution of soil total nitrogen in a typical watershed of the middle danjiang river. Acta Geographica Sinica, 2012, 67(11): 1547-1555. [徐国策, 李占斌, 李鹏等. 丹江中游典型小流域土壤总氮的空间分布. 地理学报, 2012, 67(11): 1547-1555. DOI:10.11821/xb201211011] |
| [23] |
Zhao L, Jiang X, Wang WW et al. Occurrence characteristics and bio-availability of nitrogen fractions in sediments of Danjiangkou Reservoir. Resources and Environment in the Yangtze Basin, 2016, 25(4): 630-637. [赵丽, 姜霞, 王雯雯等. 丹江口水库表层沉积物不同形态氮的赋存特征及其生物有效性. 长江流域资源与环境, 2016, 25(4): 630-637.] |
| [24] |
Zhu YY, Tian JJ, Li HL et al. Water qaulity assessment and pollution profile identification of Danjiangkou Reservoir, China. Journal of Agro-Environment Science, 2016, 35(1): 139-147. [朱媛媛, 田进军, 李红亮等. 丹江口水库水质评价及水污染特征. 农业环境科学学报, 2016, 35(1): 139-147. DOI:10.11654/jaes.2016.01.019] |
| [25] |
Chen ZJ, Chen HY, Li YY et al. Community structure and influencing factors of bacterioplankton in the main canal of the Mid-line Project of South-to-North Water Division in sections of Henan Province. China Environmental Science, 2017, 37(4): 1505-1513. [陈兆进, 陈海燕, 李玉英等. 南水北调中线干渠(河南段)浮游细菌群落组成及影响因素. 中国环境科学, 2017, 37(4): 1505-1513.] |
| [26] |
Chen ZJ, Ding CY, Zhu JY et al. Community structure and influencing factors of bacterioplankton during low water periods in Danjiangkou Reservoir. China Environmental Science, 2017, 37(1): 336-344. [陈兆进, 丁传雨, 朱静亚等. 丹江口水库枯水期浮游细菌群落组成及影响因素研究. 中国环境科学, 2017, 37(1): 336-344.] |
| [27] |
Wu H, Zeng G, Liang J et al. Changes of soil microbial biomass and bacterial community structure in Dongting Lake:Impacts of 50, 000 dams of Yangtze River. Ecological Engineering, 2013, 57: 72-78. DOI:10.1016/j.ecoleng.2013.04.038 |
| [28] |
Niu FX, Ji FY, Zhao G et al. Vertical distribution of bacterial communities in sediments of Longjing Lake. China Environmental Science, 2017, 37(6): 2322-2331. [牛凤霞, 吉芳英, 赵艮等. 龙景湖沉积物的细菌群落垂向分布特征. 中国环境科学, 2017, 37(6): 2322-2331.] |
| [29] |
Wu HN, Li Y, Zhang J et al. Sediment bacterial communities in a eutrophic lake influenced by multiple inflow-rivers. Environmental Science and Pollution Research, 2017, 24(24): 19795-19806.
|
| [30] |
Dai Y, Yang YY, Wu Z et al. Spatiotemporal variation of planktonic and sediment bacterial assemblages in two plateau freshwater lakes at different trophic status. Applied Microbiology and Biotechnology, 2016, 100(9): 4161-4175. DOI:10.1007/s00253-015-7253-2 |
| [31] |
Huang W, Jiang X. Profiling of sediment microbial community in Dongting Lake before and after Impoundment of the Three Gorges Dam. International Journal of Environmental Research and Public Health, 2016, 13(6): 617. DOI:10.3390/ijerph13060617 |
| [32] |
Liu MH, Wang JX, Yu KC et al. Community structure and geographical distribution of bacterial on surface layer sediments in the east China sea. Oceanologia et Limnologia Sinica, 2015, 46(5): 1119-1131. [刘明华, 王健鑫, 俞凯成等. 东海陆架表层沉积物细菌群落结构及地理分布研究. 海洋与湖沼, 2015, 46(5): 1119-1131.] |
| [33] |
Cheng W, Zhang JX, Wang Z et al. Bacterial communities in sediments of a drinking water reservoir. Annals of Microbiology, 2013, 64(2): 875-878. |
| [34] |
Inagaki F, Nunoura T, Nakagawa S et al. Biogeographical distribution and diversity of microbes in methane hydrate-bearing deep marine sediments on the Pacific Ocean Margin. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(8): 2815-2820. DOI:10.1073/pnas.0511033103 |
| [35] |
MacDonald DD, Ingersoll CG. A guidance manual to support the assessment of contaminated sediments in freshwater ecosystems. US EPA, 2002.
|
| [36] |
Balk M, Altınbaș, Rijpstra WIC et al. Desulfatirhabdium butyrativorans gen. nov., sp. nov., a butyrate-oxidizing, sulfate-reducing bacterium isolated from an anaerobic bioreactor. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(1): 110-115. DOI:10.1099/ijs.0.65396-0 |
| [37] |
Chen Y, Wu L, Boden R et al. Life without light:microbial diversity and evidence of sulfur-and ammonium-based chemolithotrophy in Movile Cave. ISME J, 2009, 3(9): 1093-1104. DOI:10.1038/ismej.2009.57 |
| [38] |
Coates JD, Phillips EJ, Lonergan DJ et al. Isolation of Geobacter species from diverse sedimentary environments. Applied and Environmental Microbiology, 1996, 62(5): 1531-1536. |
 2018, Vol. 30
2018, Vol. 30 


