(2: 中国科学院水生生物研究所, 武汉 430072)
(2: Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, P. R. China)
浮游动物是水生态系统的重要组成部分,在物质循环、能量流动和信息传递中发挥着重要作用[1].当浮游动物遭遇环境恶化、食物缺乏以及种群密度急剧增大等胁迫时,会产生一种体表被膜的休眠胚胎,即休眠卵[2].不同类群休眠卵形态特征明显不一,枝角类休眠卵卵体较大,长径可达400~600 μm,外被一卵鞍,卵鞍坚硬且不易碎.卵鞍内储1~2枚圆形或椭圆形休眠卵[3],卵与卵鞍呈平行或垂直,卵鞍常呈圆形、半圆形、椭圆形、方形、贝壳型等不同形态,有些卵鞍面有多边形纹饰.轮虫休眠卵较小,长径约为60~100 μm,在光镜下呈卵圆形、球形,外被一层深色硬壳,表面具不规则纹饰如弯曲小脊或蜂窝状凹陷或泡状物[4],一侧常有气室;淡水桡足类休眠卵多呈球状,而海洋桡足类休眠卵形态和结构则较为多样化,外壳较厚且表面光滑或具刺、鞘等不同以帮助其抵抗不利环境,直径约为100 μm[5].休眠卵的形状和外部结构的差异,在一定程度上可以应用于较高分类阶层甚至物种级别的物种鉴定.休眠卵的形成和萌发是浮游动物重要的生活史策略[6].表层沉积物中休眠卵在水体全同温转换时通过水体搅动,被带到水层区萌发以补充到自然种群中,从而调节浮游动物群落结构及种群的动态变化,以维持水体中的浮游动物多样性,保持浮游动物自然种群稳定[7].通过了解沉积物中休眠卵的种类多样性,可以反映水体中浮游动物种群真实的物种多样性及环境变化对水体浮游动物群落结构的影响[8].
目前鉴定浮游动物休眠卵主要有传统形态学鉴定[9]和休眠卵萌发鉴定[10]两种方法.由于浮游动物休眠卵分类学资料的匮乏,以及休眠卵体表形态的多样性,使得通过形态学鉴定浮游动物休眠卵存在一定难度而且准确率低,大多只能鉴定至属或科.休眠卵的生理结构非常复杂,孵化成功与否取决于滞育终止的程度、卵的能量含量、非存活胚胎的数量以及环境因素,需要探索培养条件,萌发周期较长,成功率低,鉴定结果也因分类学知识的储备差别巨大.因此,通过形态学鉴定浮游动物休眠卵存在一定难度而且准确率低.随着DNA条形码发展为一种可靠的物种快速鉴定工具,许多学者将其应用至浮游动物种类鉴定中.
DNA条形码技术是使用一段标准化的DNA片段进行物种鉴定的方式,形似商品条形码,建立DNA序列信息与物种信息逐一对应的关系[11].针对不同类群的生物,通常采用不同基因作为条形码序列,例如生命条形码联盟(CBOL)植物工作组推荐叶绿体基因组的rbcL和matK片段作为植物鉴定的DNA条形码[12],真菌工作组推荐核糖体DNA内转录间隔区基因(ITS)为真菌首选DNA条形码[13],而动物物种的鉴定则采用线粒体细胞色素c氧化酶第一亚基编码基因(cytochrome c oxidase I gene, COⅠ)[14]. COⅠ基因不仅广泛应用于鸟类、蝴蝶、鱼类等物种鉴定中,而且由于使用DNA条形码鉴定时不受生物生活史阶段的限制,应用至微型浮游动物群落物种组成分析中发挥了巨大的作用,如王仁诚等对胶州湾夏季浮游动物物种组成的研究中,使用COⅠ基因成功鉴定出日本蟳、太平洋牡蛎等无脊椎动物的浮游幼体[15];张翔等使用COⅠ基因鉴定了太湖流域部分底栖无脊椎动物种类[16];Machida等使用DNA条形码技术,成功鉴定了密克罗尼西亚群岛的多种浮游生物[17];Bucklin等利用该方法,研究了北冰洋浮游桡足类种类组成[18];Baek等使用线粒体细胞色素COⅠ基因对133种桡足类个体成功进行了鉴定[19].本研究采用DNA条形码技术基于COⅠ基因序列对柳叶湖表层沉积物中浮游动物休眠卵进行了分子鉴定,同时对其形态信息进行采集,对建立浮游动物休眠卵图鉴,快速鉴定休眠卵种类有积极意义.
1 材料与方法 1.1 研究区域柳叶湖(29°4′~29°8′N,111°40′~111°50′E)位于湖南省常德市境内,属于洞庭湖流域.水域面积21.8 km2,容积为4.9×108 m3,主要接纳周边山地、平原区地表径流和沟港来水,与多条河汇合后入沅江,再汇入洞庭湖,是一个典型的城市浅水湖泊.
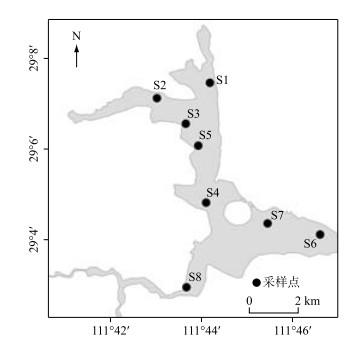
1.2 样品采集与处理根据湖泊形状特征,共设8个采样点(S1~S8,图 1),2018年6月使用采泥器(12 cm × 24 cm)采集柳叶湖表层5 cm沉积物,每个样点采集2个样品,共采集16个样品.样品采集后置于4℃冰箱保存,再带回实验室后使用蔗糖浮选法[20]对休眠卵进行分离.具体步骤如下:取20 cm3沉积物样品先后通过830 μm、64 μm筛网过滤初筛.完成初筛后,沥净筛网中的水,用饱和蔗糖溶液(W蔗糖 :W水=1 :1)将滤渣转移到50 mL离心管中,上下颠倒使其混匀,在3000 r/min下离心20 min.离心后将上清液再次转移至50 mL离心管,重复上述步骤至上清液无杂质,最后将上清液转移至30 μm筛网,用超纯水冲洗后转移至15 mL离心管中,4℃保存.
|
图 1 洞庭湖流域常德柳叶湖表层沉积物采样点 Fig.1 Sampling locations in Lake Liuye (Changde), Lake Dongting Basin, China |
将保存休眠卵的离心管轻轻上下颠倒,充分混匀后用吸管转移至细胞培养板中,置于体视显微镜下观察,选择卵体完整的休眠卵使用单通道移液器吸取到载玻片上,在光学显微镜(OLYMPUS,BX51)下对共计191个休眠卵进行图像采集,测量长短径,观察记录形态特征,并对图片进行统一编号,随后将单卵转移到PCR管中,待提取DNA.
1.4 DNA提取、PCR扩增及测序本研究采用“HotSHOT”法[21]对191个休眠卵进行单卵DNA提取.首先向含有浮游动物休眠卵的PCR管中加入10 μL裂解液,在体式显微镜下用显微镊将卵体破碎,放置于干式恒温仪95℃裂解30 min后取出,然后置于-20℃冷却5 min,最后加入10 μL中和液并吸打混匀,即完成DNA提取.
使用引物ZplankF1_t1(TGTAAAACGACGGCCAGTTCTASWAATCATAARGATATT)和ZplankR1_t1(CAGGAAACAGCTATGACTTCAGGRTGRCCRAARAATCA)对总基因组DNA的COⅠ基因进行PCR扩增[16],扩增反应体系为25 μL,包含2×Gflex PCR Buffer (Mg2+, dNTP plus) 12.5 μL,引物各0.3 μmol/L,模板为5 μL,Tks Gflex DNA Polymerase 0.5 μL,ddH2O补足至25 μL.扩增程序为95℃预变性1 min,94℃变性40 s,45℃退火40 s,72℃延伸45 s,5个循环;94℃变性40 s,51℃退火40 s,35个循环,最后72℃ 5 min充分延伸. PCR完成后,扩增产物经琼脂糖凝胶电泳检测后,选择目标条带单一且明亮的PCR扩增产物送交上海美吉生物医药科技有限公司纯化并进行双向测序,测序引物为M13F(TGTAAAACGACGGCCAGT)及M13R(CAGGAAACAGCTATGAC).
1.5 序列分析与比对将测序所得原始序列使用Chromas 2.6.4软件对峰图进行检查,并对碱基进行逐一校对.由于样品采用双向测序,使用BioEdit 7.0.5软件进行序列拼接,以确保所用序列结果的准确性.对所获得有效的序列在NCBI(National Center for Biotechnology Information)进行序列比对,同时下载相关的物种序列用于系统发育树构建.序列比对时,参考Machida等[17]和Bucklin等[23]的研究分类标准,若序列相似度大于96 %,则可鉴定至物种;若序列相似性介于83 % ~96 %间,依据相似度的高低鉴定至属或科;若序列相似性小于83 %,则归类至“门”或其他更高阶元.
1.6 系统发育分析分别对桡足类、枝角类和轮虫3个类群物种序列开展系统发育分析.使用MEGA X(10.0.5)分析所获得序列长度、碱基组成,采用Kimura-2-Parameter双参数模型(K2P)计算遗传距离,使用邻接(Neighbor-Joining)法构建系统进化树,经1000次重复抽样(bootstrap)检验其置信度.根据序列比对结果,选取每条序列相似度最高的序列作为参考序列,同时下载与比对结果相似度相近物种的序列及同源序列,其中桡足类47条、枝角类54条,轮虫38条序列分别构建进化树,进一步确定物种.
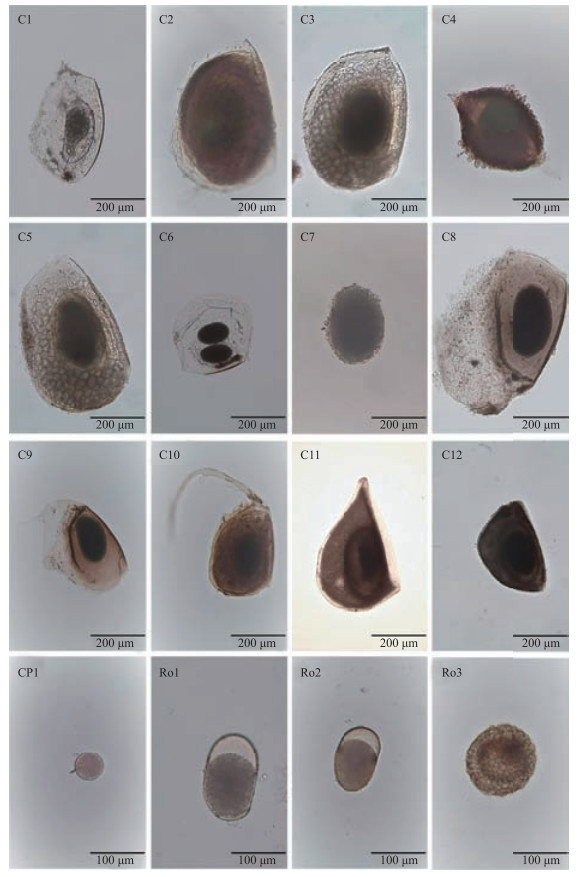
2 结果 2.1 柳叶湖浮游动物休眠卵序列特征及比对结果本研究共对191个休眠卵进行了分子鉴定,对测序结果进行拼接后成功获取101条COⅠ基因部分片段序列,单次成功率为52.88 %.扩增的COⅠ基因部分序列长度约为649 bp,序列无插入和缺失.在所有序列比对结果中核苷酸T、C、A、G的平均含量分别为35.22 %、17.78 %、27.91 %、18.98 %,且A+T含量(63.13 %)远高于C+G含量(36.76 %),碱基均出现明显的偏倚性,符合线粒体碱基组成特点.常德柳叶湖浮游动物休眠卵获得的DNA序列比对结果见表 1.本研究在191个休眠卵中所获得101条序列中,成功鉴定出17种休眠卵,其中11种鉴定至种,5种鉴定至属,1种鉴定至科;同时对成功鉴定的休眠卵进行分类并逐个编号,其中桡足类为CP1、枝角类为C1~C13、轮虫为Ro1~Ro3,同时记录该种休眠卵出现的样点及检出的个体数(表 1、图 2).
| 表 1 条形码序列NCBI比对结果与样品鉴定结果的比较1) Tab. 1 Comparison between barcode sequence NCBI alignment and sample identification |
|
图 2 休眠卵形态学信息 Fig.2 Morphological information of resting eggs |
样品C1、C2、C4、C6、C12、C13、CP1、Ro1、Ro2通过NCBI数据库比对可鉴定至种(表 1);样品C3、C5、C9、Ro3可鉴定至属(表 1);样品C7和C8鉴定至科,其中样品C8可能为高壳溞(Kurzia sp.,序列相似度为82 %);样品C10序列比对结果有两种,无刺大尾溞(Leydigia acanthocercoides)和Camptocercus dadayi,相似度分别为85.9 %和85.8 %,同属盘肠溞科;样品C11比对结果为低额溞(Simocephalus sp.),可能为黑龙江低额溞(S. heilongjiangensis,相似度88.5 %).
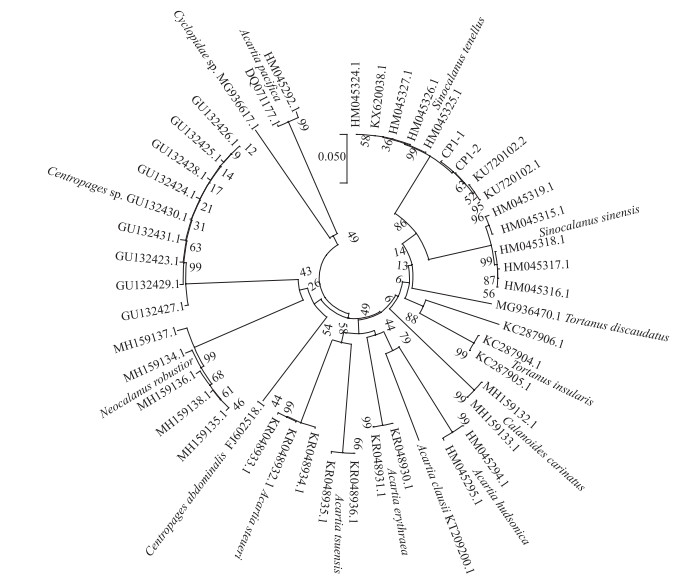
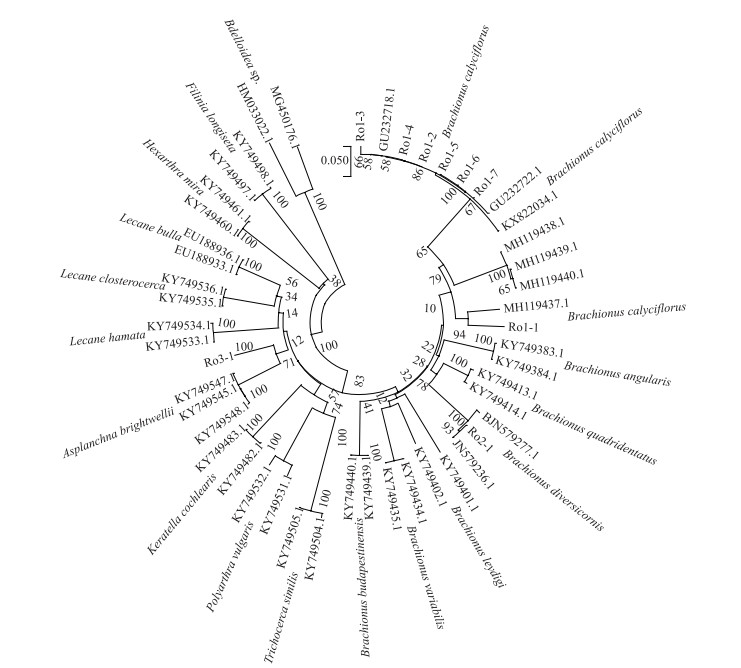
2.2 系统进化分析本研究涉及3个类群,分别基于49个、69个、48个单倍型构建3条系统发育树(图 3~5),其中桡足类系统发育树中鉴定序列为2条,下载相关同源序列为47条;枝角类系统发育树中鉴定序列为15条,下载相关序列为54条;轮虫系统发育树中鉴定序列为9条,下载相关序列为38条.观察构建的Neighbor-Joining系统发育树显示柳叶湖浮游动物不同种类休眠卵间的界限明显,置信度较高(99 % ~100 %)(图 3~5).同属物种均可聚类为一支,如:细巧华哲水蚤(Sinocalanus tenellus)先于参比序列(HM045324.1、HM045326.1)聚为一支后与同属的中华华哲水蚤(S. sinensis)聚为一支,微型裸腹溞(Moina micrura)首先与其参比序列(LC215484.1)聚为一支后与同属的远东裸腹溞(M. weismanni)聚为一支;萼花臂尾轮虫(Brachionus calyciflorus)先与其参比序列(GU232718.1、GU232722.1)独立聚为一支,后与同属的裂足臂尾轮虫(B. diversicornis)聚为一支.序列比对相似度较低的休眠卵C10、C11分别与无刺大尾溞和黑龙江低额溞的参比序列聚类至一起,可确定为相同物种.
|
图 3 基于邻接法构建的桡足类休眠卵COⅠ基因系统发育树 Fig.3 Neighbor-Joining tree resulting from analysis of COⅠ gene data of copepod resting eggs |

|
图 4 基于邻接法构建的枝角类休眠卵COⅠ基因系统发育树 Fig.4 Neighbor-Joining tree resulting from analysis of COⅠ gene data of cladocera resting eggs |
|
图 5 基于邻接法构建的轮虫休眠卵COⅠ基因系统发育树 Fig.5 Neighbor-Joining tree resulting from analysis of COⅠ gene data of rotifer resting eggs |
由于休眠卵传统鉴定较为困难,本研究用DNA条形码技术对柳叶湖表层沉积物中休眠卵种类进行初步鉴定,通过NCBI数据库比对,在物种水平上准确鉴定柳叶湖浮游动物休眠卵11种,构建了Neighbor-Joining系统发育树,发现所有物种均聚为独立分支,以属为分类阶元亦可在系统发育树中成功聚类,表明DNA条形码技术可成功应用于柳叶湖浮游动物休眠卵的种类鉴定.研究过程中鉴定出许多其他物种的休眠卵或休眠体,如斑条透明苔虫(Hyalinella punctata)、平角涡虫(Planocera reticulata)、长跗摇蚊(Tanytarsus sp.)等,同时所测序列中未鉴定出原生动物.
该技术也存在一定局限性,例如本研究中只鉴定出一种桡足类休眠卵,原因可能是桡足类休眠卵个体小,形态种类多变,在体式显微镜下挑选单个体休眠卵进行分子鉴定时易与杂质混淆,造成鉴定遗漏.另外轮虫种类休眠卵差异细微,无特殊形态特征,在对本研究中所挑选的未知休眠卵进行分子鉴定时,鉴定结果大多为萼花臂尾轮虫,其他种数量较少.除部分浮游动物休眠卵体形易区分外,大部分休眠卵与沉积物颗粒或杂质相似,显微镜下不容易挑取.
3.2 研究中出现的问题与改进首先,本研究休眠卵单次鉴定成功率约为52.88 %.镜检下许多有正常卵胚胎结构的完整卵体,由于产物浓度不达标、无扩增产物或PCR产物为假阳性,从而导致在后期PCR过程中鉴定失败.究其原因,可能主要是个别物种COⅠ序列与引物某些位点不契合致使成功率偏低,因此有学者改进设计了兼并引物jgLCO1490/jgHCO2198[24]或针对某些特定种群设计巢式PCR引物[25];卵体处于不应期内,代谢速率较低,胚胎内线粒体较少,无法提取到足够的线粒体DNA;卵体表面携带大量病毒、细菌等[26-27],干扰COⅠ基因的特异性扩增,导致扩增产物主要为细菌DNA,从而表现为假阳性.因此,当破碎枝角类的卵时应当将卵鞍剔除,以防止其携带的细菌或病毒对后续实验产生影响,或对于个体较大休眠卵DNA提取时可以采用“玻璃乳提取法”[28],以提高DNA提取的纯度及成功率.不同样点休眠卵鉴定成功率不同,靠近湖心的样点成功率较高(50 % ~60 %),而近岸的样点成功率较低(20 % ~30 %).可能是由于近岸表层沉积物中休眠卵受到某些环境因子影响使DNA受到降解,降低卵体DNA质量,例如Jiang等研究证实沉积物中的重金属含量会使太平洋纺锤水蚤(Acartia pacifica)休眠卵萌发率显著降低[29],并且表层沉积物中石油烃含量的增加会使太平洋纺锤水蚤休眠卵的存活率下降[30].
此外,若出现无法比对到具体物种的序列,可通过进一步系统发育树分析确定结果,如休眠卵C10、C11样品,样品通过系统发育分析分别与无刺大尾溞和黑龙江低额溞聚类至一支,可初步确定为相同物种.本研究中休眠卵C3、C5、C9、Ro3样品鉴定至属,C7、C8仅能鉴定至科,并且研究中所获取多条序列在数据库比对中无相关信息返回,原因主要是NCBI数据库中无相似度高的参比序列,因而无法进行比对.由于目前传统形态学鉴定仍是浮游动物鉴定的主要手段,但从事传统形态学鉴定的专家较少,鉴定主观性较强.若将形态学鉴定和DNA条形码相结合,即在分类学专家帮助下,建立本地DNA条形码数据库,这必将极大地提升物种鉴定、群落动态监测的准确性[31-33].
目前国内针对浮游动物休眠卵鉴定工作开展较少,往往都是通过单一方式鉴定.不同鉴定方式有不同的优缺点,应当将多种方式结合起来准确鉴定浮游动物休眠卵,以建立较完整的浮游动物休眠卵鉴定图库,方便进一步开展休眠卵研究工作.此外NCBI数据库中浮游动物信息不够完善,在保证精确度的同时,应加大对NCBI数据库的上传力度.随着测序技术的进步和DNA条形码技术的普及,我们相信DNA条形码技术将在物种鉴定中发挥越来越重要的作用.
4 结论本研究采用DNA条形码技术对柳叶湖表层沉积物休眠卵种类进行鉴定,成功获取17种物种信息,初步建立了柳叶湖浮游动物休眠卵图谱.在目前休眠卵传统形态学分类资料匮乏的情况下,COⅠ基因可作为浮游动物休眠卵鉴定的一种可靠、快捷、有效的方式.但NCBI数据库对于物种的收录依旧不够完整和准确,导致本研究个别样品无法鉴定至种.因此,比对数据库准确性和完整性亟待加强完善.
致谢: 感谢曾悦和刘陈在论文写作中给予的帮助.
| [1] |
Telesh IV. Plankton of the Baltic estuarine ecosystems with emphasis on Neva Estuary: a review of present knowledge and research perspectives. Marine Pollution Bulletin, 2004, 49(3): 206-219. DOI:10.1016/j.marpolbul.2004.02.009 |
| [2] |
Marcus NH. Ecological and evolutionary significance of resting eggs in marine copepods: past, present, and future studies. Hydrobiologia, 1996, 320(1/2/3): 141-152. |
| [3] |
Jiang XZ, Du NS. Fauna of China (Freshwater Cladocera). Beijing: Science Press, 1979. [蒋變治, 堵南山. 中国动物志.淡水枝角类. 北京: 科学出版社, 1979.]
|
| [4] |
Wang JJ. Freshwater rotifers of China. Beijing: Science Press, 1961. [王家楫. 中国淡水轮虫志. 北京: 科学出版社, 1961.]
|
| [5] |
Wu LS, Wang GZ, Li SJ. Advance in the diapause studies of marine planktonic copepoda. Chinese Journal of Ecology, 2003(4): 65-69. [吴荔生, 王桂忠, 李少菁. 海洋浮游桡足类滞育的研究进展. 生态学杂志, 2003(4): 65-69. DOI:10.3321/j.issn:1000-4890.2003.04.013] |
| [6] |
Radzikowski J. Resistance of dormant stages of planktonic invertebrates to adverse environmental conditions. Journal of Plankton Research, 2013, 35(4): 707-723. DOI:10.1093/plankt/fbt032 |
| [7] |
Gerhard M, Iglesias C, Clemente JM et al. What can resting egg banks tell about cladoceran diversity in a shallow subtropical lake?. Hydrobiologia, 2017, 798(1): 75-86. DOI:10.1007/s10750-016-2654-5 |
| [8] |
Huang Q, Xu SL, Xu L et al. Haplotype diversity and genetic differentiation of dormant and active populations of Daphnia galeata in Liuxihe Reservoir of Guangdong Province, southern China. J Lake Sci, 2017, 29(5): 1209-1216. [黄琦, 徐少林, 徐磊等. 广东流溪河水库盔型溞(Daphnia galeata)休眠种群与现生种群的单倍型多样性和遗传分化. 湖泊科学, 2017, 29(5): 1209-1216. DOI:10.18307/2017.0519] |
| [9] |
Mergeay J, Verschuren D, De Meester L. Daphnia species diversity in Kenya, and a key to the identification of their ephippia. Hydrobiologia, 2005, 542(1): 261-274. DOI:10.1007/s10750-004-4952-6 |
| [10] |
Santangelo JM, Lopes PM, Nascimento MO et al. Community structure of resting egg banks and concordance patterns between dormant and active zooplankters in tropical lakes. Hydrobiologia, 2015, 758(1): 183-195. DOI:10.1007/s10750-015-2289-y |
| [11] |
Hebert PDN, Gregory TR. The promise of DNA barcoding for taxonomy. Systematic Biology, 2005, 54(5): 852-859. DOI:10.1080/10635150500354886 |
| [12] |
CBOL Plant Working Group. A DNA barcode for land plants. Proceedings of the National Academy of Sciences, 2009, 106(31): 12794-12797. DOI:10.1073/pnas.0905845106 |
| [13] |
Pawlowski J, Audic S, Adl S et al. CBOL protist working group: barcoding eukaryotic richness beyond the animal, plant, and fungal kingdoms. PLoS Biology, 2012, 10(11): e1001419. DOI:10.1371/journal.pbio.1001419 |
| [14] |
Hebert PDN, Cywinska A, Ball SL et al. Biological identifications through DNA barcodes. Proceedings of the Royal Society of London. Series B: Biological Sciences, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
| [15] |
Wang CR, Wang XM, Cheng FP et al. Species composition analysis of zooplankton in Jiaozhou Bay based on mitochondrial cox1 fragment sequence. Oceanologia et Limnologia Sinica, 2011, 42(5): 711-721. [王仁诚, 王敏晓, 程方平等. 基于线粒体cox1片段序列胶州湾浮游动物物种组成分析. 海洋与湖沼, 2011, 42(5): 711-721.] |
| [16] |
Zhang X, Zang XW, Yang JH et al. Applying DNA barcoding for identification of some invertebrate macrozoobenthos at Taihu Lake Basin. Environmental Monitoring and Forewarning, 2016, 8(6): 18-25. [张翔, 张效伟, 杨江华等. 应用物种DNA条形码识别太湖流域部分底栖无脊椎动物种类. 环境监控与预警, 2016, 8(6): 18-25. DOI:10.3969/j.issn.1674-6732.2016.06.005] |
| [17] |
Machida RJ, Hashiguchi Y, Nishida M et al. Zooplankton diversity analysis through single-gene sequencing of a community sample. BMC Genomics, 2009, 10(1): 438. DOI:10.1186/1471-2164-10-438 |
| [18] |
Bucklin A, Hopcroft RR, Kosobokova KN et al. DNA barcoding of Arctic Ocean holozooplankton for species identification and recognition. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2010, 57(1/2): 40-48. |
| [19] |
Baek SY, Jang KH, Choi EH et al. DNA barcoding of metazoan zooplankton copepods from South Korea. PLoS One, 2016, 11(7): e0157307. DOI:10.1371/journal.pone.0157307 |
| [20] |
Onbé T. Sugar flotation method for sorting the resting eggs of marine cladocerans and copepods from sea-bottom sediment. Bulletin of the Japanese Society of Scientific Fisheries (Japan), 1978.
|
| [21] |
Montero-Pau J, Gómez A, Muñoz J. Application of an inexpensive and high throughput genomic DNA extraction method for the molecular ecology of zooplanktonic diapausing eggs. Limnology and Oceanography: Methods, 2008, 6(6): 218-222. DOI:10.4319/lom.2008.6.218 |
| [22] |
Prosser S, Martínez AA, Elías GM. A new set of primers for COⅠ amplification from freshwater microcrustaceans. Molecular Ecology Resources, 2013, 13(6): 1151-1155. |
| [23] |
Bucklin A, Steinke D, Blanco-Bercial L. DNA barcoding of marine metazoa. Annual Review of Marine Science, 2011, 3(1): 471-508. DOI:10.1146/annurev-marine-120308-080950 |
| [24] |
Geller J, Meyer C, Parker M et al. Redesign of PCR primers for mitochondrial cytochrome coxidase subunit I for marine invertebrates and application in all taxa biotic surveys. Molecular Ecology Resources, 2013, 13(5): 851-861. DOI:10.1111/1755-0998.12138 |
| [25] |
Robeson MS, King AJ, Freeman KR et al. Soil rotifer communities are extremely diverse globally but spatially autocorrelated locally. Proceedings of the National Academy of Sciences, 2011, 108(11): 4406-4410. DOI:10.1073/pnas.1012678108 |
| [26] |
Nealson KH. Sediment bacteria: who's there, what are they doing, and what's new?. Annual Review of Earth and Planetary Sciences, 1997, 25(1): 403-434. DOI:10.1146/annurev.earth.25.1.403 |
| [27] |
Yan DC, Dong SL, Huang J et al. White spot syndrome virus (WSSV) detected by PCR in rotifers and rotifer resting eggs from shrimp pond sediments. Diseases of Aquatic Organisms, 2004, 59(1): 69-73. |
| [28] |
Xu L, Li SJ, Wang S et al. Comparison of two methods for extracting DNA of resting eggs by Cladocera. J Lake Sci, 2014, 26(4): 632-636. [徐磊, 李思嘉, 王晟等. 两种提取枝角类休眠卵基因组DNA方法的比较. 湖泊科学, 2014, 26(4): 632-636. DOI:10.18307/2014.0419] |
| [29] |
Jiang X, Wang G, Li S et al. Heavy metal exposure reduces hatching success of Acartia pacifica resting eggs in the sediment. Journal of Environmental Sciences, 2007, 19(6): 733-737. DOI:10.1016/S1001-0742(07)60122-3 |
| [30] |
Jiang X, Wang G, Lin Q. Reduction of hydrocarbon contamination on viability of Acartia pacifica benthic resting eggs. Chinese Journal of Oceanology and Limnology, 2008, 26(1): 91-96. DOI:10.1007/s00343-008-0091-7 |
| [31] |
Li HT, Zhang BX, Gao Y et al. DNA barcoding in species identification of seashells: a case study in the ecological monitoring zone of Daya Bay, Guangdong. Biodiversity Science, 2015, 23(3): 299-305. [李海涛, 张保学, 高阳等. DNA条形码技术在海洋贝类鉴定中的实践:以大亚湾生态监控区为例. 生物多样性, 2015, 23(3): 299-305.] |
| [32] |
Lin SJ, Wang L, Zheng LM et al. Current status and future prospect of DNA barcoding in marine biology. Acta Oceanologica Sinica, 2014, 36(12): 1-17. [林森杰, 王路, 郑连明等. 海洋生物DNA条形码研究现状与展望. 海洋学报, 2014, 36(12): 1-17. DOI:10.3969/j.issn.0253-4193.2014.12.001] |
| [33] |
Li CL, Wang XM, Cheng FP et al. DNA barcoding and its application to marine zooplankton ecology. Biodiversity Science, 2011, 19(6): 805-814. [李超伦, 王敏晓, 程方平等. DNA条形码及其在海洋浮游动物生态学研究中的应用. 生物多样性, 2011, 19(6): 805-814.] |
 2020, Vol. 32
2020, Vol. 32 


