(2: 中国水产科学研究院淡水渔业研究中心农业农村部淡水渔业与种质资源利用重点实验室, 无锡 214081)
(3: 南京农业大学无锡渔业学院, 无锡 214081)
(2: Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, P. R. China)
(3: Wuxi Fisheries College, Nanjing Agricultural University, Wuxi 214081, P. R. China)
近年来,由于国家对长江资源保护力度加强,以“四大家鱼”为主的人工增殖放流使得渔业资源下降问题得到缓解.增殖放流是指将人工繁育苗种或采集到的野生苗种投放到自然水域中,突破自然种群的更新限制(recruitment limitation),修复受损水域生态系统中的生物资源,提高渔业资源产量[1].科学的增殖放流活动是有效的生态补偿措施,在维持水域生态系统平衡、促进水生生物资源恢复[2]、实现可持续的渔业生产[3]和减缓水利工程对鱼类的负面影响[4]等方面有重要作用.
鲢(Hypophthalmichthys molitrix)是我国“四大家鱼”之一[5],广泛分布于我国各大水系和湖泊[6].长江鲢增殖放流活动已开展多年,放流规模越来越大,但有关鲢增殖放流资源贡献率评估的研究较少.李小芳[7]对2010年和2011年湖北江段放流亲鱼的遗传效果进行评估,并确定2年对卵苗发生量的贡献率分别为1.33 %、6.78 %;王军红等[8]对2010-2012年长江三峡大坝至葛洲坝江段经济鱼类(鲢为主要放流和标记物种)增殖放流进行评价,确定3年回捕率分别为0.0465 %、0、0.0114 %.自此以后,很少有学者对鲢增殖放流资源贡献率展开针对性的研究.目前,评估增殖放流资源贡献率的主要方法有基于生物学统计数据的“放流效果统计量评估法”[9]和“标记放流回捕技术”[10-14].微卫星标记(microsatellite markers)作为多态性高的分子标记,被广泛应用于动物的亲缘和家系鉴定、群体遗传信息研究和遗传育种等方面[15-17].成熟的微卫星标记技术已适用于水生生物增殖放流资源贡献率评估和鱼类洄游路线研究[18-19].
鲢作为长江下游增殖放流的主要放流物种,对其增殖放流资源贡献率的评估具有代表性,一定程度上能反映放流物种整体的资源贡献率.本研究利用SSR荧光标记技术,标记8个主要原良种场(为江苏省各地政府放流工作提供苗种的单位)鲢亲本群体,结合亲子鉴定技术,评估2016-2017年增殖放流对长江江苏段鲢资源的贡献率.本次研究首次对长江下游大规模放流物种鲢资源贡献率进行评估,研究结果将为今后的增殖放流评估工作提供重要的参考依据.
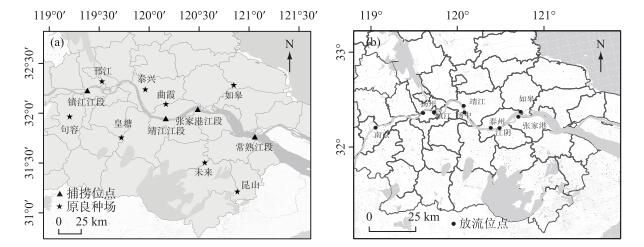
1 材料与方法 1.1 样品采集2016-2018年,共采集鲢鳍条样品1096尾.其中,2016年3月至2017年12月,采集江苏省8个原良种场鲢亲本鳍条621尾,见表 1. 2016-2017年,8个原良种场孵化的鲢幼苗陆续被放流到长江下游的9个江段(南京、镇江、扬州、扬州、泰州、靖江、江阴、张家港、如皋,位于江苏省),放流情况见表 2.放流3个月后,在1年的时间里从长江下游4个江段(镇江、靖江、张家港、常熟,距离其他省份较远,可避免其他省份增殖放流影响)陆续捕捞475尾鲢.非致命取样剪取活鱼的尾鳍,无水乙醇(江苏联海生物科技有限公司)保存,采样和放流位点见图 1.
| 表 1 8个原良种场和4个长江江段鲢采样数量 Tab. 1 The sampling number of silver carp in 8 hatcheries and 4 sections of the Yangtze River |
| 表 2 长江江苏段主要的9个放流地点鲢放流数量 Tab. 2 The releasing number of silver carp in 9 main releasing sites in Jiangsu section of the Yangtze River |
|
图 1 原良种场、长江采样位点(a)和主要放流位点(b) Fig.1 Hatcheries and the sampling sites in Yangtze River (a) and the main releasing sites (b) |
采用海洋动物组织基因组DNA提取试剂盒(北京天根生物技术有限公司)提取鳍条组织基因组DNA.提取的基因组DNA按照常规方法检测其质量,提取成功后,置于-20℃下保存.
11对荧光微卫星引物选取参照谭照君等[20],由上海睿迪生物科技有限公司合成,见表 3.
| 表 3 鲢的11对微卫星荧光引物参数 Tab. 3 Parameters of eleven pairs of fluorescent microsatellite markers of silver carp |
10 μL PCR反应体系:5 μL Premix Taq (Takara Taq Version 2.0 plus dye)(Takara,宝生物工程有限公司),0.2 μL上下游引物稀释液,1 μL DNA稀释液,3.8 μL去离子水.
PCR反应程序设定:94℃预变性120 s;94℃变性20 s,-59℃退火20 s;72℃延伸20 s;72℃延伸600 s;4℃保存,反应循环30次.
采用聚丙酰胺凝胶电泳法(Polyacrylamide Gel Electrophoresis, PAGE)检测PCR产物,凝胶浓度为8 %,取2.5 μL PCR产物,采用2.5 μL DNA Step Ladder,25~300 bp(Takara, 宝生物工程有限公司)作为参照,垂直电泳条件为250 V,150 mA,电泳2.5 h,银染后置于GBOX F3型凝胶成像分析系统(基因有限公司)上拍照.将剩余PCR产品,每3种不同荧光类型引物的扩增产物各取4 μL混合,送上海睿迪生物科技有限公司进行高效毛细电泳检测(High Performance Capillary Electrophoresis, HPCE).
1.3 数据分析使用亲缘关系分析软件Cervus 3.0.7[21]对621尾鲢亲本群体和475尾鲢回捕群体进行亲子关系分配.首先,应用软件采用基于Yates校正的Hardy-Weinberg平衡检测,当df = 1或Bonferroni校正时,计算微卫星座位的多态性参数(等位基因频率、观测杂合度和期望杂合度等),同时估计每个座位的非亲权排除率(non-exclusion probability, NEP).其次,进行模拟亲子分析:100000个重复周期,475个后代,621个未知性别的父母本,100 %的候选亲本抽样和1 %的默认分型错误率,应用LOD值(总似然比的自然对数)计算置信度[21],设置95 %的严格置信和80 %的宽松置信水平.最后,通过亲子鉴定将原良种场亲本产生的放流子代筛选出来,并计算回捕率(即放流子代数量与总捕捞数量的比值).
2 结果与分析 2.1 微卫星座位的多态性参数Cervus 3.0.7显示,11个微卫星座位共检测出191种等位基因,每个座位的等位基因种类数范围为9~36,平均值为17.36,种类数最少的座位是HLJBL203,种类数最多的座位是HLJBL217.每个座位的观测杂合度范围为0.536~0.793,平均值为0.656;每个座位的期望杂合度范围为0.597~0.941,平均值为0.794. 11个座位均表现出高度的多态性,每个座位的多态信息含量范围为0.560~0.938,平均值为0.771. Hardy-Weinberg平衡检测显示,所有的11个座位表现出不同程度地偏离Hardy-Weinberg平衡,有5个座位表现出极显著偏离Hardy-Weinberg平衡.微卫星座位多态性参数见表 4.
| 表 4 微卫星座位多态性参数 Tab. 4 Polymorphism parameters of microsatellite loci |
Cervus 3.0.7结果表明,在亲本性别未知的情况下,非亲排除率(NEP)范围为2.4 % ~42.4 %;累积非亲权排除率(combined non-exclusion probability, CEP)在第3个座位时达到95 %以上(即95.5789 %);在第10座位是达到99.9999 %以上(即99.9999994 %),见表 5.
| 表 5 鲢亲子鉴定非亲权排除率(NEP)和累积非亲权排除率(CEP) Tab. 5 Non-exclusion probability (NEP) and combined non-exclusion probability (CEP) of silver carp parentage testing |
在95 %置信度、LOD>0条件下,基于较高的座位累积非亲权排除率,621尾原良种场亲本群体和475尾捕捞鲢群体中,39尾鲢个体确认为原良种场亲本群体产生的子代.其中,镇江江段16尾,靖江江段8尾,常熟江段6尾、张家港江段9尾;对应的亲本群体,扬州市邗江长江系家鱼原种场7尾,泰兴市曲霞国平渔业合作社5尾,泰兴水产良种场4尾,皇塘水产养殖场0尾,句容良种场1尾,未来良种场8尾,昆山良种场4尾,南通市如皋水产良种场9尾,亲子鉴定结果见表 6.因此,原良种场放流的子代与总捕捞数的比率为8.21 %.
| 表 6 长江鲢群体与原良种场亲本群体的亲子鉴定结果 Tab. 6 Results of parentage testing between the Yangtze River population and hatchery population |
以往评估增殖放流资源贡献率的研究,通常采用物理和化学等方法标记放流个体,存在标记数量较小,难以覆盖全部家系的放流子代;同时存在人工标记操作流程繁琐、鱼体容易出现死亡、标记容易脱落和留存时间较短等问题,影响标记效果.本研究采用SSR标记的回捕率为8.21 %,远高于王军红等[8]采用传统方法标记以鲢为主的经济鱼类(3年回捕率分别为0.0465 %、0、0.0114 %).因此,采用SSR标记技术标记繁殖亲本,实现间接标记采样亲本的所有放流子代,消除了标记数量限制,永久标记放流个体,无人为因素造成的鱼体死亡,不受生活史影响,有利于进行长期的动态监测和鱼类洄游行为的探究[13].
本研究采用体积较小的10 μL PCR反应体系,不仅能缩短PCR反应时长,避免多重PCR反应的不足,提高PCR实验效率,而且极大地减少实验材料的消耗,降低实验成本.因此,采用10 μL PCR反应体系具有可行性,适合对大规模的群体进行亲子鉴定.
利用在引物5′端拼接荧光染料的引物进行PCR扩增,结合高效毛细管电泳法检测,可以有效区分主峰和伪影,提高基因分型的准确性[22].本研究将4种不同的荧光染料应用于11对微卫星标记,这些座位都具有高度的多态性,即PIC>0.5,高度多态性[23],可提供丰富的遗传信息,是作为亲子鉴定的有效遗传标记.本研究在第3个座位时累积排除率在95 %以上(即95.5789 %),累积排除率已经满足亲子鉴定最低标准[24],同时,本研究实际正确匹配座位数在10个位点以上,而微卫星标记的鉴别能力高,出现1~2个座位错配不影响得出正确结果[25],因此亲子鉴定结果可靠.在亲子鉴定中,累积排除率(CEP)越高,亲子鉴定准确率就越高[26],本研究中的11对微卫星座位累积非亲权排除率在99.99 %以上,鉴定准确率较高,高于李小芳[7]研究结果(14个微卫星座位,99.91 %),与成为为等[27](11对微卫星座位,放流胭脂鱼(Myxocyprinus asiaticuss)亲本,99.9983 %)、刘海映等[28](10个微卫星座位,放流三疣梭子蟹(Portunus trituberculatus)子代,99.99 %以上)等的研究结果相似.
为评估大规模的增殖放流效果,我们建立了“标记亲本-放流与再捕捞-亲子鉴定”的系统评估方法,以评估资源贡献率.第一,收集原良种场亲本鳍条样品(所有雌雄个体),包括用于供给增殖放流苗种的当年人工繁殖亲本、良种场培育池后备亲本和新引进的长江鲢,选用高度多态的微卫星座位,建立鲢亲本基因库.第二,被标记的亲本繁殖后,其子代经培育后放流至长江.第三,3个月之后,在放流区域定期定点(干流和附属河流、湖泊)进行捕捞,采集鲢DNA样品.第四,通过DNA提取、PCR反应、基因分型和亲子鉴定,确定亲本的子代数量,计算资源贡献率.此外,这种“标记亲本-放流与再捕捞-亲子鉴定”的系统评估方法同样适用于其他鱼类增殖放流效果资源贡献率的评估.
3.2 资源贡献率的影响因素在长江,鲢洄游习性(江河洄游性鱼类)可能影响贡献率的评估.每年夏天,性成熟鲢从长江的附属河流或湖泊游到长江干流水流湍急处产卵;受精卵孵化后,部分幼鱼在长江干流沿岸浅水处生长,大部分幼鱼随洪水漂流或游到附属河流和饵料丰富的湖泊中觅食生长,或在发育成稚鱼后游到附属水域,在3~5年内达到性成熟[29].同理,大量的原良种场孵化的鲢幼鱼被放流后进入附属河流或湖泊,而长江水域面积广阔,本研究在长江干流渔业捕捞水域设置4个采样点(采样点偏少),可能导致收集到的样品中放流鲢数量占比偏少.同时,由于采样时间的限制,原良种场仍然有部分亲本群体遗传信息未能采集,使得未被标记的放流子代无法鉴定出来.由此可见,长江江苏段鲢增殖放流的实际资源贡献率可能超过8.21 %.
放流物种的规格差异和渔业捕捞也可能影响鱼类增殖放流资源贡献率.湖北省将479尾(2010年)和1144尾(2011年)亲本鲢分别放流到长江监利段,李小芳[7]利用SSR标记进行亲子鉴定,确定放流鲢亲本对2010年鱼苗资源贡献率为1.33 %,对2011年鱼苗资源贡献率为6.78 %.本研究放流鱼苗对资源量的贡献率达到8.21 %,比起放流鲢亲本要高,可能受到放流规格差异、渔业捕捞的选择以及禁渔期难以覆盖整个繁殖期(鲢3~7月[30])等因素影响,使得放流的成鱼被捕获概率比鱼卵、仔稚鱼或幼鱼更高.另外,课题组在走访渔业作业现场时发现,渔获物中超过法律标准的成年鱼类(禁捕规格:如鲢,5 kg以上、65 cm以上;青鱼(Mylopharyngodon piceus),13~15 kg以上、90 cm以上[31])几乎没有被放归长江.考虑到较大的渔业捕捞强度以及长江产卵场恶化的生境,很可能存在放流的亲鱼“一次性产卵”或未产卵的情况,而育苗场环境恶化又会降低孵化率和存活率,使得放流的亲本繁殖和育苗成效低于原良种场人工繁育效果,增殖放流效果不佳.较为成功的放流案例为我国二级保护动物胭脂鱼(等同于全年禁捕),渔政部门在长江武汉至宜宾江段放流胭脂鱼幼苗,其回捕率高达16.92 % [27],高于一些经济鱼类的回捕率(例如,草鱼(Ctenopharyngodon idellus)为6.395 % ~14.316 % [32]).由此可见,渔业捕捞对增殖放流效果有一定负面影响,较长的禁渔期可以极大地提高增殖放流的资源贡献率.
此外,野生鱼类和人工养殖鱼类在放流后的扩散差异会影响回捕率.例如,野生的香鱼(Plecoglossus altivelis)与人工养殖的香鱼在河流的扩散模式也存在显著差异(野生的香鱼向上游游动,人工养殖的香鱼向下游游动),并且捕获率也保持差异[33].
3.3 增殖放流效果“量”和“质”的评估本研究表明2016-2017年鲢增殖放流活动对长江江苏段鲢资源量有较大的补充.本研究存在采样时间跨度较短、采样点设置不够密集、采样频次较少和非全部放流子代的亲鱼被采样等情况,研究结果大致反映了鲢增殖放流的资源贡献率,但无法监测增殖放流鲢群体在长江水体的种群变化趋势和扩散路线.在后续的研究中,我们将在长江干流和附属河流设置更密集的采样点,增加采样频次和时间跨度,完善亲本微卫星基因库,提高资源贡献率评估的准确性,实现对放流群体长期的动态监测.
历史上的鲢捕捞量巨大,说明历史上的鲢环境容量很大,增殖放流数量还有很大的提升空间.由于长江水域环境的恶化,环境承载力可能发生改变,而某一物种数量的增加,也会影响生态系统中该物种的捕食者、被捕食者的生物量(正效应和负效应),增强与该物种有相同食物来源生物间的竞争关系[34].放流适量的鲢和鳙可以控制浮游植物的数量,减少水华暴发的风险,提高初级生产力向次级消费者能量传递效率[35],但如果放流量过多,也有可能导致部分放流鲢因食物短缺而死亡.因此,设定合理的增殖容量不仅能提高放流物种存活率,而且可以降低对水域生态系统的风险[36],减少放流的资金浪费.林群等[37]通过构建黄河口邻近海域不同月份的Ecopath模型,初步评估了三疣梭子蟹的增殖生态容量,为渔业资源增殖养护长远发展提供了指导性建议;杨超杰等[38]基于Ecopath模型估算莱州湾朱旺人工鱼礁区刺参(Apostichopus japonicus)的增殖生态容量,并提出了阶段性的增殖管理策略.同时,实际的增殖放流活动往往同时放流多种水生生物,涉及多个不同营养级生物量的变动,势必会影响生态系统的能量转换率和稳定性,而目前增殖生态容量的研究多集中于评估某一生态系统下单一放流物种的增殖容量,很少涉及某一生态系统下组合分析多个放流物种的增殖容量配置.因此,在后续的研究中,我们将在评估放流增殖鲢资源贡献率基础的同时,对放流水域的资源和生态进行调查,构建Ecopath模型,对比放流前后生态系统各功能组的变化,推算鲢以及其他放流物种的增殖容量配置,实现增殖放流“量”的评估.
以往研究发现,增殖放流存在一定的遗传风险(genetic risk),如增加群体基因流,降低群体有效群体大小(effective population size),促进遗传同质性(genetic homogeneity)等[39-40].朱晓东等[41]利用30个微卫星标记,研究了长江中下游5个野生鲢群体,发现有10个以上基因座表现出极显著偏离的Hardy-Weinberg平衡;李小芳[7]发现放流鲢亲本后,所采集的鲢群体间呈现较低的遗传分化程度和较近的遗传距离;张敏莹等[42]发现长江下游4个放流群体呈现出一定的近交现象和群体间较低的遗传分化程度.虽然,上述研究反映出长江鲢群体遗传多样性特征可能因增殖放流发生了变化,但由于上述研究采用较为传统实验技术,或研究年代距今较为久远,未能证实鲢增殖放流已经造成了负面的遗传效果.同时,本研究主要集中于利用微卫星亲子鉴定技术评估增殖放流资源贡献率,未对各群体遗传多样性进行分析,且缺乏增殖放流前江苏段鲢群体的遗传多样性本底调查,无法评估可能存在的遗传风险.因此,在今后的研究中,我们将结合本次研究中的群体遗传数据,继续利用SSR荧光标记技术对鲢增殖放流后的群体遗传特征进行跟踪调查,探究鲢增殖放流已造成负面的遗传效果,提出有针对性的增殖放流优化方案,减少遗传风险,以实现增殖放流“质”的评估.
4 小结2016-2017年,长江江苏段增殖放流对鲢在江群体的资源贡献率在8.21 %以上.本研究是首次对长江下游大规模放流物种鲢(增殖放流活动中放流数量占比最大的物种)进行资源贡献率评估,一定程度上反映了放流物种整体的资源贡献率.本研究所采用的实验方法和技术,将对今后鲢及其他物种的增殖放流贡献率评估有重要参考价值,所得出的结论将为以后的增殖放流工作提供科学依据.
致谢: 感谢中国水产科学研究院淡水渔业研究中心李建林副研究员在实验和数据分析等方面给予的指导和帮助.感谢本研究中所涉及的原良种场的工作人员和渔民在样品采集和户外作业提供的便利和帮助.
| [1] |
Cowx IG. Stocking strategies. Fisheries Management & Ecology, 1994, 1(1): 15-30. DOI:10.1111/j.1365-2400.1970.tb00003.x |
| [2] |
Chen JH, Jiang YZ. Marine stock enhancement:Review and prospect. Journal of Fishery Sciences of China, 2010, 17(3): 610-617. [程家骅, 姜亚洲. 海洋生物资源增殖放流回顾与展望. 中国水产科学, 2010, 17(3): 610-617.] |
| [3] |
Hao YB, Yuan JL, Liu JD et al. Study on tagging techniques for release and enhancement of Xenocypris davidi and Hemibarbus maculatus. Jiangsu Agricultural Sciences, 2018, 46(22): 160-164. [郝雅宾, 原居林, 刘金殿等. 黄尾鲴和花增殖放流标记技术研究. 江苏农业科学, 2018, 46(22): 160-164.] |
| [4] |
Wen D, Zhang LL. Numerical simulation study of a broodstock rearing pond design for a fish breeding and release station. Journal of Hydroecology, 2019, 40(1): 41-47. [文典, 张陆良. 鱼类增殖放流站亲鱼池数值模拟研究. 水生态学杂志, 2019, 40(1): 41-47. DOI:10.15928/j.1674-3075.2019.01.007] |
| [5] |
Ni Y, Wu HL. Fishes of Jiangsu Province. Beijing: China Agricultural Press, 2006: 317-319. [倪勇, 伍汉霖. 江苏鱼类志. 北京: 中国农业出版社, 2006: 317-319.]
|
| [6] |
Li SF, Cai ZW, Lu WM et al. On the growth rate of Silver carps from different sources of Changjiang River and Zhujiang River. Journal of Fisheries of China, 1984, 8(3): 211-218. [李思发, 蔡正纬, 陆伟民等. 长江水系鲢鱼和珠江水系鲢鱼的生长差异. 水产学报, 1984, 8(3): 211-218.] |
| [7] |
Li XF. The genetic effect evalution of released parent Hypophthalmichthys molitrix[Dissertation]. Chongqing: Southwest University, 2012. [李小芳.鲢(Hypophthalmichthys molitrix)亲本增殖放流遗传效果评估[学位论文].重庆: 西南大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10635-1012344030.htm ]
|
| [8] |
Wang JH, Jiang W, Tang XL et al. The preliminary evaluation of economic fisheries resource enhancement and releasing effect in the Gezhouba reservoir. Freshwater Fisheries, 2014, 44(6): 100-103. [王军红, 姜伟, 唐锡良等. 长江三峡大坝-葛洲坝江段经济鱼类增殖放流效果初步评价. 淡水渔业, 2014, 44(6): 100-103. DOI:10.3969/j.issn.1000-6907.2014.06.017] |
| [9] |
Chen PM. Study on the method for assessment of enhancement effect of fishery stock. South China Fisheries Science, 2006, 2(1): 1-4. [陈丕茂. 渔业资源增殖放流效果评估方法的研究. 南方水产科学, 2006, 2(1): 1-4. DOI:10.3969/j.issn.2095-0780.2006.01.001] |
| [10] |
Navarro A, Oliva V, Zamorano MJ et al. Evaluation of PIT system as a method to tag fingerlings of gilthead seabream (Sparusauratus L.):effects on growth, mortality and tag loss. Aquaculture, 2006, 257(1-4): 309-315. DOI:10.1016/j.aquaculture.2006.02.072 |
| [11] |
Volk EC, Schroder SL, Grimm JJ. Otolith thermal marking. Fisheries Research, 1999, 43(1/2/3): 205-219. DOI:10.1016/S0165-7836(99)00073-9 |
| [12] |
Liua ZJ, Cordesb JF. DNA marker technologies and their applications in aquaculture genetics. Aquaculture, 2004, 238(1): 1-37. DOI:10.1016/j.aquaculture.2004.05.027 |
| [13] |
Skalski JR, Buchanan RA, Griswold J. Review of marking methods and release-recapture designs for estimating the survival of very small fish:examples from the assessment of salmonid fry survival. Reviews in Fisheries Science, 2009, 17(3): 391-401. DOI:10.1080/10641260902752199 |
| [14] |
Leber KM, Blankenship HL. How advances in tagging technology improved progress in a new science:marine stock enhancement. American Fisheries Society Symposium, 2011, 76: 1-12. |
| [15] |
Chistiakov DA, Hellemans B, Volckaert FA. Microsatellites and their genomic distribution, evolution, function and applications:a review with special reference to fish genetics. Aquaculture, 2006, 255. DOI:10.1016/j.aquaculture.2005.11.031 |
| [16] |
Sun JH, Ma AJ, Cui WX et al. Parentage determination of the Takifugu rubripes families by microsatellite markers. Advances in Marine Science, 2017(3): 92-103. [孙建华, 马爱军, 崔文晓等. 利用微卫星标记技术对红鳍东方鲀(Takifugu rubripes)家系系谱认证的研究. 海洋科学进展, 2017(3): 92-103.] |
| [17] |
Sun XW, Zhang XF, Zhao YY et al. Research progress and application of microsatellite marker technology for aquatic organisms. Journal of Fishery Sciences of China, 2008, 15(4): 689-703. [孙效文, 张晓锋, 赵莹莹等. 水产生物微卫星标记技术研究进展及其应用. 中国水产科学, 2008, 15(4): 689-703. DOI:10.3321/j.issn:1005-8737.2008.04.022] |
| [18] |
Oyler-Mccance S, Fike J, Lukacs P et al. Genetic mark-recapture improves estimates of maternity colony size for Indiana Bats. Journal of Fish and Wildlife Management, 2018, 9(1): 25-35. DOI:10.3996/122016-JFWM-093 |
| [19] |
Zhang XG, Tong JG, Xiong BX. Applications of microsatellite makers on studies of genetics and breeding of fishes. Journal of Agricultural Biotechnology, 2006, 14(1): 117-121. [张小谷, 童金苟, 熊邦喜. 微卫星标记在鱼类遗传及育种研究中的应用. 农业生物技术学报, 2006, 14(1): 117-121. DOI:10.3969/j.issn.1674-7968.2006.01.023] |
| [20] |
Tan ZJ, Zhang TQ, Lu CY et al. Development and characteristics of tri- and tetra- nucleotide microsatellites for Silver Carp(Hypophthalmichthys molitrix). Journal of Shanghai Ocean University, 2011, 20(3): 328-335. [谭照君, 张天奇, 鲁翠云等. 鲢三、四核苷酸重复微卫星标记的筛选及其特征分析. 上海海洋大学学报, 2011, 20(3): 328-335.] |
| [21] |
Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099-1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [22] |
Ziegle JS, Su Y, Corcoran KP et al. Application of automated DNA sizing technology for genotyping microsatellite loci. Genomics, 1992, 14(4): 1026-1031. DOI:10.1016/S0888-7543(05)80126-0 |
| [23] |
Botstein D, White RL, Skolnick M et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331. |
| [24] |
Li TM ed. Genetics of human blood groups. Shanghai: Science Press, 1987: 330-346. [李桐茂. 人类血型遗传学. 上海: 科学出版社, 1987: 330-346.]
|
| [25] |
Kong J, Zhang TS, Meng XH et al. Microsatellite markers simulation and application for parentage determination on Fenneropenaeus chinensis. Acta Hydrobiologica Sinica, 2008, 32(1): 96-101. [孔杰, 张天时, 孟宪红等. 中国对虾微卫星家系鉴定的模拟分析与应用. 水生生物学报, 2008, 32(1): 96-101. DOI:10.3321/j.issn:1000-3207.2008.01.016] |
| [26] |
Chen R, Wang G, Liu H et al. Different parental contribution to reproduction in Japanese flounder (Paralichthys olivaceus). Journal of Fishery Sciences of China, 2013, 20(4): 698-705. [陈睿毅, 王桂兴, 刘海金等. 牙鲆亲本对子代贡献率的实验研究. 中国水产科学, 2013, 20(4): 698-705.] |
| [27] |
Chen WW, Wang DQ, Wei QW et al. Effect of restocking enkancement of Chinese sucker in the middle and upper reaches of Yangtze River based on microsatellite loci. Journal of Fishery Sciences of China, 2014, 21(3): 574-580. [成为为, 汪登强, 危起伟等. 基于微卫星标记对长江中上游胭脂鱼增殖放流效果的评估. 中国水产科学, 2014, 21(3): 574-580. DOI:10.3724/SP.J.1118.2014.00574] |
| [28] |
Liu HY, Lv HB, Cui F et al. Parental contribution and genetic diversity between broodstock and offsprings in swimming crab (Portunus trituberculatus) releasing into natural waters. Fisheries Science, 2016, 35(6): 613-619. [刘海映, 吕海波, 崔帆等. 放流三疣梭子蟹遗传多样性和贡献率初步研究. 水产科学, 2016, 35(6): 613-619.] |
| [29] |
Xu CS, Ai ZQ, Xiao M. A review of influencing factors on natural reproduction of Four Major Chinese Carps in Yangtze River. Journal of China Three Gorges University:Natural Sciences, 2017, 39(4): 27-30. [许承双, 艾志强, 肖鸣. 影响长江四大家鱼自然繁殖的因素研究现状. 三峡大学学报:自然科学版, 2017, 39(4): 27-30.] |
| [30] |
Cao WX, Chang JB, Qiao Y et al. Fish resource of early life history stages in Yangtze River. Beijing: China Water Power Press, 2007: 177-180. [曹文宣, 常剑波, 乔晔等. 长江鱼类早期资源. 北京: 水利水电出版, 2007: 177-180.]
|
| [31] |
Wang L, Cui H, Cao WX. A brief discussion on fishing gear and fishing law and fishery resources protection in the Yangtze River. Journal of Hydroecology, 2007, 27(5): 108-109. [汪亮, 崔鸿, 曹文宣. 略论长江渔具渔法与渔业资源保护. 水生态学杂志, 2007, 27(5): 108-109.] |
| [32] |
Ji XF. Construction and application of microsatellite gene bank of released broodgrass carp[Dissertation]. Wuhan: Huazhong Agriculture University, 2016. [季晓芬.草鱼放流亲本微卫星基因库的构建与应用研究[学位论文].武汉: 华中农业大学, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10504-1016156183.htm ]
|
| [33] |
Tsukamoto K, Masuda S, Endo M et al. Influence of fish stocks on the recapture rate of ayu[Plecoglossus altivelis] released in the river Tsubusa[Japan]. Nippon Suisan Gakkaishi, 1990, 56(8): 1169-1176. DOI:10.2331/suisan.56.1169 |
| [34] |
Yang LL, Jiang YZ, Yuan XW et al. Evaluation on the ecosystem structure and function of Xiangshan Bay based on Ecopath model. Marine Fisheries, 2015(5): 13-22. [杨林林, 姜亚洲, 袁兴伟等. 象山港生态系统结构与功能的Ecopath模型评价. 海洋渔业, 2015(5): 13-22.] |
| [35] |
Liu QG, Zhang Z. Controlling the nuisance algae by silver and bighead carps in eutrophic lakes:disputes and consensu. J Lake Sci, 2016, 28(3): 463-475. [刘其根, 张真. 富营养化湖泊中的鲢、鳙控藻问题:争议与共识. 湖泊科学, 2016, 28(3): 463-475. DOI:10.18307/2016.0301] |
| [36] |
Jiang Y, Ling N, Yang L et al. The ecological risk of stock enhancement and the measures for prevention and control. Journal of Fishery Sciences of China, 2014, 21(2): 413-422. [姜亚洲, 林楠, 杨林林等. 渔业资源增殖放流的生态风险及其防控措施. 中国水产科学, 2014, 21(2): 413-422. DOI:10.3724/SP.J.1118.2014.00413] |
| [37] |
Ling Q, Wang J, Li ZY et al. Assessment of ecosystem energy flow and carrying capacity of swimming crab enhancement in the Yellow River estuary and adjacent waters. Chinese Journal of Applied Ecology, 2015, 26(11): 3523-3531. [林群, 王俊, 李忠义等. 黄河口邻近海域生态系统能量流动与三疣梭子蟹增殖容量估算. 应用生态学报, 2015, 26(11): 3523-3531. DOI:10.13287/j.1001-9332.20150812.009] |
| [38] |
Yang CJ, Wu ZX, Liu HY et al. The fishing strategy of Charybdis japonica and Rapana venosa and the carrying capacity of Apostichopus japonucus in Zhuwang, Laizhou Artificial Reef Ecosystem based on ecopath model. Periodical of Ocean University of China:Natural Sciences, 2016, 46(11): 168-177. [杨超杰, 吴忠鑫, 刘鸿雁等. 基于Ecopath模型估算莱州湾朱旺人工鱼礁区日本蟳、脉红螺捕捞策略和刺参增殖生态容量. 中国海洋大学学报:自然科学版, 2016, 46(11): 168-177.] |
| [39] |
Liu Q, Cui F, Hu P et al. Using of microsatellite DNA profiling to identify hatchery-reared seed and assess potential genetic risks associated with large-scale release of swimming crab Portunus trituberculatus in Panjin, China. Fisheries Research, 2018, 207: 187-196. DOI:10.1016/j.fishres.2018.05.003 |
| [40] |
Tringali MD, Bert TM. Risk to genetic effective population size should be an important consideration in fish stock-enhancement programs. Bulletin of Marine Science, 1998, 62(2): 641-659. |
| [41] |
Zhu XD, Geng B, Li J et al. Analysis of genetic diversity among silver carp(Hypophthalmichthys molitrix) populations in the middle and lower Yangtze River using thirty microsatellite markers. Hereditas(Beijing), 2007, 29(6): 705-713. [朱晓东, 耿波, 李娇等. 利用30个微卫星标记分析长江中下游鲢群体的遗传多样性. 遗传, 2007, 29(6): 705-713. DOI:10.3321/j.issn:0253-9772.2007.06.010] |
| [42] |
Zhang MY, Xu DP, Liu K et al. Analysis of genetic diversity among the artificially released populations of silver carp(Hypophthalmichthys molitrix) in the lower reach of the Yangtze River with microsatellite markers. Acta Agriculturae Universitatis Jiangxiensis, 2012, 34(1): 141-146. [张敏莹, 徐东坡, 刘凯等. 长江下游放流鲢群体遗传多样性的微卫星标记分析. 江西农业大学学报, 2012, 34(1): 141-146. DOI:10.3969/j.issn.1000-2286.2012.01.026] |
 2020, Vol. 32
2020, Vol. 32 


