(2: 东南大学无锡分校, 无锡市生物芯片重点实验室, 无锡 214135)
(3: 江苏宏众百德生物科技有限公司, 无锡 214028)
(4: 江苏省环境监测中心, 南京 210019)
(2: Wuxi Key Laboratory of Biochip, Southeast University Wuxi Campus, Wuxi 214135, P. R. China)
(3: Jiangsu MetaBio Science & Technology Co., Ltd., Wuxi 214028, P. R. China)
(4: Jiangsu Environmental Monitoring Center, Nanjing 210019, P. R. China)
藻类是一类能够以叶绿素a作为主要光合色素,没有真正根、茎、叶分化,利用单细胞的孢子或合子进行繁殖的低等植物[1]. 藻类大多存在于水体中,淡水、咸水和半咸水均有分布,但是在空气和土壤等其他环境要素中也广泛分布,可以说几乎存在于地球的每一个角落. 藻类是重要的生物资源,在生物能源、保健食品、药物开发、生物饲料等领域具有重要的作用和价值[2-4]. 同时,藻类与环境有着非常密切的关系,往往是环境的指示生物,尤其是水华、赤潮、褐潮和绿潮等藻类暴发事件频发及其对公共卫生的危害已引起世界范围内的关注[5-7]. 上述工作离不开藻类物种的鉴定,即对物种的定性. 对于物种的定义, 至今仍然存在广泛的争论. 本质上,物种是一个可检验的科学假设,经反复检验并确认无误后,即是一个合格的科学假设,为有效物种;反之,经不住检验的物种就是不合格的科学假设,则需要被合并和修订. 物种的形态特征、生理结构和遗传学信息等往往构成了科学假设的条件. 藻类物种主要包括形态种(morphological species)、进化种(evolutionary species)、生态种(ecotypic species)、发育种(phylogenetic species)和单源种(monophyletic species)5个概念[8]. 形态种是主要依赖形态学特征来进行藻类分类的概念,然而往往存在有些形态难以区分,但实际在遗传上已经发生显著分化的情况. 进化种概念适用于真核生物种的界定而不太适用于原核生物种的界定. 发育种概念中说明了属是包含属内所有种的单系类群,但是分支分析并没有延伸到属内的分类单元,即该概念没有界定“种”也是单源类群. 生态种概念强调了生态特征对藻类分类学的重要作用,也反映了物种间的进化关系,在某类群仅有形态特征和生态特征而缺乏分子数据的时候特别适用. 单源种是最适用于蓝藻种的概念,解决了大多数蓝藻种是多系起源的问题,但种的界定识别主要是基于基因类型的相似性,相似性阈值一直是争议的焦点. 因此,上述5个种的概念各有局限性[8].
藻类分类学是对藻类进行准确描述、命名、分群归类,并探索各类群之间亲缘关系远近和趋向的一门学科. 其不仅包括藻类的准确鉴定,也包含藻类进化与系统发育的内容[9]. 2006年,胡鸿钧等结合藻类系统发育和演化,将藻类分为微藻(主要包括蓝藻门(Cyanophyta)、硅藻门(Bacillariophyta)、金藻门(Chrysophyta)、黄藻门(Xanthophyta)、隐藻门(Cryptophyta)、甲藻门(Pyrrophyta)、裸藻门(Euglenophyta)、绿藻门(Chlorophyta)等)和大型藻类(主要包括红藻门(Rhodophyta)、褐藻门(Phaeophyta)、轮藻门(Charophyta)等)[10]. 2018年,Lee根据内共生学说作为藻类系统演化的基本理论,将藻类分为原核藻类和真核藻类,其中原核藻类为蓝藻门,真核藻类根据叶绿体及其内质网膜的进化分为双层被膜叶绿体(Chloroplast)类群:灰色藻门(Glaucophyta)、红藻门和绿藻门3门;单层叶绿体内质网膜(one membrane of chloroplast endoplasmic reticulum)类群:裸藻门、甲藻门、顶复门(Apicomplexa)3门;双层叶绿体内质网膜(two membranes of chloroplast endoplasmic reticulum)类群:隐藻门、异鞭藻门(Heterokontophyta)、普林藻门(Prymnesiophyta)和网绿藻门(Chlorarachniophyta)4门[1].
藻类鉴定作为藻类分类学中的重要组成部分,曾经主要以藻类的形态结构为基础,结合生理、生化和生态等特点为补充的传统分类系统为依据. 但是,对于一些形态分类特征没有出现或不典型的藻类,无法进行准确的物种鉴定. 例如脱落于群体的单个细胞藻类,孢子(akinete)、异形胞(heterocyte)、孢囊(stomatocyst)、似亲孢子(autospores)和鞭毛(flagellum)等分类特征未出现的藻类,以及需要根据生活史来进行鉴定的类群等. 同时,难以进行物种鉴定的藻类还包括一些不容易采集和显微镜难以观察的样本;生物群落复杂,但待测藻类物种丰度又非常低的样本;采样环境恶劣,采样过程会对人体造成危害,例如生长在极端环境和高辐射环境的样本[11]. 此外,基于形态的藻种鉴定对分类学专业知识依赖度高,技术人员需要经多年培养和实践才能擅长某一门类的分类,且基于形态特征描述不可避免的存在主观理解和判断的偏差,传统分类学专家队伍正在急剧缩减. 基于分子标记的藻类鉴定是通过获取和分析一段或多段基因序列实现藻种鉴定,具有高效、可靠、易于标准化等优点,是生态学研究和藻类监测的重要手段. 因此,分子标记作为藻类鉴定的通用手段已经被广泛应用[9, 12-13]. 近年来,随着藻类学的不断发展,尽管藻类分类系统不断更新和完善,然而至今尚没有一个可被国内外学者完全认可的系统. 为此,本文主要参照国内应用较为广泛的胡鸿钧等所著《中国淡水藻类——系统、分类及生态》中的分类系统[10]. 同时,由于藻类类群众多,本文仅就主要常见门类(蓝藻、硅藻、绿藻、甲藻、裸藻、隐藻、金藻、黄藻、红藻和褐藻等)分子标记的物种鉴定研究进展进行总结.
1 藻类鉴定分子标记的选择分子标记可以简单的分为两类. 第一类主要用于系统发育树的构建,要求分子标记序列足够长,且具有直系同源性,以提供尽可能多的系统发育信息,其鉴定一般采用一代Sanger测序技术. 第二类主要用于生物学评价(Bioassessment),选择的基本原则主要包括:1) 片段长度适中,既不能过长影响测序又不能过短影响文库构建和序列比对;2) DNA片段两端连接相对保守的区域,用于设计通用引物实现在尽可能多的物种中扩增该分子标记;3) DNA片段要有足够的变异,既包含足够的种系进化信息,又可将物种区分开;4)分子标记标准化,尽量采用同一DNA片段鉴定相关物种;5)所选分子标记数据库需准确和完善. 相较第一类分子标记,第二类分子标记往往采用高通量测序(NGS)技术,数据通量高但序列读长短,因此在引物的选择上和第一类有较大差异. 图 1展示了藻类分子标记法鉴定的常规流程.

|
图 1 藻类分子鉴定流程图 Fig.1 Workflow of algae identification based on DNA |
目前分子标记基因测序方法已相对成熟. 对于藻类分子标记体系建立的研究,需要依据分子标记选择的原则,并通过实验筛选出适宜待测藻类的单个或组合分子标记,其中分子标记的确定、引物选择和序列数据库构建和选择是研究的重点. 对于未知藻类的分子鉴定,了解待检藻类群体已建立分子标记的适用范围,并根据研究目的选定合适的分子标记是研究的重点. 分子标记鉴定体系的完善和未知藻类的分子鉴定,两方面研究是相辅相成的,未知藻的鉴定可以发现新种、扩充藻种分子数据库资源;而鉴定体系的构建完善,使得通过系统发育进化分析鉴定未知藻类变得更加快速和准确.
用于藻类分子鉴定的常见分子标记包括编码核糖体RNA的基因(rDNA)、核糖体基因内转录间隔区(internal transcribed spacer, ITS)以及保守蛋白编码基因. 核糖体基因普遍存在于细胞中,进化保守性相对较高,一般用于属及以上分类阶元的藻种初步鉴定. 其作为分子标记发展早,应用广泛,相应数据库较完善. 编码原核藻类的核糖体亚基包含5S rDNA、16S rDNA和23S rDNA,目前应用最广泛的是16S rDNA[9, 14];编码真核藻类的核糖体亚基的基因包含5S rDNA、5.8S rDNA、18S rDNA和28S rDNA等,其中18S rDNA在真核藻类鉴定中应用最广泛[9, 15]. 对于属内种或株系水平的区分可以结合进化速率较快的种间保守基因间区序列,如核糖体基因内转录间隔区ITS[16]. 蛋白编码基因通常用于在较低的分类阶元,如在种水平区分物种. 蛋白编码同源基因在用于藻种鉴别的同时,还可进行编码蛋白功能研究,对生态学、生物地球化学研究具有重要意义,例如固氮基因nifH[17]. 基于藻类特点和以上原则,本文列举了常见藻类在分子鉴定中常用的分子标记,详见表 1.
| 表 1 藻类鉴定常用分子标记 Tab. 1 Universal molecular markers for algae identification |
分子标记的数据库完善情况是藻类分子标记体系构建进展的重要指征. 表 2详细列举了藻类分子鉴定常用分子标记的信息、应用范围及数据库情况,其中常见的数据库有:
| 表 2 常用藻类分子标记信息 Tab. 2 Information of universal molecular makers for algae |
1) GenBank(https://www.ncbi.nlm.nih.gov/genbank/): 由美国国立生物技术信息中心(NCBI)建立和维护的,数据直接来源于测序工作者提交的序列以及与其它数据机构协作交换数据而来,数据库包含了所有已知的核酸序列和蛋白质序列,以及与它们相关的文献著作和生物学注释. 另外,Genbank每天都会与欧洲分子生物学实验室(EMBL)的数据库、日本的DNA数据库(DDBJ)交换数据,使这3个数据库实现同步更新.
2) RDP数据库: 全称“Ribosomal Database Project”,该数据库提供质控、比对、注释的细菌、古菌16S rRNA基因和真菌28S rRNA基因序列. 目前其数据库最新版本为RDP Release 11.5,于2016年9月30日更新. 更新后的数据库包含3356809条比对、注释的原核16S rRNA基因序列和125525条真菌28S rRNA基因序列.
3) GreenGene:该数据库是针对细菌和古菌16S rRNA基因的数据库,该数据库更新较慢,目前版本为2013年8月更新的gg_13_8版本. 由于是人工整理,准确度较高. 分类层级采用七级界门纲目科属种,方便理解和阅读,目前也是很多科研工作者的选择.
4) Silva数据库:是一个包含三域微生物(细菌、古菌、真核)rRNA基因序列的综合数据库. 其数据库涵盖了原核和真核微生物的小亚基rRNA基因序列(SSU,即16S和18S rRNA)和大亚基rRNA基因序列(LSU,即23S和28S rRNA),更新频繁,但其缺点是假阳性较高.
5) Cyanotype:是对NCBI数据库中具有代表性的蓝藻基因组信息的罗列,包括分类学和进化地位信息等信息,但收录条目较少,包括124个属,332个株系.
6) CyanoHub数据库:于2019年构建,目前收录245个属的16S rRNA基因序列,其收录序列通过人工矫正保证其准确性,并采用了最新的八目分类系统,属水平注释率为100 %,为蓝藻藻种鉴定和16S多样性分析提供准确而全面的数据库. 同时,作为Algae-Hub数据库的重要组成部分,将逐步建设为序列和图片共有的藻类数据库.
7) PR2(Protist Ribosomal Reference database)数据库:是针对真核微生物小亚基SSU rRNA(即18S rRNA)基因的数据库. 该数据库主要由核编码的原生生物序列构成,但为方便分析18S的高通量测序数据,也包含了后生生物、陆地植物、大型真菌和真核细胞器(线粒体、质体等)的SSU序列.
8) UNITE数据库:是针对真菌ITS序列的数据库,目前已经更新至版本7.1,更新时间为2016年11月20日,包含8180条高质量ITS参考序列. 另外,UNITE网站也可对单条ITS序列进行进行在线鉴定(https://unite.ut.ee/analysis.php).
9) BOLD数据库:收录了来源于动物、植物、真菌和原生生物基因组的cytochrome c-oxidase subunit 1(COI, cox 1)基因,包括226184个物种的4099776条序列,并且提供了在线序列比对功能.
10) Rsyst:: diatom数据库:收入的基因包括硅藻的rbcL和18S rRNA两类基因,经过3个步骤的过滤和优化,最终收录1813条高质量硅藻rbcL基因序列.
11) FunGene(functional gene)数据库:这是RDP延伸的一个针对微生物功能基因序列的数据库. 其按照功能分为抗性基因(antibiotic resistances)、植物致病基因(plant pathogenicity)、生物地球化学循环(biogeochemical cycles)、系统进化marker(phylogenetic markers)、生物降解(biodegradation)、金属循环(metal cycling)及其他(other)七类功能基因. 每类都包含几到上百种功能marker基因,可被用于功能marker基因高通量测序后的比对及功能基因引物设计等.
分子标记数据库构建之初,用于引物设计和比对分析的原始序列基本来自GenBank,GenBank数据全面但存在大量未经验证或校对的和冗余的数据,导致数据库中存在错误及大量冗余序列. 各门类专业研究人员通过序列搜集、去冗余、人工校正或分离纯化培养藻种进行分子标记测序分析等方式构建相应物种的分子标记数据库[50],集约共享,促进藻类分子数据库的完善.
2 分子标记在藻类研究中的应用进展 2.1 蓝藻门蓝藻是地球上最古老的生物之一,蓝藻类群的分类极具复杂性和特殊性,经历了多次分类系统的变革. 2014年Komárek等根据植物命名系统中的传统分类体系,结合分子和形态等多相特征,将蓝藻分类划分为更小的属,每个属包含更少的种并选定相应的模式种,形成现代蓝藻分类体系[51]. 依照该体系,蓝藻分类学者致力于完善体系中各个分子标记序列和形态信息的结合.
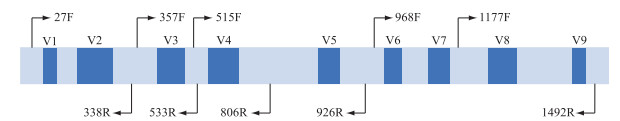
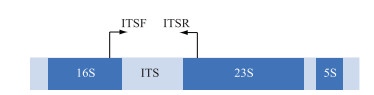
16S rDNA是目前应用最广泛的蓝藻分子标记,普遍存在于原核细胞中,含量较高、功能同源性高、遗传信息量适中、种属内较保守,一般用于揭示蓝藻的属及其以上分类单元的进化关系. 16S V4区、V3~V4区、V5~V6区、V9区以及16S全长序列(图 2)在蓝藻分类鉴定中均有应用[52],这些标记的引物通用性好,PCR扩增成功率高. 原核生物中ITS指16S-23S rDNA间隔区序列,其二级结构(图 3)为分类提供了更多信息,在蓝藻精细分类水平的划分中应用广泛[53-54]. 目前,16S rDNA和ITS的组合已经成为蓝藻定性到种水平的重要分子鉴定标记.
|
图 3 原核ITS结构示意图 Fig.3 Schematic diagram of structure of ITS for prokaryote |
藻蓝蛋白(phycocyanin, PC)是蓝藻中的一种捕光色素蛋白,cpcBA-IGS是PC基因序列上的一个间隔区,对于蓝藻种内的区分具有显著作用. 谭文华等发现cpcBA-IGS可以将惠氏微囊藻(Microcystis wesenbergii)与其他微囊藻区分开来[56]. 王中杰等基于cpcBA-IGS对太湖中的水华长孢藻(Dolichospermum flos-aquae)进行了系统发育分析,结果表明其可以区分长孢藻和其他丝状蓝藻属[57]. Choi等对来自世界各地的10株可食用蓝藻Arthrospira藻株基于16S rDNA和cpcBA-IGS进行了系统进化树构建,发现Arthrospira为单系类群,cpcBA-IGS比16S rDNA更能体现种内差异性[58]. cpcBA-IGS已成为继16S rDNA和ITS后颇具潜力的低分类水平鉴定分子标记.
近年来不断有蓝藻门新属和新种的报道,结合形态、超微结构、生理生化和分子生物学等的多相手段已经是蓝藻新物种鉴定的主流方式. 目前,多个分子标记的组合应用是蓝藻分子鉴定的主流. Cellamare等采用物种分离培养的方式对盐碱火山口湖泊中的光养微生物多样性进行分析,分子鉴定采用16S rDNA、16S-23S ITS和cpcBA-IGS等标记,描述了蓝藻门中颤藻目的2个新属和4个新种[59]. Nelson等从苔藓共生蓝藻中分离筛选到4株蓝藻,其16S rDNA未呈现差异性,但通过全基因组信息揭示了4株藻之间的差异,并通过比较基因组分析获得了一种保守基因簇vnf[60]. González-Resendiz等在对从沙漠中分离得到的颤藻目藻株进行形态、分子、生理生态等多相学的鉴定中,发现基于16S rDNA和ITS的结果与其他结果存在不完全匹配的问题[61]. Pietrasiak等在北非及南非26处沙漠位点分离得到42株丝状蓝藻,从形态和分子层面进行识别,发现了2个新种[62]. 水华危害是研究学者重点关注的问题之一,Casero等使用16S rDNA和多个产毒基因(mcyE、anaF、sxtl)作为分子标记,分析了水库中的有害藻类,发现主要产毒蓝藻为低丰度物种,采用分子生物学的方法对有害藻的评估和监控非常有必要[63]. 刘平等在湖南长沙分离培养,并鉴定得到一株产毒微囊藻,根据16S rDNA提供的信息鉴定为Microcystis sp. YFM1,并研究了其产毒特性[64]. 基于形态学和分子标记的微囊藻属内种类分类,在几十年来一直争议很大[65-66]. 微囊藻属在水体中可形成水华,并与其附生微生物形成稳定的藻类群体颗粒,共同承担相应的生态学功能,从而在水体生态系统中占据有利生态位[67]. 张军毅等利用基因组学和宏基因组学的手段从附生微生物的角度进行微囊藻属内种类的分类探索,认为微囊藻属内个别物种的宿主特异性明显[68-70]. 蓝藻的分子鉴定已经从单纯的分子标记逐步拓展到了基因组、宏基因组和宏转录组水平的探索.
2.2 硅藻门硅藻是一类营光合作用的单细胞真核生物. 在自然界中,硅藻通常是单细胞或细胞彼此连接成带状、链状、辐射状或丛状群体,浮游或着生生活. 硅藻的分类系统目前主要有3种:Hustedt(1930)系统、Krammer & Lange-Bertalot(1988)系统和Round等(1990)系统,其中Round等(1990)系统是国际上比较通用的一个系统. 我国硅藻学家金德祥先生(1978)根据壳面花纹的特征将硅藻类群分为两个纲:中心纲和羽纹纲,纲下设9目,提出了符合我国现状的硅藻分类系统[71]. 然而,近年来Lee根据叶绿素体系统演化又将硅藻归为异鞭藻门的一个纲[1]. 在分子标记建立及应用方面,硅藻门有以下特点:1)硅藻类群众多,形态学分类体系中仍存在大量同物异名的情况,需要对其进一步整合修订,而硅藻分子系统发育研究为硅藻分类提供了分子证据,加快了硅藻分类系统修订的步伐[72];2)虽然硅藻分子分类体系尚不完善,但因18S rDNA序列信息较全面,基于18S rDNA V4区扩增子的硅藻多样性研究已经广泛开展,而rbcL等其他分子标记有待进一步发展,目前主要用作对某特定类群的分类系统重构中的形态学佐证[73];3)相关数据库的命名信息不统一,一定程度上阻碍了硅藻分子标记应用的发展,如在Silva数据库中的真核藻类的分类等级为14级分类,存在亚门和亚纲等分类等级信息,而非常规的七级分类体系(界门纲目科属种).
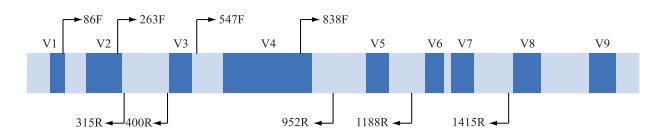
18S rDNA、28S rDNA、ITS、rbcL、psbA以及COI是常用的硅藻鉴定标记[74-75]. 18S rDNA保守区域反映物种间亲缘关系,可变区则能体现物种间的差异,适用于种以上分类阶元的鉴定. 18S rDNA结构示意图详见图 4. 18S rDNA V4区因数据库信息较全、引物通用性好、扩增成功率高而被最广泛使用. 目前对于硅藻分类体系的修订和完善通常在特定类群中进行. Stepanek等采用一组四标记(SSU、LSU、rbcL、psbC)方法对美国及日本沿海和内陆栖息地的淡水、微咸水及咸水中Amphora属的31个分类单元和Halamphora属的77个分类单元进行分子系统发育分析,结果表明在基于形态学和生态学的进化中存在一个复杂的模式. 因此,需要更加有效的形态学分类特征,提供一个可靠的属内形态学鉴定依据,从而平衡这种系统进化上的模棱两可[76]. Gargas等选择分离培养的藻种、藻种库购买的藻种以及GenBank数据库中209个物种为研究对象,进行基于核SSU和叶绿体rbcL、psbC、psbA和psaB基因的多基因系统发育分析,确定了硅藻中Orthoseira的系统发育位置,从而建议将直链目(Orthoseirales)转移到角毛藻亚纲(Chaetocerotophycidae)[77]. Gaonkar等从443株经形态学鉴定为角毛藻科(Chaetocerotaceae)的藻株中收集得到413个28S rDNA片段序列和216个18S rDNA序列,形成参考序列集并构建系统进化树,通过2个基因分子标记之间的比较,为该科藻株分子鉴定及应用提供了参考数据[78]. 因此,硅藻的分类修订需要分子标记技术发挥更多的作用[79-80].
|
图 4 18S rDNA结构示意图 Fig.4 Schematic diagram of 18S rDNA structure |
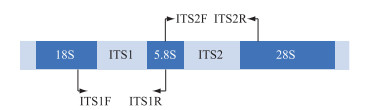
在真核生物中,18S rDNA和5.8S rDNA形成基因间区ITS1,5.8S rDNA和28S rDNA形成基因间区ITS2. 真核ITS结构示意图详见图 5. ITS是进化过程中的中度较保守区域,种间差异值一般大于14 %,常作为研究藻属内种间水平的一个分子标记. rbcL(RuBis-Co)是光合作用中促进初级CO2固定的酶,具有催化CO2还原和1, 5-二磷酸核酮糖氧化的双重功能,广泛存在于光合细菌、藻类和高等植物中,GenBank里的rbcL序列信息多且保守,作为分子标记具有通用、易扩增、易比对的特点. 龚少华等总结了DNA分子标记技术在硅藻中的应用,推荐使用的分子标记组合为rbcL-3P、5.8S rDNA和ITS2,对于Sellaphora、Pinnularia、Eunotia、Tabularia等种属的鉴定则通常使用COI[81]. COI指线粒体细胞色素C氧化酶第一亚基,无内含子且大多严格遵循母系遗传,重组频率极低. 绝大多数类群的COI序列存在明显的分子标记间隙,蕴含从种内到种间不同水平的系统发育信号,可以分辨种间物种及种内不同株系. COI基因5′末端约658 bp的片段两侧序列非常保守,易于引物设计,较高的拷贝数使得其PCR扩增简单且可靠. COI在GenBank中的资源虽少,但其高变区序列可区分关系较近的硅藻藻种,其在褐藻、红藻及硅藻鉴定中均取得良好的效果[48]. 郭立亮等对18S rDNA、ITS、UPA、COI和rbcL作为硅藻分子标记的有效性进行了评估,研究发现18S rDNA和rbcL对硅藻种类在更高分类阶元的区分表现良好,ITS和COI提供更多的基因差异性信息,而UPA过于保守不适用于硅藻的分类鉴定[24]. 在满足扩增子获取及测序成功率的条件下,COI可以应用于一些属内种水平的分类,但其在硅藻某些类群中较难扩增和测序. 综上,DNA分子标记能够对硅藻大部分种类正确鉴定,其中以18S rDNA、ITS、COI和rbcL应用最为广泛,且多标记组合已经成为必然选择.
|
图 5 真核ITS结构示意图 Fig.5 Schematic diagram of ITS structure for eukaryocyte |
绿藻种类繁多,淡水、海洋、潮湿地表、池塘等均有绿藻分布. 应用于绿藻分子鉴定的分子标记主要包括18S rDNA、ITS、rbcL、tufA等. tufA基因在藻类中普遍存在,tufA在绿藻和轮藻中为叶绿体编码,在其他藻门类为核基因编码. 绿藻tufA序列数据几乎都不含内含子,扩增成功率高;相较于LSU、rbcL、UPA而言有更高的进化速率,分辨效果明显,污染水平较低,是绿藻门分类的标准分子标记[82];tufA在GenBank中有大量可获取的序列信息,利于该分子标记的应用和发展. Sauvage等构建了包含4057个非冗余序列的tufA数据库,设计通用引物完成珊瑚着生物种Ostreobium spp.的系统发育树构建,进一步完善了tufA在绿藻分子鉴定中的应用[82].
绿藻的分类鉴定通常针对特定生境中的藻种进行,选择多个分子标记联合分析. Muggia等提出通过无菌培养获得纯藻种,记录整合藻种的形态、生境信息与基因信息,从而构建稳定可信的共球藻属(Trebouxia)分类体系和数据库. 数据库收集该属已发表藻株的超过1600个样本及序列信息,这些序列信息主要来自前期公开发表的研究、无菌培养的株系、及人迹罕至的样本,包含多位点序列数据(ITS、rbcL、cox2)信息,推动了地衣类共生藻类的研究[83]. Martins等从南极采集的样品中分离纯化培养得到一个绿藻新种,基于18S rDNA全长序列信息,发现其在进化树上形成独立的一支,并结合ITS二级结构信息确认为新种[84]. 与蓝藻和硅藻相同,分子标记在进化关系上的数据支撑,让绿藻新种的发现更易被同行接受.
此外,近年来基于叶绿体基因组的分类学手段逐渐被实践和应用. Cremen等利用来自叶绿体基因组的数据集重新评估了绿藻门羽藻目(Bryopsidales)的系统发育,通过对32个新的叶绿体基因组进行测序,并增加了分类单元的取样量,提出了一种新的分类方案,能很好的支撑羽藻目主要谱系(亚目和大多数科)的划分[85]. 朱欢等通过形态学鉴定确认了从海南热带植物园分离得到的Trentepohlia odorata,使用18S rDNA和rbcL对该新种进行了系统发育分析,并完成了该藻种的叶绿体基因组测序,获得43个核心绿藻叶绿体基因组中31个编码基因的分析结果,为石莼纲(Ulvophyceae)藻种的进化分析提供了重要参考[86]. 刘本文等基于叶绿体基因组对绿藻门胶毛藻目(Chaetophorales)的进化关系进行重构,从中国各地采集样品中分离培养属于胶毛藻目中各科的12株藻种,测定18S rDNA、ITS(包含部分28S rDNA序列)以及叶绿体基因组,同时收集了7个完整的叶绿体基因组和5个叶绿体片段基因组,同步进行叶绿体基因组结构分析、共线性分析、游动孢子萌发分析与基于叶绿体基因组的系统发育分析,有力地确定了胶毛藻目独特的分类模式. 同时发现,基于叶绿体基因组的分析结果与基于18S rDNA和ITS片段的结果具有差异[87]. 一定意义上,叶绿体基因组是一种超级分子标记,其基因序列长度远远超过其他分子标记序列,包含了巨大的信息量,是目前分子标记应用的延伸和发展. 更多样本的系统发育分析结果,更具说服力的基因组数据,以及对叶绿体基因组进化的进一步理解,或许会指向叶绿体基因组分析成为构建可靠的绿藻分子分类系统的重要突破口.
2.4 甲藻门根据林森杰等的研究,在2014年之前已经尝试应用于甲藻的分子标记包括ITS、28S rDNA、COI和cob,其中ITS和28S rDNA显示出更好的适用性[88],而甲藻的特殊性导致甲藻分子标记技术应用过程中存在较多问题. 已知的甲藻具有真核生物中最大的核基因组,且基因组结构复杂. 受到基因组内多态性的影响,ITS的使用需要其他分子标记的辅助;cob种类分辨率不理想,且引物通用性差、数据库不健全;COI虽然较cob表现出更强的种间差异性,但也无法完成对特定属内所有物种的区分[89].
经长期实践,甲藻群体关系仍有待解决,为此Janouškovec等建立了甲藻转录组的数据集,基于系统转录组学揭示甲藻进化的主要转变及群体内部的系统发育关系,通过甲藻主要分子和形态转变的重建,强调了水平基因转移在其独特核结构起源中的作用,提出了一个预测框架,用于研究甲藻群体的细胞生物学(核组织、质体进化)、分子生物学和古生物学等多方面的问题[89]. 针对ITS和cob无法区分Apocalathium属中形态不同的3个种系的问题,Annenkova等基于系统基因组学的分析方式,选用转录组数据作为分析对象,成功确认了3个种系的系统发育关系[90]. 转录组数据包含一系列同源蛋白编码基因,且目前测序成本较全基因组低、分析组装难度较全基因组小,是解决甲藻分子鉴定及分类体系存在问题的有效方法. 但基于转录组多个同源蛋白序列的分析方法并非万全之策,一些非系统发育信号的干扰会给分析带来障碍,因此结合分子标记、甲藻生境信息的多相学分析方法,将为甲藻的分类鉴定带来更多可能.
2.5 裸藻门和隐藻门裸藻门和隐藻门类下的分支相较于其他藻类比较少,采用分子标记法对属及其以上的分类阶元具有较好的效果. 裸藻是单细胞真核生物的一个单系类群,其特征是具有二次内共生的叶绿体. Ciugulea等采用分子标记和电镜技术对裸藻存在争议的2个属Strombomonas和Trachelomonas的物种多样性进行了分析,指出需要收集来自单细胞分离藻种的形态学和分子数据,找到特有而稳定的形态学特征来匹配分子鉴定的物种,才能实现两个属的区分[91]. 2010年,Kim等利用核18S rDNA、叶绿体16S rDNA和叶绿体23S rDNA分析了108株裸藻,得到两个大分支的进化树,同时结合该两大分支中藻株在超微结构上的差异,建议从原裸藻科(Euglenaceae)中分离出一个新科Phacaceae,在Phacaceae下设Phacus、Lepocinlis和Discoplastis 3个属[35]. 2013年,Kim等基于分离得到的46个藻株对Euglenaceae下的Monomorphina属进行了深入的分析,发现该属中的藻种具有广泛的基因多样性,建议将核基因组18S rDNA的二级结构作为各个藻种区分的重要标识之一[92]. 随后在2014年,Kim等又基于细胞质核糖体大小亚基(18S rDNA和28S rDNA)以及叶绿体核糖体大小亚基序列,结合形态学特征对Phacaceae下Phacus属内系统发育关系进行了分析,再一次丰富了裸藻的精细分类[93]. 王艳梅使用16S rDNA、18S rDNA和23S rDNA三种基因序列对8种不同形态性状的裸藻进行系统发育关系分析,发现这几种基因序列对裸藻性状进化具有指示作用[36]. 2020年,Łukomska-Kowalczyk等利用形态学和SSU rDNA数据对波兰和捷克境内的常见种类扁裸藻属进行了较为系统的研究,对于确认的19个种类,不仅修订了分类学特征,而且指定了表位型(epitypes)[94]. 2021年,Gumińska等利用18S rDNA V2高可变区的特殊引物对分子标记和形态学方法进行了对比研究,发现几乎90 % 的序列可以鉴定到种水平,同时前者所获50个物种在光镜下几乎均有发现,验证了分子鉴定的有效性和可靠性[95]. 此外,对裸藻叶绿体基因组的分析显示其具有属内特异性,主要体现在基因簇排列以及内含子多样性,可用来识别基因组的元特征从而补充完善裸藻的系统发育分析[96-97].
隐藻是重要的光合单细胞真核生物群,其质体来源于内共生的红藻,宿主细胞中保留4个不同的基因组(宿主核、线粒体、质体和藻核型体). 隐藻分子标记的选择主要包括18S rDNA、ITS、rbcL等,目前应用最多的为18S rDNA. 夏爽在对隐藻的分类研究中指出应用分子标记的系统发育学分析显示了隐藻与红藻之间密切的亲缘关系,且分子系统发育分析的结果与基于形态学和色素的隐藻门分类系统在属以上水平上相一致,但是在属以下水平上存在分歧. 例如基于系统发育分析无法很好区分弯隐藻属和隐藻属内的种[98],通过形态学观察与18S rDNA分子系统发育分析相结合可有效改善隐藻鉴定效果. Majaneva等应用18S rDNA和ITS分子标记完成了采集样品中隐藻纲的分子系统发育分析[37]. Kim等测定了4株典型隐藻的叶绿体基因组并与数据库中已有数据集进行比较分析,基于88个蛋白构建系统发育树,该研究拓展了藻类数据库的广度,有助于确定细胞器基因组进化的总体趋势[99]. 对于隐藻的线粒体,Kim等同样对隐藻7个代表藻种的线粒体基因组进行了分析,发现其线粒体基因组保留了其他真核生物中发现的几乎所有基因,基因聚类分析表明,隐藻具有与雅各巴虫(Jakoba)和异养鞭毛虫(Reclinomonas)相似的基因序列,分别基于叶绿体和线粒体基因组数据集的系统发育树的比较分析显示,隐藻宿主和内共生体成分的不同进化历史[100]. 由于裸藻和隐藻进化的特殊性,分子层面的系统发育分析对藻种的分类和进化史研究具有更强的指导作用,叶绿体和线粒体基因组的测定及分析为该两大类藻的分类鉴定提供了更多支撑.
2.6 金藻门和黄藻门金藻门和黄藻门主要分子标记选择包括18S rDNA、ITS、psaA、rbcL和COI等. 杨泽民对金藻部分类群的分子系统进行了研究,测定了我国常见的包括等鞭金藻属(Isochrysis)、棕囊藻属(Phaeocystis)和巴夫藻属(Pavlova)在内的8株金藻的18S rDNA基因序列,并对相关属种进行了系统发育关系分析,同时将psaA和psbA基因序列和RNA二级结构应用于特定藻属的亲缘关系分析[39]. 姜小蝶等在金藻门锥囊藻科的研究中,采用18S rDNA、ITS1、5.8S rDNA和ITS2等分子标记,同时结合形态学特征提议了新种Dinobryon taiyuanensis[40]. Daniel等分离得到金藻纲两个新种,测定并分析18S rDNA和rbcL基因序列及其在进化树中的位置[41]. 金藻在分子水平的研究并不深入,为了解金藻的营养策略及系统发育关系,Beisser等完成了18株金藻转录组测序及深度分析,为金藻的系统发育研究及物种多样性分析提供了新方法和新方向[101]. Bråte等完成了金树藻Hydrurus faetidus的基因草图及转录组图谱,这些新的数据有助于更好地理解金藻的进化和生态作用,以及在更大的系统发育规模上解决分枝模式问题[102].
黄藻门的分子分类系统发展研究较金藻门薄弱,Rybalka等在区分黄藻门中亲缘关系较近物种的研究中使用高变性的psbA/rbcL间隔区序列和rbcL分子标记,为黄藻门黄丝藻科(Tribonemataceae)的物种区分提供了大量信息[103]. Negrisolo等基于18S rDNA和rbcL的分析揭示了黄藻门进化过程中的形态趋同特征,发现这2个分子标记组合可以很好地区分一些近缘问题物种,对于黄藻门是可靠的分子标记[42]. 基于分子标记的黄藻分类研究较少,通常存在用于研究的藻株数量过少或只关注于某一狭窄的黄藻类群等问题. 2009年,Maistro等为了研究黄藻的分类系统,选用18S rDNA、rbcL和psaA 3种分子标记对31个藻株进行测定,结合已有黄藻相应分子标记的数据集对单个分子标记及不同分子标记组合(SSU、psaA、rbcL、rbcL+psaA、SSU+psaA、SSU+rbcL、SSU+rbcL+psaA)进行系统发育分析,确定了黄藻纲中的主要类群的进化关系[104].
2.7 红藻门和褐藻门红藻和褐藻是海洋中的常见藻类,两者体形庞大、形态差异大且易变化,形态学分类较困难. 已研究应用于红藻的分子标记包括COI、UPA、28S rDNA、rbcL、tufA、psbA等基因. Zuccarello等分别基于rbcL单个分子标记以及18S rDNA、psbA、tufA和rbcL间隔序列4种分子标记组合对红藻门Stylonematales纲的系统发育关系进行分析,探究世界各地样品的生物地理学关系,也为该类群的分类体系提供了参考数据集[105]. Lin等利用rbcL序列分析了台湾和印度洋地区红藻Yonagunia属和部分海膜藻科(Halymeniaceae)的系统发育,很好地解决了Yonagunia属内种的系统发育问题,可用于阐明物种边界、多样性和生物地理格局,并描述了分离自台湾的2个新种[106]. Gomes等应用分子和形态学相结合的方法对来自巴西22个不同采样位点的68株盖氏藻属(Ceramieae)进行rbcL、cox1、rbcL+cox1、rbcL+cox1+LSU 4种分子标记组合的系统发育分析,发现了该属的4个新种[107]. 2014年茅云翔等综述了红藻DNA分子标记研究进展,对常用分子标记COI、UPA、28S rDNA和rbcL的优缺点进行了总结,COI和rbcL被认为较适合作为红藻门的分子标记,为国内红藻分子标记研究提供了参考[108].
褐藻的分子系统学研究主要集中于rDNA、COI、rbcL和rbcS等序列. 薛红凡对用于褐藻分子鉴定的COI、ITS和28S rDNA进行了评估,结果发现COI的扩增效率高于其他2个基因,在鉴定能力方面,COI基因适合鉴定种水平的物种[109]. COI相对于rbcL无法揭示种间的系统进化关系,但仍可用于种内的物种鉴定,所以2个基因相结合才能准确鉴定该类群并反映它们的系统进化关系. Montecinos等基于COI-5P和ITS1两种分子标记对褐藻门的Ectocarpus属进行系统发育分析,发现其至少存在15个隐存种[110]. Ng等利用ITS2核标记和COI线粒体标记对西北太平洋(NWP)分布范围内采集的褐藻种群连通性和系统地理结构的进化影响进行了研究,解释了海藻的种群连接和分布问题[111]. Bruno等使用COI、23S rDNA和23S-tRNAVal intergenic spacer (mt-spacer)3个分子标记分析了Cystoseira 的系统发育关系,将该群体划分为更精细的3个进化枝,对更准确地分离和鉴定天然化合物以及实施目标物种保护措施具有重要意义[112]. Ortega等致力于创建一种新的大型藻类分子识别方法,从组织样本中寻找能够识别海洋植物的短条形码并创建了DNA参考数据库,用于从沿海沉积物的eDNA中识别大型植物,并选用rbcL、matK、trnL、ITS2、COI和18S rDNA等分子标记的18对引物,对海草、红树林和海洋大型藻类(绿藻门、红藻门和褐藻科)进行了扩增分析,促进了分子标记法在大型藻类监测中的应用[113].
Saunders等总结了包括褐藻、红藻、绿藻在内的大型藻类和硅藻等的DNA分子标记研究方法,提出了针对不同藻群体的双分子标记组合法:LSU D2/D3作为这几类藻的二级分子标记,用于种间水平的分类,分别配合用于种内水平分类的COI-5P、rbcL、tufA作为褐藻和红藻、硅藻、绿藻的一级分子标记,能达到较好的分类效果. 该研究提供了从DNA提取、分子标记选用到引物设计及序列扩增测序的详细技术方案,为构建大型藻类分子分类体系提供了行之有效的参考方法[26]. 在很长一段时间内,针对特定环境及特定类群的多个分子标记相结合的系统发育分析仍是红藻和褐藻分类系统研究的主要内容.
3 展望 3.1 测序技术的发展测序技术的不断发展极大地促进了分子标记在藻类鉴定领域的运用. 2020年,Hatfield等首次利用纳米孔测序技术(Oxford Nanopore technologies)的超读长优势,借助MinION测序平台(MinION sequencing platform)以真核藻类为研究对象,目标扩增区域包含了几乎整个18S rDNA、完整的ITS1、5.8S rDNA、ITS2以及28S rDNA的D1和D2,有效地完成了甲藻有害水华物种的鉴定. 这种技术成本低,测序读长长(超过20 kb),而且设备体积小便于携带,被认为是未来解决野外藻类物种快速检测和多样性分析的重要手段[114]. 目前,测序成本进一步下降,三代测序技术趋于成熟,生物信息学分析方法逐渐标准化,线粒体基因组、叶绿体基因组、核基因组、转录组和宏基因组等也逐渐成为藻类系统进化和分类鉴定的重要补充. 尤其是叶绿体基因组包含大量的遗传信息,相对单个或少数几个分子标记而言,叶绿体基因组提供了更多可供分析的位点,尤其适用于难以鉴定的近缘物种或分类混乱的类群[115]. 此外,测序技术的不断突破与生物信息学发展也极大地促进了分子标记的筛选工作. 尽管目前不断有新的分类标记和引物被报道和应用[116-117],但是充分利用基因组信息筛选候选基因,仍将是一段时间内藻类分子标记研究的主要任务. 单一分子标记已经很难准确完成藻类鉴定,多标记组合已是必然选择.
3.2 数据库的建设和完善基于分子标记的藻类鉴定离不开基础数据库的构建及完善,尤其是在种水平上的鉴定[118]. 然而,藻类是一个庞大的生物体系,藻类类群的复杂性和生境的特殊性,往往给该项工作带来了很大的挑战. 数据库面临的主要挑战是不断扩充更多的物种信息,以及提升数据准确性和剔除冗余信息. 样品采集及藻种分离培养方法,三代测序和单细胞捕获微流控技术,以及生物信息学等技术的发展都是影响数据库建设的重要方面. AlgaeBase数据库(www.algaebase.org)是一个在国际上广泛使用的综合性藻类数据库,遗憾的是其缺乏DNA序列方面的内容. 近年来,为了整合藻类形态学和DNA方面的信息,在国内藻类学家的努力下,Algae-Hub数据库(www.algaehub.cn)获得了一定的发展. 值得注意的是,一个准确、全面和有效的数据库往往需要形态学、生理生态、分子生物学等内容的同步构建. 同时,需要各个类群和不同研究方向的藻类学者发挥自身优势,在现有研究成果的基础上,更加积极和系统性地投入,才能保障数据库的准确性、全面性和系统性.
3.3 藻类快速检测方法目前,部分科研人员,尤其是一些藻类监测部门对于快速藻类检测方法具有很大的需求. 而将分子标记与微流控、生物芯片等技术结合是一个备受期待的解决方案[119-120]. 尽管生物芯片具有快速和低成本的优势,但是准确性和稳定性一直是其难以逾越的瓶颈. 这些瓶颈的突破主要依赖于目标物种特异性DNA序列的筛选,但由于过长的DNA序列会产生非线性效应,且位置结构影响杂交效果,故特异性DNA序列长短一般小于200 bp,进一步限制了生物芯片检测技术的应用范围. 随着藻类数据库的不断完善,尤其是分子生物学数据的不断扩充和优化,以及更准、更长和更快的高通量测序技术的发展[121-122],基于测序技术的藻类高通量快速检测必将会成为一种主流藻类鉴定方法.
3.4 多相学的藻类鉴定方法分子标记法能够在基因水平反映物种系统发育和进化,用于物种鉴定. 而形态学作为一种经典鉴定方法,往往更加容易开展和推广. 然而,对于藻类鉴定,分子标记法和形态学已经不再是“非此即彼”的选择了. 因此,结合形态学、生理生化、分子生物学特征等的多相学方法,才能全面而准确地完成鉴定工作[123-124]. 目前,多相学的鉴定方法已在蓝藻门[125-126]、绿藻门[127-128]、硅藻门[129-130]、甲藻门[131]和红藻门[132]等类群开展了广泛的应用和实践.
4 结论尽管分子标记已经成为物种鉴定的一种常用手段,但是由于藻类类群众多且差异很大,分子标记的选择仍然很难. 此外,分子标记数据库也成为影响选择的重要因素之一. 总体而言,核糖体DNA基因相对保守,常用于较低分类阶元,例如属的鉴定;ITS序列进化压力较小,进化速度快,可以辅助应用在更低分类阶元,如种的鉴定;对于一些蛋白编码序列,可以进行种内株系的区分. 此外,在陆生植物中有良好表现的rbcL也是藻类鉴定效果较好的分子标记,COI对真核藻类尤其硅藻的鉴定效果显著,tufA在绿藻门的分类中具有一定的优越性,UPA也同样适合于藻类的研究. 值得注意的是,尽管单一分子标记在多样性调查和生物学评价中具有一定优势,但是对于一些特殊类群往往难以鉴定. 因此,分子标记的组合应用以及针对特殊类群的特定分子标记筛选将是一种必然选择. 此外,基于分子标记的藻类鉴定离不开基础数据库的建设和完善,藻类分离培养、测序、生物信息学等技术的不断突破也必将给数据库建设带来新的机遇.
| [1] |
Lee RE. Phycology. Cambridge: Cambridge University Press, 2018, 1-535. DOI:10.1017/9781316407219 |
| [2] |
Jones CS, Mayfield SP. Algae biofuels: Versatility for the future of bioenergy. Current Opinion in Biotechnology, 2012, 23(3): 346-351. DOI:10.1016/j.copbio.2011.10.013 |
| [3] |
Besednova NN, Zaporozhets TS, Kuznetsova TA et al. Extracts and marine algae polysaccharides in therapy and prevention of inflammatory diseases of the intestine. Marine Drugs, 2020, 18(6): 289. DOI:10.3390/md18060289 |
| [4] |
Barsanti L, Gualtieri P. Algae: Anatomy, biochemistry, and biotechnology, Second Edition. Pisa, Italy: CRC Press Taylor & Francis Group, 2014, 1-361. DOI:10.1201/b16544 |
| [5] |
Huisman J, Codd GA, Paerl HW et al. Cyanobacterial blooms. Nature Reviews Microbiology, 2018, 16(8): 471-483. DOI:10.1038/s41579-018-0040-1 |
| [6] |
Ho JC, Michalak AM, Pahlevan N. Widespread global increase in intense lake phytoplankton blooms since the 1980s. Nature, 2019, 574(7780): 667-670. DOI:10.1038/s41586-019-1648-7 |
| [7] |
Qin BQ, Zhu GW, Gao G et al. A drinking water crisis in Lake Taihu, China: Linkage to climatic variability and lake management. Environmental Management, 2010, 45(1): 105-112. DOI:10.1007/s00267-009-9393-6 |
| [8] |
Li XC. The taxonomy and molecular diversity of filamentous cyanobacteria Cylindrospermopsis, Raphidiopsis and Sphaerospermopsis [Dissertation]. Wuhan: Institute of Hydrobiology, Chinese Academy of Sciences, 2016. [李小闯. 丝状蓝藻拟柱孢藻、尖头藻和拟圆孢藻的分类学和分子多样性研究[学位论文]. 武汉: 中国科学院水生生物研究所, 2016. ]
|
| [9] |
Manoylov KM. Taxonomic identification of algae (morphological and molecular): Species concepts, methodologies, and their implications for ecological bioassessment. Journal of Phycology, 2014, 50(3): 409-424. DOI:10.1111/jpy.12183 |
| [10] |
Hu HJ, Wei YX. The freshwater algae of China—Systems, taxonomy and ecology. Beijing: Science Press, 2006. [胡鸿钧, 魏印心. 中国淡水藻类——系统、分类及生态. 北京: 科学出版社, 2006.]
|
| [11] |
Bellinger EG, Sigee DC. Introduction to freshwater algae. In: Freshwater algae identification, enumeration and use as bioindicators, 2015, 1-42. DOI:10.1002/9781118917152.ch1 |
| [12] |
Kim K, Park C, Yoon Y et al. Harmful cyanobacterial material production in the north Han river (south Korea): Genetic potential and temperature-dependent properties. International Journal of Environmental Research and Public Health, 2018, 15(3): E444. DOI:10.3390/ijerph15030444 |
| [13] |
Jiang YG, Xiao P, Liu Y et al. Targeted deep sequencing reveals high diversity and variable dominance of bloom-forming cyanobacteria in eutrophic lakes. Harmful Algae, 2017, 64: 42-50. DOI:10.1016/j.hal.2017.03.006 |
| [14] |
Han D, Fan Y, Hu Z. An evaluation of four phylogenetic markers inNostoc: Implications for cyanobacterial phylogenetic studies at the intrageneric level. Current Microbiology, 2009, 58(2): 170-176. DOI:10.1007/s00284-008-9302-x |
| [15] |
Gaonkar CC, Piredda R, Minucci C et al. Annotated 18S and 28S rDNA reference sequences of taxa in the planktonic diatom family Chaetocerotaceae. PLoS One, 2018, 13(12): e0208929. DOI:10.1371/journal.pone.0208929 |
| [16] |
Hajibabaei M, Singer GAC, Hebert PDN et al. DNA barcoding: How it complements taxonomy, molecular phylogenetics and population genetics. Trends in Genetics, 2007, 23(4): 167-172. DOI:10.1016/j.tig.2007.02.001 |
| [17] |
Warshan D, Bay G, Nahar N et al. Seasonal variation in nifH abundance and expression of cyanobacterial communities associated with boreal feather mosses. The ISME Journal, 2016, 10(9): 2198-2208. DOI:10.1038/ismej.2016.17 |
| [18] |
Seo PS, Yokota A. The phylogenetic relationships of cyanobacteria inferred from 16S rRNA, gyrB, rpoC1 and rpoD1 gene sequences. The Journal of General and Applied Microbiology, 2003, 49(3): 191-203. DOI:10.2323/jgam.49.191 |
| [19] |
Tomitani A, Knoll AH, Cavanaugh CM et al. The evolutionary diversification of cyanobacteria: Molecular-phylogenetic and paleontological perspectives. PNAS, 2006, 103(14): 5442-5447. DOI:10.1073/pnas.0600999103 |
| [20] |
Wu ZX, Shi JQ, Xiao P et al. Phylogenetic analysis of two cyanobacterial genera Cylindrospermopsis and Raphidiopsis based on multi-gene sequences. Harmful Algae, 2011, 10(5): 419-425. DOI:10.1016/j.hal.2010.05.001 |
| [21] |
Dyble J, Paerl HW, Neilan BA. Genetic characterization of Cylindrospermopsis raciborskii (cyanobacteria) isolates from diverse geographic origins based on nifH and cpcBA-IGS nucleotide sequence analysis. Applied and Environmental Microbiology, 2002, 68(5): 2567-2571. DOI:10.1128/aem.68.5.2567-2571.2002 |
| [22] |
Park HK, Kwon MA, Lee HJ et al. Molecular verification of Bloom-forming Aphanizomenon flos-aquae and their secondary metabolites in the nakdong river. International Journal of Environmental Research and Public Health, 2018, 15(8): E1739. DOI:10.3390/ijerph15081739 |
| [23] |
Moniz MB, Kaczmrska I. Barcoding diatoms: Is there a good marker?. Molecular Ecology Resources, 2009, 9(S1): 65-74. DOI:10.1111/j.1755-0998.2009.02633.x |
| [24] |
Guo LL, Sui ZH, Zhang S et al. Comparison of potential diatom 'barcode' genes (the 18S rRNA gene and ITS, COI, rbcL) and their effectiveness in discriminating and determining species taxonomy in the Bacillariophyta. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Pt 4): 1369-1380. DOI:10.1099/ijs.0.000076 |
| [25] |
Nakov T, Ruck EC, Galachyants Y et al. Molecular phylogeny of the Cymbellales (Bacillariophyceae, Heterokontophyta) with a comparison of models for accommodating rate variation across sites. Phycologia, 2014, 53(4): 359-373. DOI:10.2216/14-002.1 |
| [26] |
Saunders GW, Kucera H. An evaluation of rbcL, tufA, UPA, LSU and ITS as DNA barcode markers for the marine green macroalgae. Cryptogamie Algologie, 2010, 31(4): 487-528. |
| [27] |
Song HY. Taxonomic and phylogenetic studies of the Chlorellaceae from China[Dissertation]. Wuhan: Institute of Hydrobiology, Chinese Academy of Sciences, 2018. [宋会银. 中国小球藻科的分类及系统发育研究[学位论文]. 武汉: 中国科学院水生生物研究所, 2018. ]
|
| [28] |
Wang QH. Taxonomic and phylogenetic studies of Scenedesmaceae(Sphaeropleales, Chlorophyta)from China[Dissertation]. Wuhan: Institute of Hydrobiology, Chinese Academy of Sciences, 2019. [王清华. 中国栅藻科绿藻的分类学及系统发育学研究[学位论文]. 武汉: 中国科学院水生生物研究所, 2019. ]
|
| [29] |
Lin S, Zhang H, Hou Y et al. High-level diversity of dinoflagellates in the natural environment, revealed by assessment of mitochondrial cox1 and cob genes for Dinoflagellate DNA barcoding. Applied and Environmental Microbiology, 2009, 75(5): 1279-1290. DOI:10.1128/AEM.01578-08 |
| [30] |
Sherwood AR, Presting GG. Universal primers amplify a 23S rDNA plastid marker in eukaryotic algae and cyanobacteria. Journal of Phycology, 2007, 43(3): 605-608. DOI:10.1111/j.1529-8817.2007.00341.x |
| [31] |
Ki GS. Nuclear 28S rDNA phylogeny supports the basal placement of Noctiluca scintillans (Dinophyceae, Noctilucales) in dinoflagellates. Journal of Phycology, 2010, 46(2): 111-120. DOI:10.1016/j.ejop.2009.11.001 |
| [32] |
Mordret S, Piredda R, Vaulot D et al. dinoref: A curated dinoflagellate (Dinophyceae) reference database for the 18S rRNA gene. Molecular Ecology Resources, 2018, 18(5): 974-987. DOI:10.1111/1755-0998.12781 |
| [33] |
Łukomska-Kowalczyk M, Karnkowska A, Krupska M et al. DNA barcoding in autotrophic euglenids: Evaluation of COI and 18S rDNA. Journal of Phycology, 2016, 52(6): 951-960. DOI:10.1111/jpy.12439 |
| [34] |
Marin B, Palm A, Klingberg MAX et al. Phylogeny and taxonomic revision of plastid-containing euglenophytes based on SSU rDNA sequence comparisons and synapomorphic signatures in the SSU rRNA secondary structure. Protist, 2003, 154(1): 99-145. DOI:10.1078/143446103764928521 |
| [35] |
Kim JI, Shin W, Triemer RE. Multigene analyses of photosynthetic euglenoids and new family, Phacaceae (euglenales). Journal of Phycology, 2010, 46(6): 1278-1287. DOI:10.1111/j.1529-8817.2010.00910.x |
| [36] |
Wang YM. Phylogenetic and morphological evolution of green Euglenophytes[Dissertation]. Taiyuan: Shanxi University, 2019. [王艳梅. 绿色裸藻类植物系统发育及性状进化[学位论文]. 太原: 山西大学, 2019. ]
|
| [37] |
Majaneva M, Remonen I, Rintala JM et al. Rhinomonas nottbecki n. sp. (Cryptomonadales) and Molecular Phylogeny of the Family Pyrenomonadaceae. Journal of Eukaryotic Microbiology, 2014, 61(5): 480-492. DOI:10.1111/jeu.12128 |
| [38] |
Hoef-Emden K, Marin B, Melkonian M. Nuclear and nucleomorph SSU rDNA phylogeny in the cryptophyta and the evolution of cryptophyte diversity. Journal of Molecular Evolution, 2002, 55(2): 161-179. DOI:10.1007/s00239-002-2313-5 |
| [39] |
Yang ZM. Molecular systematics of Chrysophyta groups[Dissertation]. Guangzhou: Jinan University, 2005. [杨泽民. 金藻类部分类群的分子系统学研究[学位论文]. 广州: 暨南大学, 2005. ]
|
| [40] |
Jiang XD, Nan FR, Lv JP et al. Dinobryon taiyuanensis (Chrysophyta, Dinobryaceae), a new freshwater species described from Shanxi Province, China. Phytotaxa, 2019, 404(1): 41. DOI:10.11646/phytotaxa.404.1.4 |
| [41] |
Daniel R, Lenka P, Linda N. Two new Kremastochrysopsis species, K. austriaca sp. nov. and K. Americana sp. nov. (Chrysophyceae). Journal of Phycology, 2020, 56(1): 135-145. DOI:10.1111/jpy.12937 |
| [42] |
Negrisolo E, Maistro S, Incarbone M et al. Morphological convergence characterizes the evolution of Xanthophyceae (Heterokontophyta): evidence from nuclear SSU rDNA and plastidial rbcL genes. Molecular Phylogenetics and Evolution, 2004, 33(1): 156-170. DOI:10.1016/j.ympev.2004.04.016 |
| [43] |
Freshwater DW, Fredericq S, Butler BS et al. A gene phylogeny of the red algae (Rhodophyta) based on plastid rbcL. PNAS, 1994, 91(15): 7281-7285. DOI:10.1073/pnas.91.15.7281 |
| [44] |
Robba L, Russell SJ, Barker GL et al. Assessing the use of the mitochondrial cox1 marker for use in DNA barcoding of red algae (Rhodophyta). Am J Bot, 2006, 93(8): 1101-1108. DOI:10.3732/ajb.93.8.1101 |
| [45] |
Sciuto K, Moschin E, Alongi G et al. Tethysphytum antarcticum gen. et sp. nov. (Hapalidiales, Rhodophyta), a new non-geniculate coralline alga from Terra Nova Bay (Ross Sea, Antarctica): morpho-anatomical characterization and molecular phylogeny. European Journal of Phycology, 2021, 1-12. DOI:10.1080/09670262.2020.1854351 |
| [46] |
Mattio L, Payri C. Assessment of five markers as potential barcodes for identifying Sargassum subgenus Sargassum species (Phaeophyceae, Fucales). Cryptogamie Algologie, 2010, 31(4): 467-485. |
| [47] |
Lane CE, Lindstrom SC, Saunders GW. A molecular assessment of northeast Pacific Alaria species (Laminariales, Phaeophyceae) with reference to the utility of DNA barcoding. Molecular Phylogenetics and Evolution, 2007, 44(2): 634-648. DOI:10.1016/j.ympev.2007.03.016 |
| [48] |
McDevit DC, Saunders GW. On the utility of DNA barcoding for species differentiation among brown macroalgae (Phaeophyceae) including a novel extraction protocol. Phycological Research, 2009, 57(2): 131-141. DOI:10.1111/j.1440-1835.2009.00530.x |
| [49] |
Lee W, Bae K. Phylogenetic relationship among several genera of Dictyotaceae (Dictyotales, Phaeophyceae) based on 18S rRNA and partial rbcL gene sequences. Marine Biology, 2002, 140(6): 1107-1115. DOI:10.1007/s00227-002-0799-4 |
| [50] |
Sherwood AR, Wang N, Carlile AL et al. The Hawaiian Freshwater Algal Database (HfwADB): A laboratory LIMS and online biodiversity resource. BMC Ecology, 2012, 12: 22. DOI:10.1186/1472-6785-12-22 |
| [51] |
Komárek J, Kastovsky J, Mares J et al. Taxonomic classification of cyanoprokaryotes (cyanobacterial genera) 2014, using a polyphasic approach. Preslia, 2014, 86(4): 295-335. |
| [52] |
Struneck y ' O, Kopejtka K, Goecke F et al. High diversity of thermophilic cyanobacteria in Rupite hot spring identified by microscopy, cultivation, single-cell PCR and amplicon sequencing. Extremophiles, 2019, 23(1): 35-48. DOI:10.1007/s00792-018-1058-z |
| [53] |
Zhang YG, Wang YL, Yang P et al. Morphological diversity and molecular characteristics of bloom forming Dolichospermum species in Lake Zhelin, Jiangxi Province. J Lake Sci, 2020, 32(4): 1076-1087. [张毅鸽, 王一郎, 杨平等. 江西柘林湖水华蓝藻——长孢藻(Dolichospermum)的形态多样性及其分子特征. 湖泊科学, 2020, 32(4): 1076-1087. DOI:10.18307/2020.0416] |
| [54] |
Gu YL, Gao XF, Jiang YG et al. Molecular characterization and toxin quantification of Microcystis panniformis: A microcystin producer in Lake Taihu, China. Journal of Environmental Sciences, 2019, 76: 359-367. DOI:10.1016/j.jes.2018.05.029 |
| [55] |
Zhang JY, Zhu BC, Xu C et al. Strategy of selecting 16S rRNA hypervariable regions for matagenome-phylogenetic marker genes based analysis. Chinese Journal of Applied Ecology, 2015, 26(11): 3545-3553. [张军毅, 朱冰川, 徐超等. 基于分子标记的宏基因组16S rRNA基因高变区选择策略. 应用生态学报, 2015, 26(11): 3545-3553. DOI:10.13287/j.1001-9332.20150812.005] |
| [56] |
Tan WH, Liu Y, Wu ZX et al. cpcBA-IGS as an effective marker to characterize Microcystis wesenbergii (Komárek) in Kondrateva (cyanobacteria). Harmful Algae, 2010, 9(6): 607-612. DOI:10.1016/j.hal.2010.04.011 |
| [57] |
Wang ZJ, Liu Y, Xu Y et al. The divergence of cpcBA-IGS sequences between Dolichospermum and Aphanizomenon (Cyanobacteria) and the molecular detection of Dolichospermum flos-aquae in Taihu Lake, China. Phycologia, 2013, 52(5): 447-454. DOI:10.2216/12-125.1 |
| [58] |
Choi GG, Ahn CY, Oh HM. Phylogenetic relationships of Arthrospira strains inferred from 16S rRNA gene and cpcBA-IGS sequences. ALGAE, 2012, 27(2): 75-82. DOI:10.4490/algae.2012.27.2.075 |
| [59] |
Cellamare M, Duval C, Drelin Y et al. Characterization of phototrophic microorganisms and description of new cyanobacteria isolated from the saline-alkaline crater-lake Dziani Dzaha (Mayotte, Indian Ocean). FEMS Microbiology Ecology, 2018, 94(8): fiy108. DOI:10.1093/femsec/fiy108 |
| [60] |
Nelson JM, Hauser DA, Gudiño JA et al. Complete genomes of symbiotic cyanobacteria clarify the evolution of vanadium-nitrogenase. Genome Biology and Evolution, 2019, 11(7): 1959-1964. DOI:10.1093/gbe/evz137 |
| [61] |
González-Resendiz L, Johansen JR, León-Tejera H et al. A bridge too far in Naming species: A total evidence approach does not support recognition of four species in Desertifilum (Cyanobacteria). Journal of Phycology, 2019, 55(4): 898-911. DOI:10.1111/jpy.12867 |
| [62] |
Pietrasiak N, Osorio-Santos K, Shalygin S et al. When Is A Lineage A Species? A Case Study In Myxacorys gen. nov. (Synechococcales: Cyanobacteria) With The Description of Two New Species From The Americas. Journal of Phycology, 2019, 55(5): 976-996. DOI:10.1111/jpy.12897 |
| [63] |
Casero MC, Velázquez D, Medina-Cobo M et al. Unmasking the identity of toxigenic cyanobacteria driving a multi-toxin bloom by high-throughput sequencing of cyanotoxins genes and 16S rRNA metabarcoding. Science of the Total Environment, 2019, 665: 367-378. DOI:10.1016/j.scitotenv.2019.02.083 |
| [64] |
Liu P, Wei J, Yang K et al. Isolation, molecular identification, and characterization of a unique toxic cyanobacterium Microcystis sp. found in Hunan Province, China. Journal of Toxicology and Environmental Health, Part A, 2018, 81(21): 1142-1149. DOI:10.1080/15287394.2018.1532716 |
| [65] |
le Ai Nguyen V, Tanabe Y, Matsuura H et al. Morphological, biochemical and phylogenetic assessments of water-bloom-forming tropical morphospecies of Microcystis (Chroococcales, Cyanobacteria). Phycological Research, 2012, 60(3): 208-222. DOI:10.1111/j.1440-1835.2012.00650.x |
| [66] |
Komárek J, Komárková J. Review of the European Microcystis-morphospecies (Cyanoprokaryotes) from nature. Czech Phycology Olomouc, 2002, 2: 1-24. |
| [67] |
Deng J, Chen XC, Huang YY et al. Biogeochemical cycling processes associated with cyanobacterial aggregates. Acta Microbiologica Sinica, 2020, 60(9): 1922-1940. [邓杰, 陈雪初, 黄莹莹等. 蓝藻群体颗粒驱动元素地球化学循环研究进展. 微生物学报, 2020, 60(9): 1922-1940.] |
| [68] |
Zhang JY, Guan R, Zhang HJ et al. Complete genome sequence and genomic characterization of Microcystis panniformis FACHB 1757 by third-generation sequencing. Standards in Genomic Sciences, 2016, 11(1): 1-13. DOI:10.1186/s40793-016-0130-5 |
| [69] |
Zhang JY. Metagenomic studies on cyanobacterial blooms in Lake Taihu[Dissertation]. Nanjing: Southeast University, 2018. [张军毅. 太湖蓝藻水华的宏基因组学研究[学位论文]. 南京: 东南大学, 2018. ]
|
| [70] |
Tu J, Chen L, Gao S et al. Obtaining genome sequences of mutualistic bacteria in single Microcystis colonies. International Journal of Molecular Sciences, 2019, 20(20): E5047. DOI:10.3390/ijms20205047 |
| [71] |
Yu P. Establishment of comprehensive diatom index and application in lake eutrophication evaluation[Dissertation]. Shanghai: Shanghai Normal University, 2019. [于潘. 综合硅藻指数(CDI)的建立及在湖泊富营养化评价中的应用[学位论文]. 上海: 上海师范大学, 2019. ]
|
| [72] |
Yang L. The study on the classification and molecular systematics of Neidium in China[Dissertation]. Taiyuan: Shanxi University, 2019. [杨琳. 中国长篦藻属的分类及分子系统学研究[学位论文]. 太原: 山西大学, 2019. ]
|
| [73] |
Chen WW. Study on the morphology, classification and physiology of Skeletonema subsalsum [Dissertation]. Shanghai: Shanghai Normal University, 2019. [陈婉婉. 近盐骨条藻的形态、分类及生理研究[学位论文]. 上海: 上海师范大学, 2019. ]
|
| [74] |
Li Y, Zhu SY, Lundholm N et al. Morphology and molecular phylogeny of Chaetoceros dayaensis sp. nov. (Bacillariophyceae), characterized by two 90° rotations of the resting spore during maturation. Journal of Phycology, 2015, 51(3): 469-479. DOI:10.1111/jpy.12290 |
| [75] |
Alverson AJ. Molecular systematics and the diatom species. Protist, 2008, 159(3): 339-353. DOI:10.1016/j.protis.2008.04.001 |
| [76] |
Stepanek JG, Patrick Kociolek J. Molecular phylogeny of the diatom genera Amphora and Halamphora (Bacillariophyta) with a focus on morphological and ecological evolution. Journal of Phycology, 2019, 55(2): 442-456. DOI:10.1111/jpy.12836 |
| [77] |
Gargas CB, Theriot EC, Ashworth MP et al. Phylogenetic analysis reveals that the 'radial centric' diatom Orthoseira thwaites (Orthoseiraceae, bacillariophyta) is a member of a 'multipolar' diatom lineage. Protist, 2018, 169(6): 803-825. DOI:10.1016/j.protis.2018.08.005 |
| [78] |
Gaonkar CC, Kooistra WHCF, Lange CB et al. Two new species in the Chaetoceros socialis complex (Bacillariophyta): C. sporotruncatus and C. dichatoensis, and characterization of its relatives, C. radicans and C. cinctus. Journal of Phycology, 2017, 53(4): 889-907. DOI:10.1111/jpy.12554 |
| [79] |
Alverson AJ, Jansen RK, Theriot EC. Bridging the Rubicon: Phylogenetic analysis reveals repeated colonizations of marine and fresh waters by thalassiosiroid diatoms. Molecular Phylogenetics and Evolution, 2007, 45(1): 193-210. DOI:10.1016/j.ympev.2007.03.024 |
| [80] |
Bruder K, Medlin LK. Molecular assessment of phylogenetic relationships in selected species/genera in the naviculoid diatoms (Bacillariophyta). I. The genus Placoneis. Nova Hedwigia, 2007, 85(3/4): 331-352. DOI:10.1127/0029-5035/2007/0085-0331 |
| [81] |
Gong SH, Ding YF, Wang Y et al. Advances in DNA barcoding of toxic marine organisms. International Journal of Molecular Sciences, 2018, 19(10): 2931. DOI:10.3390/ijms19102931 |
| [82] |
Sauvage T, Schmidt WE, Suda S et al. A metabarcoding framework for facilitated survey of endolithic phototrophs with tufA. BMC Ecology, 2016, 16: 8. DOI:10.1186/s12898-016-0068-x |
| [83] |
Muggia L, Nelsen MP, Kirika PM et al. Formally described species woefully underrepresent phylogenetic diversity in the common lichen photobiont genus Trebouxia (Trebouxiophyceae, Chlorophyta): An impetus for developing an integrated taxonomy. Molecular Phylogenetics and Evolution, 2020, 149: 106821. DOI:10.1016/j.ympev.2020.106821 |
| [84] |
Martins TP, Ramos V, Hentschke GS et al. The Extremophile Endolithella mcmurdoensis gen. et sp. nov. (Trebouxiophyceae, Chlorellaceae), A New Chlorella-like Endolithic Alga From Antarctica. Journal of Phycology, 2020, 56(1): 208-216. DOI:10.1111/jpy.12940 |
| [85] |
Cremen MCM, Leliaert F, West J et al. Reassessment of the classification of Bryopsidales (Chlorophyta) based on chloroplast phylogenomic analyses. Molecular Phylogenetics and Evolution, 2019, 130: 397-405. DOI:10.1016/j.ympev.2018.09.009 |
| [86] |
Zhu H, Hu YX, Liu F et al. Characterization of the chloroplast genome of Trentepohlia odorata (trentepohliales, chlorophyta), and discussion of its taxonomy. International Journal of Molecular Sciences, 2019, 20(7): 1774. DOI:10.3390/ijms20071774 |
| [87] |
Liu BW, Hu YX, Hu ZY et al. Taxonomic scheme of the order Chaetophorales (Chlorophyceae, Chlorophyta) based on chloroplast genomes. BMC Genomics, 2020, 21(1): 1-12. DOI:10.1186/s12864-020-06845-y |
| [88] |
Lin SJ, Wang L, Zheng LM et al. Current status and future prospect of DNA barcoding in marine biology. Acta Oceanologica Sinica, 2014, 36(12): 1-17. [林森杰, 王路, 郑连明等. 海洋生物DNA条形码研究现状与展望. 海洋学报: 中文版, 2014, 36(12): 1-17.] |
| [89] |
Janouškovec J, Gavelis GS, Burki F et al. Major transitions in dinoflagellate evolution unveiled by phylotranscriptomics. PNAS, 2017, 114(2): E171-E180. DOI:10.1073/pnas.1614842114 |
| [90] |
Annenkova NV, Ahrén D, Logares R et al. Delineating closely related dinoflagellate lineages using phylotranscriptomics. Journal of Phycology, 2018, 54(4): 571-576. DOI:10.1111/jpy.12748 |
| [91] |
Ciugulea I, Nudelman MA, Brosnan S et al. Phylogeny of the euglenoid loricate genera Trachelomonas and Strombomonas (Euglenophyta) inferred from nuclear ssu and lsu rDNA. Journal of Phycology, 2008, 44(2): 406-418. DOI:10.1111/j.1529-8817.2008.00472.x |
| [92] |
Kim JI, Shin W, Triemer RE. Phylogenetic reappraisal of the genus Monomorphina (Euglenophyceae) based on molecular and morphological data. Journal of Phycology, 2013, 49(1): 82-91. DOI:10.1111/jpy.12018 |
| [93] |
Kim JI, Shin W. Molecular phylogeny and cryptic diversity of the genus Phacus (Phacaceae, Euglenophyceae) and the descriptions of seven new species. Journal of Phycology, 2014, 50(5): 948-959. DOI:10.1111/jpy.12227 |
| [94] |
Łukomska-Kowalczyk M, Chaber K, Fells A et al. Molecular and morphological delimitation of species in the group of Lepocinclis ovum-like taxa (Euglenida). Journal of Phycology, 2020, 56(2): 283-299. DOI:10.1111/jpy.12949 |
| [95] |
Gumińska N, Łukomska-Kowalczyk M, Chaber K et al. Evaluation of V2 18S rDNA barcode marker and assessment of sample collection and DNA extraction methods for metabarcoding of autotrophic euglenids. Environmental Microbiology, 2021, 23(6): 2992-3008. DOI:10.1111/1462-2920.15495 |
| [96] |
Karnkowska A, Bennett MS, Triemer RE. Dynamic evolution of inverted repeats in Euglenophyta plastid genomes. Scientific Reports, 2018, 8: 16071. DOI:10.1038/s41598-018-34457-w |
| [97] |
Dabbagh N, Preisfeld A. Intrageneric variability between the chloroplast genomes of Trachelomonas grandis and Trachelomonas volvocina and phylogenomic analysis of phototrophic euglenoids. Journal of Eukaryotic Microbiology, 2018, 65(5): 648-660. DOI:10.1111/jeu.12510 |
| [98] |
Xia S. Taxonomic and ecological studies on freshwater cryptoalgae in China[Dissertation]. Wuhan: Institute of Hydrobiology, Chinese Academy of Sciences, 2013. [夏爽. 中国淡水隐藻类的分类学和生态学研究[学位论文]. 武汉: 中国科学院水生生物研究所, 2013. ]
|
| [99] |
Kim JI, Moore CE, Archibald JM et al. Evolutionary dynamics of cryptophyte plastid genomes. Genome Biology and Evolution, 2017, 9(7): 1859-1872. DOI:10.1093/gbe/evx123 |
| [100] |
Kim JI, Yoon HS, Yi GM et al. Comparative mitochondrial genomics of cryptophyte algae: Gene shuffling and dynamic mobile genetic elements. BMC Genomics, 2018, 19(1): 275. DOI:10.1186/s12864-018-4626-9 |
| [101] |
Beisser D, Graupner N, Bock C et al. Comprehensive transcriptome analysis provides new insights into nutritional strategies and phylogenetic relationships of chrysophytes. PeerJ, 2017, 5: e2832. DOI:10.7717/peerj.2832 |
| [102] |
Bråte J, Fuss J, Jakobsen KS et al. Draft genome assembly and transcriptome sequencing of the golden algae Hydrurus foetidus (Chrysophyceae). F1000Research, 2019, 8: 401. DOI:10.12688/f1000research.16734.1 |
| [103] |
Rybalka N, Andersen RA, Kostikov I et al. Testing for endemism, genotypic diversity and species concepts in Antarctic terrestrial microalgae of the Tribonemataceae (Stramenopiles, Xanthophyceae). Environmental Microbiology, 2009, 11(3): 554-565. DOI:10.1111/j.1462-2920.2008.01787.x |
| [104] |
Maistro S, Broady PA, Andreoli C et al. Phylogeny and taxonomy of xanthophyceae (Stramenopiles, Chromalveolata). Protist, 2009, 160(3): 412-426. DOI:10.1016/j.protis.2009.02.002 |
| [105] |
Zuccarello GC, West JA, Kikuchi N. Phylogenetic relationships within the stylonematales (Stylonematophyceae, Rhodophyta): Biogeographic patterns do not apply to Stylonema alsidii. Journal of Phycology, 2008, 44(2): 384-393. DOI:10.1111/j.1529-8817.2008.00467.x |
| [106] |
Lin SM, De Clerck O, Leliaert F et al. Systematics and biogeography of the red algal genus Yonagunia (Halymeniaceae, Rhodophyta) from the indo-Pacific including the description of two new species from Taiwan. Journal of Phycology, 2020, 56(6): 1542-1556. DOI:10.1111/jpy.13055 |
| [107] |
Gomes FP, Maggs CA, Barros-Barreto MBB. Integrative approach reveals four new cryptic species in the genus Gayliella (Ceramiaceae, Rhodophyta). Journal of Phycology, 2020, 56(2): 437-457. DOI:10.1111/jpy.12961 |
| [108] |
Mao YX, Wu FF, Du GY. DNA barcoding techniques in the systematic study of rhodophyta. Periodical of Ocean University of China, 2014, 44(8): 48-53. [茅云翔, 吴菲菲, 杜国英. 红藻DNA条形码分析技术研究进展. 中国海洋大学学报: 自然科学版, 2014, 44(8): 48-53.] |
| [109] |
Xue HF. Classification research of maroalgae of brown algae and green algae from the intertidal zone of Shandong peninsula based on DNA barcoding technology[Dissertation]. Qingdao: Ocean University of China, 2014. [薛红凡. 基于DNA条形码技术的山东半岛潮间带褐藻和绿藻分类研究[学位论文]. 青岛: 中国海洋大学, 2014. ]
|
| [110] |
Montecinos AE, Couceiro L, Peters AF et al. Species delimitation and phylogeographic analyses in the Ectocarpus subgroup siliculosi (Ectocarpales, Phaeophyceae). Journal of Phycology, 2017, 53(1): 17-31. DOI:10.1111/jpy.12452 |
| [111] |
Ng PK, Chiou YS, Liu LC et al. Phylogeography and genetic connectivity of the marine macro-alga Sargassum ilicifolium (Phaeophyceae, Ochrophyta) in the northwestern Pacific. Journal of Phycology, 2019, 55(1): 7-24. DOI:10.1111/jpy.12806 |
| [112] |
Bruno de Sousa C, Cox CJ, Brito L et al. Improved phylogeny of brown algae Cystoseira (Fucales) from the Atlantic-Mediterranean region based on mitochondrial sequences. PLoS One, 2019, 14(1): e0210143. DOI:10.1371/journal.pone.0210143 |
| [113] |
Ortega A, Geraldi NR, Díaz-Rúa R et al. A DNA mini-barcode for marine macrophytes. Molecular Ecology Resources, 2020, 20(4): 920-935. DOI:10.1111/1755-0998.13164 |
| [114] |
Hatfield RG, Batista FM, Bean TP et al. The application of nanopore sequencing technology to the study of dinoflagellates: A proof of concept study for rapid sequence-based discrimination of potentially harmful algae. Frontiers in Microbiology, 2020, 11: 844. DOI:10.3389/fmicb.2020.00844 |
| [115] |
Douchi D, Mosey M, Astling DP et al. Nuclear and chloroplast genome engineering of a productive non-model alga Desmodesmus armatus: Insights into unusual and selective acquisition mechanisms for foreign DNA. Algal Research, 2021, 53: 102152. DOI:10.1016/j.algal.2020.102152 |
| [116] |
Sauriau PG, Dartois M, Becquet V et al. Multiple genetic marker analysis challenges the introduction history of Ulva australis (Ulvales, Chlorophyta) on French coasts. European Journal of Phycology, 2021, 1-13. DOI:10.1080/09670262.2021.1876249 |
| [117] |
Zhan SH, Shih CC, Liu SL. Reappraising plastid markers of the red algae for phylogenetic community ecology in the genomic era. Ecology and Evolution, 2020, 10(3): 1299-1310. DOI:10.1002/ece3.5984 |
| [118] |
Prasanthi N, Prasannakumar C, Annadurai D et al. Identifying seaweeds species of Chlorophyta, Phaeophyta and Rhodophyta using DNA barcodes. bioRxiv, 2020. DOI:10.1101/2020.08.30.274456.DOI:10.1101/2020.08.30.274456 |
| [119] |
Schaap A, Rohrlack T, Bellouard Y. Lab on a chip technologies for algae detection: A review. Journal of Biophotonics, 2012, 5(8/9): 661-672. DOI:10.1002/jbio.201200051 |
| [120] |
Galluzzi L, Cegna A, Casabianca S et al. Development of an oligonucleotide microarray for the detection and monitoring of marine dinoflagellates. Journal of Microbiological Methods, 2011, 84(2): 234-242. DOI:10.1016/j.mimet.2010.11.024 |
| [121] |
Shen BX, Piskunen P, Nummelin S et al. Advanced DNA nanopore technologies. ACS Applied Bio Materials, 2020, 3(9): 5606-5619. DOI:10.1021/acsabm.0c00879 |
| [122] |
Slatko BE, Gardner AF, Ausubel FM. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology, 2018, 122(1): e59. DOI:10.1002/cpmb.59 |
| [123] |
Komárek J. Several problems of the polyphasic approach in the modern cyanobacterial system. Hydrobiologia, 2018, 811(1): 7-17. DOI:10.1007/s10750-017-3379-9 |
| [124] |
Komárek J. A polyphasic approach for the taxonomy of cyanobacteria: Principles and applications. European Journal of Phycology, 2016, 51(3): 346-353. DOI:10.1080/09670262.2016.1163738 |
| [125] |
Cegłowska M, Toruńska-Sitarz A, Stoń-Egiert J et al. Characteristics of cyanobacterium Pseudanabaena galeata CCNP1313 from the Baltic Sea. Algal Research, 2020, 47: 101861. DOI:10.1016/j.algal.2020.101861 |
| [126] |
Martins MD, Machado-De-lima NM, Branco LHZ. Polyphasic approach using multilocus analyses supports the establishment of the new aerophytic cyanobacterial genus Pycnacronema (Coleofasciculaceae, Oscillatoriales). Journal of Phycology, 2019, 55(1): 146-159. DOI:10.1111/jpy.12805 |
| [127] |
Saber AA, Fučíková K, McManus HA et al. Novel green algal isolates from the Egyptian hyper-arid desert oases: A polyphasic approach with a description of Pharao desertorum gen. et sp. nov. (Chlorophyceae, Chlorophyta). Journal of Phycology, 2018, 54(3): 342-357. DOI:10.1111/jpy.12645 |
| [128] |
Procházková L, Remias D, Holzinger A et al. Ecophysiological and morphological comparison of two populations of Chlainomonas sp. (Chlorophyta) causing red snow on ice-covered lakes in the High Tatras and Austrian Alps. European Journal of Phycology, 2018, 53(2): 230-243. DOI:10.1080/09670262.2018.1426789 |
| [129] |
Kollár MJ. Polyphasic approach to the study of diatom diversity[Dissertation]. Olomouc: Palack y ' University Olomouc Faculty of Science Department of Botany, 2020.
|
| [130] |
Kollár J, Pinseel E, Vanormelingen P et al. A polyphasic approach to the delimitation of diatom species: A case study for the genus Pinnularia (Bacillariophyta). Journal of Phycology, 2019, 55(2): 365-379. DOI:10.1111/jpy.12825 |
| [131] |
Delmail D, Labrousse P, Crassous P et al. Prorocentrum rivalis sp. nov. (Dinophyceae) and its phylogenetic affinities inferred from analysis of a mixed morphological and LSU rRNA data set. Biologia, 2011, 66(3): 418-424. DOI:10.2478/s11756-011-0029-y |
| [132] |
Soares LP, Guimarães S, Fujii MT et al. Rhodachlya westii sp. nov. (Rhodachlyales, Rhodophyta), a new species from Brazil, revealed by an integrative taxonomic approach. Phycologia, 2020, 59(4): 346-354. DOI:10.1080/00318884.2020.1763771 |
 2021, Vol. 33
2021, Vol. 33