(2: 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008)
(3: 北京化工大学生命科学与技术学院, 北京 100029)
(2: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
(3: College of Life Science and Technology, Beijing University of Chemical Technology, Beijing 100029, P. R. China)
近几十年来,全球不同类型水体(近海、河口、湖库等)均面临着严重富营养化威胁,蓝藻水华频发严重影响着全球公共卫生安全和水生态系统健康[1]. 目前,全球范围内大约有60 % 的湖泊处于不同程度富营养化状态[2]. 在我国,2018年监测的不同地区107个淡水湖泊中,富营养、中营养和贫营养湖泊的比例分别为29 %、61.7 % 和9.3 % [3]. 水体中部分蓝藻(例如微囊藻、鱼腥藻、念珠藻、节球藻等)会生成有害次生代谢物,向水体释放微囊藻毒素(microcystin)、节球藻毒素(nodularin)、柱胞藻毒素(cylindrospermopsin)等大分子有毒物质. 这些毒素可以在水生生物体内累积,并随着水生食物链迁移,最终作用于人体肝脏、皮肤、神经系统等靶器官,从而严重威胁人类健康[4-5]. 考虑到蓝藻水华对水生态影响和人群健康威胁,如何降低水体中蓝藻的丰度、控制蓝藻水华发生频率一直是水生态领域的研究焦点.
噬藻体(cyanophage)是一类以蓝藻为宿主的病毒,广泛分布在全球不同的水体中(湖泊、海洋、鱼塘、河流等)[6]. 现阶段,已经从淡水和海水中分离出数百株裂解微囊藻、聚球藻、鱼腥藻、节球藻等的噬藻体. 噬藻体在水环境中适应能力强,在北极等极端环境中也检测到了不同种类的噬藻体[7]. 微囊藻属(铜绿微囊藻、水华微囊藻)是导致蓝藻水华暴发的主要浮游生物类型,但其生长、繁殖和凋亡会受到水体中噬藻体的显著影响[8]. 现有研究已详细报道了能够裂解8种微囊藻的噬藻体信息[9]. 自然水体中噬藻体浓度较高,淡水中能够感染铜绿微囊藻的噬藻体浓度约为5.6×104 pfu/mL[10],水体中每天有超过51 % 的蓝藻被噬藻体裂解死亡[11];海水中侵染聚球藻、原绿球藻的噬藻体浓度为104~105 pfu/mL[12],导致聚球藻的日致死率可达到5 % ~14 %,降低海洋中78 % 的初级生产力[12].
噬藻体作为水体中浮游病毒的重要组成部分,其在控制蓝藻种群密度、减少蓝藻水华发生频率等方面具有重要的研究意义和应用价值. 浮游动物捕食和病毒裂解是控制自然水体中藻类消长的主要因素,且有研究发现捕食作用和病毒裂解对蓝藻的致死率相当[13-14]. 噬藻体在适宜的环境中可快速侵染蓝藻、导致宿主藻细胞裂解死亡,改变水体中优势藻种、降低宿主细胞丰度,最终改变水体生物群落结构[15-18]. 噬藻体侵染蓝藻后向水体中释放大量的碳、氮、磷等营养物质,促进了水体有机物质循环,减少其向更高营养水平的转移,增加水体的初级生产力. 噬藻体裂解蓝藻释放颗粒性有机碳(POC)到水体中,在细菌作用下转化为溶解性有机碳(DOC),增加水体中初级生产力,可重新被浮游生物利用[19]. 噬藻体与蓝藻相互作用过程对水环境中生物化学循环的相关研究仍较为薄弱;但噬藻体感染、裂解蓝藻过程是否会造成藻毒素在短时间内大量释放到自然水体的水生态风险仍需要进行评估.
本文从噬藻体基本形态入手,回顾了噬藻体发现史,总结其与宿主藻的侵染机制与过程,详细介绍噬藻体对蓝藻种群密度调节作用,探讨应用噬藻体裂解蓝藻对水体中营养物质循环、浮游生物群落结构变化、产毒蓝藻的藻毒素释放的潜在影响,这可以为后续噬藻体在富营养化水体治理中的潜在应用提供一定的理论和实践依据.
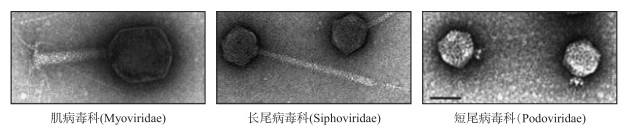
1 噬藻体的发现及侵染机制 1.1 噬藻体发现及分类噬藻体是一类能够特异性感染蓝藻(如微囊藻、聚球藻、念珠藻等)的病毒. 根据噬藻体形态差异(主要是其尾部特征),国际病毒学分类委员会细菌病毒分会最初将噬藻体主要分为肌病毒科(Moyviidrae)、短尾病毒科(Podoviirdae)、长尾病毒科(Siphovirdae)3大类[20-21],3种类型噬藻体的形态特征如图 1所示. 但近年来国际病毒学分类委员会对细菌病毒进行了更细致的划分,主要分为肌病毒科、短尾病毒科、长尾病毒科、双尾噬菌体科、球状噬菌体科、原体噬菌体科等十几类[22]. 常见的T4型噬藻体属于肌病毒科,T7型噬藻体属于短尾病毒科,已报道的噬藻体大多为肌病毒科,长尾病毒科和短尾病毒科则较少见. 根据噬藻体侵染的宿主藻的形态差异,可分为以侵染颤藻、鱼腥藻、念珠藻等为主的丝状蓝藻噬藻体,和以侵染微囊藻、聚球藻、组囊藻为主的单细胞蓝藻噬藻体,这两类噬藻体侵染宿主细胞机制存在一定的差异.
|
图 1 透射电子显微镜下3类噬藻体典型形态特征[23] Fig.1 Typical morphology characteristics of three types of cyanophage under transmission microscope |
在1963年,Safferman和Morris[24]分离出第一株能够同时感染鞘丝藻(Lyngbya)、织线藻(Plectonema)、席藻(Phormidium)的噬藻体,并将其命名为“LPP”噬藻体. 在之后60年的研究中,陆续报道了从全球不同水体中分离出能够感染鱼腥藻、微囊藻、节球藻、聚球藻等的噬藻体(表 1). 在1989和1990年,Bergh、Suttle、Proctor等[7, 12, 20]分别在Nature上发表论文,首次指出噬藻体在海洋中广泛存在,并阐述了其在海洋生态系统中具有重要的生物功能和环境意义. 在1992年9月,Wilson等[25]从3个不同海洋中分离出5株感染聚球藻的噬藻体,根据形态被鉴定为肌病毒科和长尾病毒科. 在1993年,Waterbury等从近海和远洋水体中分离了75株能够感染聚球藻的噬藻体[26],这些噬藻体表现出显著的形态差异,包括肌病毒科、长尾病毒科和短尾病毒科3个类别,且大多数分离物种为带有收缩尾的肌病毒. 在2002年,我国科学家赵以军等[27]在武汉市一处富营养化池塘中分离能够特异性感染织线藻和席藻的淡水噬藻体PP,该噬藻体为在我国分离的第一株淡水噬藻体. 在2006年,Jenkins等[28]在波罗的海分离出17株能够裂解节球藻的噬藻体,并通过电镜观察噬藻体形态,主要分为肌病毒科和长尾病毒科. 在2008年,Deng等[29]在瑞士苏黎世湖和英国科茨沃尔德水上公园分离出35株可成功侵染微囊藻、鱼腥藻和浮丝藻的噬藻体. 在后续的研究中,诸多学者分别从海洋、湖泊、池塘、沼泽等水体中分离出多株噬藻体,分析其与宿主的关系并探究了其侵染机制[30]. 截至2019年,已有文献记载了318株聚球藻肌病毒科噬藻体,9株聚球藻短尾噬藻体和13株聚球藻长尾噬藻体,并记录了完整的基因组序列. 迄今为止,已报道了9株侵染微囊藻的噬藻体的相关信息,但仅对4株噬藻体(MaMV-DC、Ma-LMM01、Mic1和PhiMa05)进行了完整的基因组测定,表 1给出了全球已经分离、鉴定完的部分淡水和海水噬藻体.
| 表 1 现有研究已报道的部分噬藻体基本特征 Tab. 1 Basic information on some reported cyanophage in existing studies |
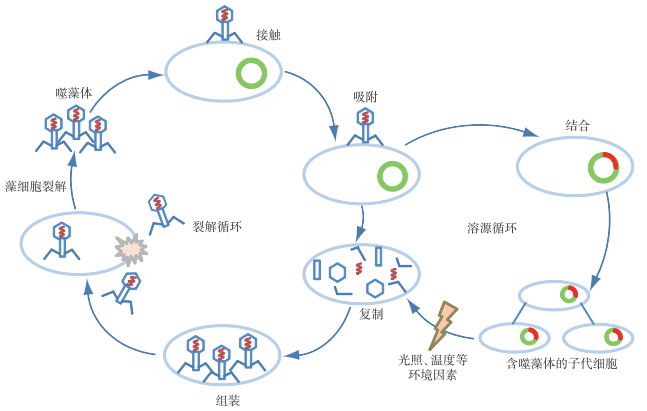
噬藻体的增殖机制由基因表达控制,其与宿主细胞的侵染作用主要分为裂解性循环和溶源性循环[60-61]. 裂解性循环通常被认为是噬藻体的遗传物质在宿主细胞内复制并合成蛋白质,宿主细胞生理功能被破坏,裂解并释放子代噬藻体. 溶源性循环是指噬藻体进入蓝藻,其DNA结合在宿主藻染色体,并随其一起复制且长期共存. 在外界环境因素刺激下如光照、温度及其他生物因素,噬藻体从溶源性转化为裂解性完成增殖作用并使宿主藻裂解死亡. 噬藻体裂解性循环和溶源性循环如图 2所示. 本部分主要介绍噬藻体的裂解性增殖机制及环境因素对该过程的影响.
1.2.1 吸附噬藻体与宿主细胞充分接触,吸附是感染宿主的关键环节. 水体中宿主细胞浓度和噬藻体浓度影响二者接触率,进而影响吸附效果. 噬藻体对宿主细胞的吸附率还会随光照强度、水体中二价阳离子浓度、Na+浓度等因素而改变[63-64]. 现有研究发现,裂解原绿球藻的噬藻体P-HM2、P-SSM2在光照条件下的吸附率比黑暗条件下高,光暗循环对噬藻体吸附、增殖具有显著的影响[65]. 水体中Na+对噬藻体吸附宿主细胞具有促进作用,导致这种现象的主要原因可能是光照和Na+均会使藻细胞表面容易发生电中和、促进噬藻体吸附[66].
1.2.2 增殖噬藻体吸附到宿主细胞表面,将DNA注入宿主细胞后,利用宿主细胞物质和能量进行复制、转录和翻译,合成自身需要的DNA和蛋白质. 但噬藻体对单细胞蓝藻和丝状蓝藻的感染机制不同[31],感染丝状蓝藻的噬藻体通常可以在侵染成功后立即终止宿主光合作用,后续噬藻体增殖所需的能量主要通过氧化磷酸化获得[67]. 然而,在感染单细胞蓝藻过程中,被感染的宿主细胞的光合速率可以一直保持,直到宿主细胞完全裂解[68].
光照强度会影响宿主细胞的光合作用,进而影响细胞内ATP生成,间接影响噬藻体增殖. Puxty等[69]探究了光照强度对噬藻体增殖的影响,在黑暗条件下噬藻体潜伏期会延长,释放量减少,甚至会停止其在宿主细胞内的复制. 由于噬藻体进入宿主细胞后的生命活动依赖于宿主的生长状态,因此水体中氮磷等营养物质浓度均会影响噬藻体增殖;氮磷浓度充足时促进藻细胞生长、加速噬藻体增殖. CO2浓度和温度升高会加快宿主裂解和噬藻体释放,Cheng等[70]发现在高浓度CO2条件下,噬藻体PP感染鲍氏织线藻的潜伏期缩短但子代噬藻体释放量减少,而温度升高导致噬藻体PP潜伏期缩短,释放量增加. 温度和CO2浓度的变化会影响宿主细胞内部分酶的活性,提高蛋白质、核酸等大分子合成效率,加快宿主细胞裂解[71].
1.2.3 裂解和释放宿主细胞裂解和噬藻体释放速率与宿主生长状态、细胞分裂以及宿主细胞内酶活性等密切相关. 处于对数生长期的宿主藻极易被噬藻体裂解,并释放新合成的子代噬藻体,而噬藻体侵入处于稳定期宿主细胞则不能导致宿主的快速裂解,当其恢复生长后才能释放子代噬藻体[72]. 噬菌体F116L侵染铜绿假单胞菌(Pseudomonas aeruginosa)的研究证明,宿主细胞处于饥饿状态导致噬菌体潜伏期显著增加,且子代噬藻体释放量减少[73-74]. 宿主细胞与噬藻体存在的相互作用机制,既能使噬藻体自身的种群得以维持,又能够对宿主的数量进行有效控制.
1.3 噬藻体与宿主间的关系噬藻体在自然水体中广泛存在,尤其是在富营养化水体中浓度更高. 野外监测结果显示,季节变化对噬藻体丰度和蓝藻种群密度具有显著的影响. 夏秋季水温较高时,水体中蓝藻种类丰富且浓度较高,而此时水体中噬藻体数量也达到峰值,二者保持动态平衡过程[75]. Sandaa等[76]发现挪威沿海水域的聚球藻密度和噬藻体浓度变化趋势一致,均在水温最高的8月下旬和9月其种群数量达到最大值. 聚球藻爆发性繁殖导致的蓝藻水华发生在噬藻体数量达到峰值之前;随后,水体中噬藻体的丰度和多样性均增加,这表明噬藻体对水体中聚球藻种群的季节性演替具有重要调节作用. 然而在自然界中尽管噬藻体浓度较高,但蓝藻水华却没有得到较好的控制,可能是由于噬藻体对易感的宿主细胞具有致死作用,但长期接触导致藻细胞对噬藻体产生抗性. 研究发现,海洋中的噬藻体-原绿球藻的相互作用,使得一些宿主藻由易感细胞向抗性细胞转化,对噬藻体产生抗性的原绿球藻生长速率不断增加,抗性宿主藻的快速生长使其在环境中占主导地位,并实现与噬藻体长期共存[77-79].
噬藻体与宿主藻间存在水平基因转移,早期研究发现在噬藻体基因组中检测到蓝藻细胞中的一些重要基因,如光合基因(psbA、psbD)、辅助代谢基因(auxiliary metabolic genes, AMGs)等. 在武汉东湖分离的噬藻体PaV-LD和云南滇池分离的以微囊藻为宿主的噬藻体MaMV-DC的基因组中均发现了nblA基因,其与蓝藻NblA基因具有相似的生物学功能,均可降解宿主的藻胆蛋白,这一现象证实了噬藻体和宿主细胞间存在水平基因转移[80]. 噬藻体基因组中携带蓝藻的光合基因及代谢基因,使其在侵染宿主藻后,通过增加蓝藻的光合作用提高宿主代谢速率为噬藻体的复制和衣壳蛋白的合成提供能量. 噬藻体诱导的水平基因转移不仅存在病毒与宿主之间,还会导致不同蓝藻间的基因转移、促进蓝藻与噬藻体协同进化[81-83]. 因此,噬藻体通过抗性选择、水平基因转移和操纵细菌代谢来影响宿主,从而维持或增强宿主遗传多样性,并实现长期共存[84-85].
2 噬藻体对环境中蓝藻种群密度的调节作用水体中噬藻体种类较多、分布广泛,对藻细胞具有特异性致死作用,且对部分微囊藻、聚球藻等具有较高的日致死率. 因此,在理论上,噬藻体的存在会导致蓝藻丰度降低、减少蓝藻水华发生频率. 实验室研究已经充分证实,噬藻体可以快速裂解宿主藻细胞、调控培养体系的蓝藻丰度. 近年来,一些学者在红海[86]、夏威夷海域[87]、切萨皮克湾[88]等自然水体中发现较高浓度的噬藻体,其对水体中宿主藻密度具有一定的调控作用, 影响原有蓝藻群落构成. 此部分内容总结了近年来室内实验报道的噬藻体对蓝藻裂解效果,以及噬藻体在湖泊、海洋等水体中对蓝藻丰度的调控规律.
2.1 实验室条件下噬藻体对蓝藻的裂解效果近十年来,研究人员开始关注噬藻体对蓝藻的侵染效果及其在调节种群密度中的作用. 利用从自然水体中分离的噬藻体,一些学者在室内环境中进行侵染试验,探究噬藻体对宿主藻丰度的影响规律. Wang等[89]探究从滇池分离的肌病毒科噬藻体MaMV-DC对8种微囊藻的裂解效果,噬藻体与宿主藻以1 ∶1比例混合培养7 d后,部分藻液出现了黄化. 噬藻斑实验证明,噬藻体MaMV-DC能够裂解铜绿微囊藻FACHB524、水华微囊藻TF09、铜绿微囊藻TA09、惠氏微囊藻DW09 4种微囊藻. 与未添加噬藻体的对照组相比,4种微囊藻浓度分别降低了75 %、61.5 %、71.4 % 和61.2 %;而培养体系中噬藻体浓度由7.7×106 pfu/mL分别增加到3.1×108、1.41×108、1.57×108和1.08×108 pfu/mL. 噬藻体MaMV-DC可以有效抑制易感微囊藻生长、控制宿主藻种群数量. 2005年Tucker等[10]在实验室内探究了从澳大利亚巴罗翁湖分离的噬藻体Ma-LBP对产毒铜绿微囊藻密度的影响. 培养初期噬藻体与宿主藻比例分别为0.1 ∶1、1 ∶1、10 ∶1和100 ∶1,在培养第6天添加噬藻体的实验组铜绿微囊藻密度显著降低. 噬藻体-宿主藻比例为100 ∶1的实验组,宿主藻的丰度降低了95 %;而噬藻体的浓度增加了9.2×105 pfu/mL. 然而,在含有存活藻细胞的培养体系重新添加噬藻体,宿主细胞在培养3周后种群密度快速增加,这可能由于宿主藻细胞对噬藻体产生了一定的抗性.
噬藻体PP是赵以军等分离出裂解织线藻和席藻的淡水噬藻体. 2013年Zhou等[90]在实验室内分析了噬藻体PP对23种蓝藻侵染效果. 将0.5 mL浓度为108 pfu/mL的噬藻体分别加入处于对数生长期的23株藻液中进行初次筛选,发现15株藻液颜色由绿转化为不同程度的黄. 在筛选出的15株蓝藻中添加噬藻体进行二次筛选,鲍氏织线藻(IU594、FACHB-402、FACHB-246、FACHB-240)、席藻(FACHB-238、FACHB-239、FACHB-161)7株不同类型宿主藻在培养12 h内出现裂解、黄化,说明噬藻体PP宿主范围较广,可裂解不同种类蓝藻宿主. 在2019年,You等[52]在中国黄海分离出一株新型短尾病毒科噬藻体S-B64,根据一步生长曲线发现噬藻体的潜伏期约为3 h,宿主藻在与噬藻体接触8 h后裂解,每个藻细胞约释放23个子代病毒. 为了确定噬藻体S-B64宿主范围,用其侵染33株聚球藻,发现该噬藻体仅对聚球藻(WH8102、WH7803、LTW Red 1、MW01)具有裂解效果,实验结果说明噬藻体S-B64的宿主范围较窄,对聚球藻的不同株系侵染具有一定的特异性. 噬藻体对单一藻细胞或几种藻细胞的特异性侵染,导致其在复杂水环境中对蓝藻丰度调控具有一定的局限性. 而在几种藻共存的混合体系中,噬藻体侵染易感宿主使其种群密度降低,改变群落中优势藻种,可能促进其他蓝藻的快速生长. Coloma等[48]在固氮节球藻和聚球藻培养体系中添加从波罗的海分离的噬藻体,实验结果表明该噬藻体可以快速裂解节球藻,使其种群密度显著降低. 但节球藻裂解所释放的氮被聚球藻吸收利用、导致聚球藻细胞密度增加了7倍. 廖明军[91]在室内模拟试验中发现,噬藻体裂解宿主织线藻后,随着天然水体带入的铜绿微囊藻快速生长、生物量出现显著增加,并成为新的优势种. 由于培养体系中宿主细胞的裂解导致营养物组成改变,使得不同培养时期细菌的优势种群也发生变化. 宿主细胞裂解提供再生的营养物质可能刺激了群落中产生竞争机制的藻类生长,改变培养体系内的优势种群并发生群落演替.
噬藻体不仅可以通过裂解宿主细胞调节蓝藻种群密度,还能够改变水体中蓝藻与浮游动物的相对数量. Šulčius等[92]通过室内实验评估了大型溞捕食和噬藻体感染的协同作用对水华束丝藻种群变化的影响. 研究发现,在噬藻体侵染束丝藻的第5~7天内,藻细胞大量裂解,菌丝长度和数量下降,总细胞丰度降低. 噬藻体侵染束丝藻,诱导其种群结构向较短丝状体方向进化,促进大型溞对束丝藻的捕食,使大型溞与水华束丝藻之间由非捕食作用转变为捕食作用. 因此,噬藻体的裂解作用对水体大型溞的存活和摄食具有协同效应.
噬藻体与宿主细胞的长期共存使宿主藻突变体快速出现,突变藻通过改变细胞膜结构阻止噬藻体的吸附[93],这使得噬藻体-蓝藻相互作用关系的复杂性和不可预测性增加. 已有研究证据表明,噬藻体vB_NpeS-2AV2可以诱导节球藻种群变化,导致其从易感细胞为主的种群向抗性细胞为主的种群进化[48]. 噬藻体Vb-AphaS-CL131与束丝藻长期接触,使束丝藻对噬藻体的敏感性降低,耐病毒基因和易感基因分布改变、导致抗感染菌丝出现,使宿主藻在几天内恢复到初始种群丰度[92]. Avrani等[94]发现海洋原绿球藻和噬藻体长期接触会产生抗性,在实验室内将4种初始浓度为106 cells/mL的原绿球藻和噬藻体共培养40个月,分离出抗性原绿球藻. 原绿球藻向提高生长速度和增大抗性范围的方向进化,导致原绿球藻对噬藻体产生更大的抗性,最终其可以与多种噬藻体长期共存.
目前,室内实验已证实多株噬藻体对蓝藻具有裂解效果,噬藻体对宿主藻种群密度具有显著的调节作用,且可改变水体中其他浮游生物群落. 但噬藻体对蓝藻侵染具有特异性,多数噬藻体仅能裂解一种或几种同一属性藻细胞;同时,噬藻体-宿主藻存在相互作用,导致宿主细胞易对噬藻体产生抗性. 因此,在实验室单一体系中,噬藻体裂解蓝藻往往效果较好,但在复杂的实际环境中的使用效果仍需要进一步验证.
2.2 自然水体噬藻体对蓝藻丰度的影响噬藻体作为水生态系统中重要的微观生物调控因子,其在自然水体浮游生物群落演替中发挥着重要的作用. 但由于水体中噬藻体分离、鉴定极为困难,同时涉及到跨学科领域的背景知识,现有研究很少把噬藻体作为水生态系统中的重要组成来进行考虑,而噬藻体在实际水体中存在的具体作用也常被忽略. 在Web of Science中,以“cyanophage”、“lysis”、“cyanobacterial populations”为关键词进行检索后表明,迄今为止,在自然环境中研究噬藻体的实际应用案例仅为10个,由此可见,关于自然水体噬藻体与蓝藻之间相互作用规律的研究仍然极少.
Hewson等[87]在夏威夷附近的海域监测到能裂解束毛藻的噬藻体浓度为3.96×103 pfu/mL,其对束丝藻的日致死率为0.3 % ~6.5 %,可有效降低海水中束毛藻种群密度. Coello-Camba等[86]在贫营养的红海进行围栏试验,通过宏基因组学方法探究海水噬藻体丰度变化及其对聚球藻密度的影响. 在实验期间,噬藻体基因序列的相对丰度从总病毒的12.6 % 增加到40 %;在聚球藻暴发几天后,海水中噬藻体的数量大量增加、聚球藻密度快速降低. 噬藻体侵染聚球藻后,降低了其在浮游群落中竞争能力、短时间内改变水体优势藻种. Wang等[88]研究发现,美国切萨皮克湾的聚球藻的丰度与噬藻体浓度变化呈现出强烈的季节模式,噬藻体丰度与聚球藻密度具有强烈相关性. 夏季蓝藻水华期间,噬藻体与宿主藻的接触率较高,日致死率达到1 % ~10 %,但冬季日接触率低于0.1 %,这表明环境因子对噬藻体的侵染效果具有重要影响. 切萨皮克湾聚球藻丰度和噬藻体浓度之间的密切关系表明,噬藻体在调节聚球藻丰度和生产力方面可能发挥重要的作用.
铜绿微囊藻广泛分布在湖泊、池塘等水体中,是最常见蓝藻水华藻种. 目前,已分离出的多株感染铜绿微囊藻噬藻体,并在室内实验中证实其对微囊藻丰度的调节作用. 有研究证明,自然水体中铜绿微囊藻的密度变化与噬藻体作用相关. Manage等[95]在1997年3月到1998年1月监测到日本一个超富营养化池塘中铜绿微囊藻和噬藻体密度的季节变化特征. 铜绿微囊藻是池塘水华优势藻种,占浮游植物总量的83 % 以上. 当池塘中噬藻体浓度较高时,铜绿微囊藻密度急剧下降;而铜绿微囊藻在冬季密度较低,池塘中则检测不到噬藻体. Yoshida等[96]在日本米卡塔湖首次证实,淡水噬藻体群落季节性动态变化与宿主铜绿微囊藻丰度呈现显著负相关;当宿主藻的细胞数量降低时,噬藻体浓度明显增加. 蓝藻细胞丰度同步监测结果显示,噬藻体丰度变化会影响产生微囊藻毒素和不产生微囊藻毒素的蓝藻种群相对结构变化,产毒藻种在所有微囊藻生物量中由47.1 % 下降到0.5 %. 在4--7月,米卡塔湖中噬藻体浓度增加,微囊藻在所有蓝藻生物量中的比例从0.3 % 下降到0.07 %,这显示了噬藻体对蓝藻群落构成具有较强调控能力. 在自然水体中,噬藻体对微囊藻丰度具有一定的调节作用,可降低微囊藻相对种群密度;研究发现通常在蓝藻水华发生后噬藻体密度快速增加,可减缓蓝藻水华持续时间[86],但并不能阻止水华的发生,这可能会是应用噬藻体治理蓝藻水华面临的一个问题.
水体中蓝藻被噬藻体裂解、影响原有食物链结构,导致各营养级均会发生变化. 例如,在肯尼亚纳库鲁湖,火烈鸟主要以湖泊中藻类为食. 当湖泊中占有优势的节旋藻被噬藻体感染大规模死亡时,节旋藻种群密度降低导致火烈鸟数量急剧减少. 这表明,噬藻体可以通过裂解水生食物链初级消费者(蓝藻)自下而上影响不同营养级,从而影响较高营养等级消费者的种群数量[97]. 噬藻体在调节水体蓝藻丰度、改变浮游群落结构上发挥着重要的作用,但噬藻体侵染导致的蓝藻裂解对水生食物网产生的进一步影响仍需要探究.
3 噬藻体对水体中物质循环的影响噬藻体裂解宿主后释放大量营养物质,促进了营养元素在水体中生物地球化学循环. 水体中蓝藻自然死亡的主要原因为浮游动物的捕食和噬藻体的裂解[13-14]. 尽管有研究认为,捕食作用和病毒裂解对蓝藻致死效力相当,但2种致死作用对水体中营养元素和有机物质的影响却不相同. 捕食作用使营养物质随着水生食物链向更高营养级转移,释放的有机物质主要为颗粒态;而噬藻体裂解释放出溶解态有机物及和易被微生物利用的颗粒态物质,从而有利于促进水体中营养物质循环利用.
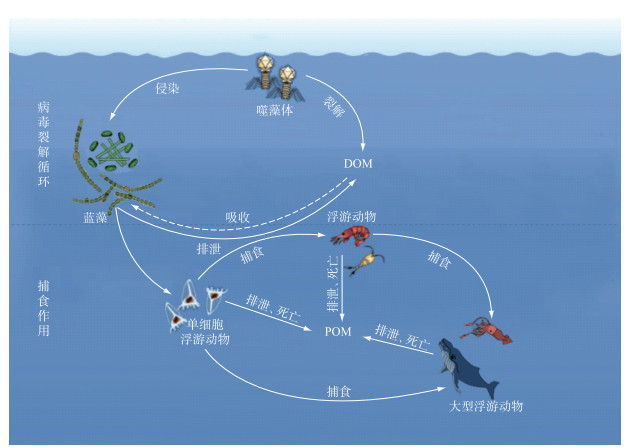
3.1 噬藻体对水体中碳循环过程的影响水体中病毒的裂解过程每年约向海洋释放30亿吨碳[75]. 海洋中溶解性有机物主要由病毒裂解、细胞分泌以及细胞机械破解3种机制释放,其中病毒裂解是溶解性有机质(DOM)最主要来源[98-99]. Breitbart等[62]总结了病毒裂解作用在海洋中所导致的DOM再循环过程. 捕食作用将碳沿着食物链从细菌到原生动物、浮游动物再到鱼类和更大的生物方向移动,所有营养级都通过产生颗粒性有机物(POM)下沉影响海洋有机物库. 而病毒的裂解作用导致碳和营养物质通过病毒裂解的方式流动,并被微生物循环中的细菌重新矿化、促进水体中DOM快速循环这一过程被称为病毒分流(viral shunt)[62]. 噬藻体作为浮游病毒的重要组成,其对蓝藻的裂解作用(图 3)对促进有机物循环具有重要影响. 噬藻体裂解聚球藻、原绿球藻等蓝藻促进DOM循环,提高海洋碳库存量. Suttle等[11]发现墨西哥湾聚球藻属在噬藻体的作用下日致死率达到5 % ~14 %,子代噬藻体的每日释放量为3.1× 107 pfu/L,聚球藻每日碳释放量为0.15 μg/L(假设单个聚球藻细胞碳含量为125 fg)[100].
|
图 3 噬藻体裂解蓝藻对海洋中有机物循环的影响 Fig.3 Effects of cyanophage on organic matter cycling in the ocean |
噬藻体裂解宿主细胞释放大量溶解性有机碳,改变了水体中碳源分配,使大量碳从颗粒有机碳库流入溶解性有机碳库. Zheng等[101]通过噬藻体感染聚球藻实验探究噬藻体对水体中有机碳分布的影响. 在噬藻体裂解期间,培养体系中POC浓度降低了约680 μmol/L;而噬藻体添加组DOC相对于对照组则增加约140 μmol/L. 水体中有机碳向无机碳的转化效率约为20 %,大概有80 % 的聚球藻裂解被吸收利用或转化为CO2. 且添加噬藻体实验组的碳遵循“DOC-细菌-DOC”循环,相对较高浓度的DOC可持续5 d[102]. 噬藻体的裂解作用对蓝藻和异养细菌之间有机物质重新分配、无机养分再利用以及细菌生活方式具有显著的影响.
3.2 噬藻体对水体中氮磷循环过程影响蓝藻裂解释放的物质中还包含不同形态的氮和磷,溶解态氮和磷可直接被浮游植物利用,从而增加水体初级生产力. 细胞裂解释放的蛋白质、核酸以及一些细胞成分中富含大量的有机态氮、磷,有机态磷在微生物作用下转化为易被浮游植物吸收的无机态化合物. 与氮(主要是氨氮)相比,水体中磷元素的矿化效率较低. 在聚球藻-异氧细菌培养体系中添加噬藻体,发现噬藻体裂解作用并未使培养体系中无机磷浓度增加. 含磷有机物主要进入异养细菌或病毒颗粒中,或者保留在最初降解的化合物中,而不是被重新矿化为无机磷酸盐[101]. 溶解性有机氮在细菌矿化作用下转化为氨,氨是海洋初级生产力的最主要氮源,噬藻体诱导的氨氮再生会导致浮游植物生物量增加[103]. 夏威夷附近海域的束毛藻每天对N2固氮率约为43 pmol,而噬藻体裂解、释放3 % ~65 % 氮[87],从而影响周围水域氮循环过程. Kuznecova等[104]利用噬藻体vB_AphaS-CL131感染丝状固氮水华束丝藻(Aphanizomenon flos-aquae),发现噬藻体抑制宿主藻生长,但藻细胞氮固定率并没有显著差异,这说明细胞内噬藻体基因组复制和子代噬藻体产生不会影响宿主藻异形胞中N2固定. 异形胞两端氮含量增加,营养细胞氮转运减少,藻丝内固定氮流动方向发生变化,而噬藻体的裂解作用导致细胞内氨释放到水体中. 因此,噬藻体感染蓝藻后,导致丝状宿主细胞内的氮分布改变,诱导胞内氮转化为含氮化合物并释放到水体,促进了颗粒态氮向溶解态氮转化. 噬藻体侵染蓝藻对自然水体中氮、磷循环过程具有显著影响,但现阶段关于噬藻体侵染作用对水体中营养物质循环的相关研究仍主要在海洋领域,噬藻体在淡水水体中对氮、磷等营养物质循环过程影响仍处于探索阶段.
3.3 噬藻体裂解蓝藻对水体中藻毒素影响噬藻体裂解蓝藻后是否能引起水体中藻毒素的变化一直是研究人员非常关切的话题. 最近一项关于藻毒素的报道引起了全球关注,在2020年5--6月,非洲大草原330多头大象离奇死亡. Wang等[105]认为非洲大陆水体微囊藻毒素平均浓度超过8800 μg/L,这可能是导致大象死亡的直接原因. 自从1995年以来,美国伊利湖每年夏季都会暴发以微囊藻为优势群落的蓝藻水华. 在2014年夏季伊利湖中噬藻体大量爆发,噬藻体裂解微囊藻释放藻毒素导致水体中藻毒素浓度快速升高(主要为微囊藻毒素MC-LR、MC-RR、MC-RR,最高浓度可达37.1 μg/L),且来自西北方向的强风导致毛米湾富含微囊藻毒素的水沿着南部海岸线向东输送经过托莱多取水口,导致该市饮用水供水中断2 d. 该研究发现噬藻体对微囊藻群落的大规模感染可能有利于藻毒素在水体中从颗粒态向溶解态转化,使水体藻毒素浓度在短期内快速增加,导致藻毒素在浮游生物体内累积、危害水生态系统[106].
现阶段,噬藻体裂解蓝藻释放的大分子物质对水生态系统影响的相关研究仍然非常少. 在2018年,Šulčius等[49]首次在实验室条件下探究了噬藻体感染和裂解对蓝藻产生的节球藻毒素和其他非核糖体肽的影响. 在单一培养体系内利用噬藻体vB_NodS-kac68v162-1感染丝状宿主节球藻,培养初期水体中节球藻毒素浓度为19.8 μg/L;噬藻体侵染宿主藻144 h后,培养体系中溶解性节球藻毒素浓度增加了3倍. 相比之下,未加入噬藻体的培养体系中节球藻毒素浓度减少至1.0 μg/L. 这说明噬藻体感染对丝状蓝藻释放藻毒素存在较大影响,并证明病毒导致的宿主细胞裂解对节球藻毒素从颗粒态向溶解态转化的重要作用. 研究还发现噬藻体裂解使节球藻丝状体断裂,较短丝状体数量相对增加,水体中剩余具有抗性的宿主藻具有更高浓度的节球藻毒素,使其在水体中比其他藻类更具有竞争优势. 但该研究是在实验室单一培养体系下完成的,自然水体中微生物种类繁多,在噬藻体和细菌等联合作用下水体中藻毒素形态及浓度可能与实验室内得到的结果有所差异. Lin等通过微宇宙实验探究噬藻体vB_MelS-Me-ZS1对复杂水体蓝藻丰度的调节作用以及对培养体系中锦鲤的影响. 研究发现,加入噬藻体可以有效抑制蓝藻生长、使培养体系内蓝藻丰度降低;噬藻体裂解宿主藻,有机物释放后被水体中细菌利用,且腐螺菌科相对丰度增加,其对活性污泥的膨胀具有诱导作用. 噬藻体加入可能导致活性污泥快速、大量形成,对水质有较好的净化功能;且噬藻体的加入使水体中锦鲤存活率高于未加入噬藻体的对照组,说明其对水中生物有明显的保护作用[58]. Naknaen等[59]在最近的研究中揭示了噬藻体PhiMa05可以裂解水体中单一的或聚集的微囊藻细胞,并首次提出噬藻体会干扰微囊藻毒素生成. 噬藻体PhiMa05侵染微囊藻后,胞内藻毒素浓度显著下降. 宿主藻裂解向水体中释放微囊藻毒素,但显著低于对照组的胞内藻毒素浓度. 噬藻体裂解微囊藻使得总的微囊藻毒素浓度明显减少,这证明噬藻体对微囊藻毒素的产生可能具有抑制作用.
噬藻体侵染可能会使产毒蓝藻迅速裂解,并在短期内向水体释放大量藻毒素,促进颗粒态藻毒素向溶解态转化,从而使水体中藻毒素含量增加,危害水生态系统. 而在自然水体中,噬藻体-蓝藻相互作用会导致水体中有机物、微生物群落等发生变化,这也可能会对藻毒素浓度产生一定影响. 噬藻体通过影响蓝藻丰度进而影响水体中藻毒素浓度的相关研究仍较少.
4 总结与展望本文从噬藻体侵染机制,噬藻体对蓝藻种群的调节作用以及对水生态系统的潜在生态效应等方面分析了现有研究现状及其进展. 噬藻体作为自然环境中快速裂解蓝藻的生物因子,其对蓝藻水华的治理具有重要研究意义和潜在应用价值. 噬藻体裂解宿主藻可以通过提供营养物质来促进群落中相互竞争的藻类生长,使水体中生物群落发生改变,可能间接影响水生态系统的生物多样性. 蓝藻在噬藻体作用下裂解,破坏原有的水生态捕食食物链、改变了浮游植物的群落结构,使有机物向食物链顶端传递效率降低,并改变水体中关键元素的循环过程. 由于噬藻体对蓝藻侵染具有较强特异性,同时宿主藻易产生抗性并且可能导致藻毒素释放风险增加,使噬藻体在实际环境中的应用仍然存在困难;目前,噬藻体对蓝藻水华的调控作用仍停留在室内实验阶段. 尽管室内研究结果显示噬藻体可有效地控制蓝藻种群密度,但在更为复杂的自然水体中,这些结论是否成立仍然值得进一步探索和明确,尤其应该关注在实际应用过程中的水生态风险变化,这可以为全面地评估噬藻体作为蓝藻水华潜在生物治理方案可行性提供重要基础.
| [1] |
Ndlela LL, Oberholster PJ, van Wyk JH et al. An overview of cyanobacterial bloom occurrences and research in Africa over the last decade. Harmful Algae, 2016, 60: 11-26. DOI:10.1016/j.hal.2016.10.001 |
| [2] |
Freedman B. Environmental ecology. San Diego: Academic Press, 2002.
|
| [3] |
Ministry of Ecology and Environment of the People's Republic of China ed. 2018 Bulletin of China's Ecological Environment, 2019. [中华人民共和国生态环境部. 2018年中国生态环境状况公报, 2019. ]
|
| [4] |
Loftin KA, Graham JL, Hilborn ED et al. Cyanotoxins in inland lakes of the United States: Occurrence and potential recreational health risks in the EPA National Lakes Assessment 2007. Harmful Algae, 2016, 56(6): 77-90. DOI:10.1016/j.hal.2016.04.001 |
| [5] |
Palagama DSW, Baliu-Rodriguez D, Snyder BK et al. Identification and quantification of microcystins in western Lake Erie during 2016 and 2017 harmful algal blooms. Journal of Great Lakes Research, 2020, 46(2): 289-301. DOI:10.1016/j.jglr.2020.01.002 |
| [6] |
Liu TT, Liu L, Wei DQ et al. The role of horizontal gene transfer and co-evolution mechanism between cyanophage and cyanobacteria. Biotechnology Bulletin, 2011(7): 12-17. [刘腾腾, 刘丽, 魏大巧等. 噬藻体和蓝藻间的基因转移及协同进化作用. 生物技术通报, 2011(7): 12-17.] |
| [7] |
Bergh Ø, Børsheim KY, Bratbak G et al. High abundance of viruses found in aquatic environments. Nature, 1989, 340(6233): 467-468. DOI:10.1038/340467a0 |
| [8] |
van Wichelen J, Vanormelingen P, Codd GA et al. The common bloom-forming cyanobacterium Microcystis is prone to a wide array of microbial antagonists. Harmful Algae, 2016, 55: 97-111. DOI:10.1016/j.hal.2016.02.009 |
| [9] |
Jaskulska A, Mankiewicz-Boczek J. Cyanophages specific to cyanobacteria from the genus Microcystis. Ecohydrology & Hydrobiology, 2020, 20(1): 83-90. DOI:10.1016/j.ecohyd.2019.06.001 |
| [10] |
Tucker S, Pollard P. Identification of cyanophage Ma-LBP and infection of the cyanobacterium Microcystis aeruginosa from an Australian subtropical lake by the virus. Applied and Environmental Microbiology, 2005, 71(2): 629-635. DOI:10.1128/AEM.71.2.629-635.2005 |
| [11] |
Suttle CA, Chan AM. Dynamics and distribution of cyanophages and their effect on marine Synechococcus spp. Applied and Environmental Microbiology, 1994, 60(9): 3167-3174. DOI:10.1128/aem.60.9.3167-3174.1994 |
| [12] |
Suttle CA, Chan AM, Cottrell MT. Infection of phytoplankton by viruses and reduction of primary productivity. Nature, 1990, 347(6292): 467-469. DOI:10.1038/347467a0 |
| [13] |
Wommack KE, Colwell RR. Virioplankton: viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews, 2000, 64(1): 69-114. DOI:10.1128/mmbr.64.1.69-114.2000 |
| [14] |
Tuomi P, Kuuppo P. Viral lysis and grazing loss of bacteria in nutrient and carbon manipulated brackish water enclosures. Journal of Plankton Research, 1999, 21(5): 923-937. DOI:10.1093/plankt/21.5.923 |
| [15] |
Wilhelm SW, Matteson AR. Freshwater and marine virioplankton: A brief overview of commonalities and differences. Freshwater Biology, 2008, 53(6): 1076-1089. DOI:10.1111/j.1365-2427.2008.01980.x |
| [16] |
Sandaa RA. Burden or benefit? Virus-host interactions in the marine environment. Research in Microbiology, 2008, 159(5): 374-381. DOI:10.1016/j.resmic.2008.04.013 |
| [17] |
Weinbauer MG. Ecology of prokaryotic viruses. FEMS Microbiology Reviews, 2004, 28(2): 127-181. DOI:10.1016/j.femsre.2003.08.001 |
| [18] |
Bouvier T, del Giorgio PA. Key role of selective viral-induced mortality in determining marine bacterial community composition. Environmental Microbiology, 2007, 9(2): 287-297. DOI:10.1111/j.1462-2920.2006.01137.x |
| [19] |
Weitz JS, Wilhelm SW. Ocean viruses and their effects on microbial communities and biogeochemical cycles. F1000 Biology Reports, 2012, 4(1): 17. DOI:10.3410/B4-17 |
| [20] |
Proctor LM, Fuhrman JA. Viral mortality of marine bacteria and cyanobacteria. Nature, 1990, 343(6253): 60-62. DOI:10.1038/343060a0 |
| [21] |
Mendzhul MI, Koltukova NV, Lysenko TG et al. Effect of reproduction of the LPP-3 cyanophage on glutamate dehydrogenase and glutamine synthetase activity in the cyanobacterium Plectonema boryanum. Ukrainskii Biokhimicheskii Zhurnal, 1995, 67(6): 33-37. |
| [22] |
King AMQ, Adams MJ, Carstens EB et al. Virus taxonomy: ninth report of the international committee on taxonomy of viruses. Amsterdam: Elsevier Academic Press, 2012. DOI:10.1007/BF01309873
|
| [23] |
Krupovic M, Prangishvili D, Hendrix RW et al. Genomics of bacterial and archaeal viruses: Dynamics within the prokaryotic virosphere. Microbiology and Molecular Biology Reviews, 2011, 75(4): 610-635. DOI:10.1128/MMBR.00011-11 |
| [24] |
Safferman RS, Morris ME. Algal virus: Isolation. Science, 1963, 140(3567): 679-680. DOI:10.1126/science.140.3567.679 |
| [25] |
Wilson WH, Joint IR, Carr NG et al. Isolation and molecular characterization of five marine cyanophages propagated on Synechococcus sp. strain WH7803. Applied and Environmental Microbiology, 1993, 59(11): 3736-3743. DOI:10.1128/aem.59.11.3736-3743.1993 |
| [26] |
Waterbury JB, Valois FW. Resistance to co-occurring phages enables marine Synechococcus communities to coexist with cyanophages abundant in seawater. Applied and Environmental Microbiology, 1993, 59(10): 3393-3399. DOI:10.1002/yea.320091115 |
| [27] |
Zhao YJ, Cheng K, Shi ZL et al. Isolation and identification of the first cyanophage in China. Progress in Natural Science, 2002, 12(9): 923-927. [赵以军, 程凯, 石正丽等. 我国首株噬藻体(蓝藻病毒)的分离与鉴定. 自然科学进展, 2002, 12(9): 923-927.] |
| [28] |
Jenkins CA, Hayes PK. Diversity of cyanophages infecting the heterocystous filamentous cyanobacterium Nodularia isolated from the brackish Baltic Sea. Journal of the Marine Biological Association of the United Kingdom, 2006, 86(3): 529-536. DOI:10.1017/s0025315406013439 |
| [29] |
Deng L, Hayes PK. Evidence for cyanophages active against bloom-forming freshwater cyanobacteria. Freshwater Biology, 2008, 53(6): 1240-1252. DOI:10.1111/j.1365-2427.2007.01947.x |
| [30] |
Wang GH, Asakawa S, Kimura M. Spatial and temporal changes of cyanophage communities in paddy field soils as revealed by the capsid assembly protein gene g20. FEMS Microbiology Ecology, 2011, 76(2): 352-359. DOI:10.1111/j.1574-6941.2011.01052.x |
| [31] |
Sherman LA, Connelly M. Isolation and characterization of a cyanophage infecting the unicellular blue-green algae A. nidulans and S. cedrorum. Virology, 1976, 72(2): 540-544. DOI:10.1016/0042-6822(76)90186-0 |
| [32] |
Fox JA, Booth SJ, Martin EL. Cyanophage SM-2: A new blue-green algal virus. Virology, 1976, 73(2): 557-560. DOI:10.1016/0042-6822(76)90420-7 |
| [33] |
Adolph KW, Haselkorn R. Isolation and characterization of a virus infecting the blue-green alga Nostoc muscorum. Virology, 1971, 46(2): 200-208. DOI:10.1016/0042-6822(71)90023-7 |
| [34] |
Sallal AKJ, Nimr ND, Al-Sharif HF. Isolation of Nostoc muscorum cyanophages from a domestic sewage. Microbial Ecology, 1987, 13(3): 261-268. DOI:10.1007/BF02025002 |
| [35] |
Chen F, Lu JR. Genomic sequence and evolution of marine cyanophage P60: A new insight on lytic and lysogenic phages. Applied and Environmental Microbiology, 2002, 68(5): 2589-2594. DOI:10.1128/AEM.68.5.2589-2594.2002 |
| [36] |
Gao EB, Gui JF, Zhang QY. A novel cyanophage with a cyanobacterial nonbleaching protein A gene in the genome. Journal of Virology, 2012, 86(1): 236-245. DOI:10.1128/JVI.06282-11 |
| [37] |
Yoshida T, Takashima Y, Tomaru Y et al. Isolation and characterization of a cyanophage infecting the toxic cyanobacterium Microcystis aeruginosa. Applied and Environmental Microbiology, 2006, 72(2): 1239-1247. DOI:10.1128/AEM.72.2.1239-1247.2006 |
| [38] |
Pope WH, Weigele PR, Chang J et al. Genome sequence, structural proteins, and capsid organization of the cyanophage Syn5: A "horned" bacteriophage of marine Synechococcus. Journal of Molecular Biology, 2007, 368(4): 966-981. DOI:10.1016/j.jmb.2007.02.046 |
| [39] |
Liu XY, Shi M, Kong SL et al. Cyanophage Pf-WMP4, a T7-like phage infecting the freshwater cyanobacterium Phormidium foveolarum: Complete genome sequence and DNA translocation. Virology, 2007, 366(1): 28-39. DOI:10.1016/j.virol.2007.04.019 |
| [40] |
Wu WZ, Zhu Q, Liu XY et al. Isolation of a freshwater cyanophage (F1) capable of infecting Anabaena flos-aquae and its potentials in the control of water bloom. International Journal of Environment and Pollution, 2009, 38(1/2): 212. DOI:10.1504/ijep.2009.026663 |
| [41] |
Kuznetsov YG, Martiny JBH, McPherson A. Structural analysis of a Synechococcus myovirus S-CAM4 and infected cells by atomic force microscopy. The Journal of General Virology, 2010, 91(Pt 12): 3095-3104. DOI:10.1099/vir.0.025254-0 |
| [42] |
Ou T, Li SH, Liao XY et al. Cultivation and characterization of the MaMV-DC cyanophage that infects bloom-forming cyanobacterium Microcystis aeruginosa. Virologica Sinica, 2013, 28(5): 266-271. DOI:10.1007/s12250-013-3340-7 |
| [43] |
Hargreaves KR, Anderson NJ, Clokie MRJ. Recovery of viable cyanophages from the sediments of a eutrophic lake at decadal timescales. FEMS Microbiology Ecology, 2013, 83(2): 450-456. DOI:10.1111/1574-6941.12005 |
| [44] |
Gao XC, Liao XY, Tong Z et al. Unraveling the genome structure of cyanobacterial podovirus A-4L with long direct terminal repeats. Virus Research, 2015, 203: 4-9. DOI:10.1016/j.virusres.2015.03.012 |
| [45] |
Li SH, Ou T, Zhang QY. Two virus-like particles that cause lytic infections in freshwater cyanobacteria. Virologica Sinica, 2013, 28(5): 303-305. DOI:10.1007/s12250-013-3339-0 |
| [46] |
Šulčius S, Šimoliūnas E, Staniulis J et al. Characterization of a lytic cyanophage that infects the bloom-forming cyanobacterium Aphanizomenon flos-aquae. FEMS Microbiology Ecology, 2015, 91(2): 1-7. DOI:10.1093/femsec/fiu012 |
| [47] |
Chénard C, Chan AM, Vincent WF et al. Polar freshwater cyanophage S-EIV1 represents a new widespread evolutionary lineage of phages. The ISME Journal, 2015, 9(9): 2046-2058. DOI:10.1038/ismej.2015.24 |
| [48] |
Coloma SE, Dienstbier A, Bamford DH et al. Newly isolated Nodularia phage influences cyanobacterial community dynamics. Environmental Microbiology, 2017, 19(1): 273-286. DOI:10.1111/1462-2920.13601 |
| [49] |
Šulčius S, Mazur-Marzec H, Vitonyté I et al. Insights into cyanophage-mediated dynamics of nodularin and other non-ribosomal peptides in Nodularia spumigena. Harmful Algae, 2018, 78: 69-74. DOI:10.1016/j.hal.2018.07.004 |
| [50] |
Watkins SC, Smith JR, Hayes PK et al. Characterisation of host growth after infection with a broad-range freshwater cyanopodophage. PLoS One, 2014, 9(1): e87339. DOI:10.1371/journal.pone.0087339 |
| [51] |
Martin RM, Moniruzzaman M, Mucci NC et al. Cylindrospermopsis raciborskii virus and host: Genomic characterization and ecological relevance. Environmental Microbiology, 2019, 21(6): 1942-1956. DOI:10.1111/1462-2920.14425 |
| [52] |
You SY, Wang M, Jiang Y et al. The genome sequence of a novel cyanophage S-B64 from the Yellow Sea, China. Current Microbiology, 2019, 76(6): 681-686. DOI:10.1007/s00284-019-01680-1 |
| [53] |
Zhang D, You F, He Y et al. Isolation and characterization of the first freshwater cyanophage infecting Pseudanabaena. Journal of Virology, 2020, 94(17). DOI:10.1128/JVI.00682-20 |
| [54] |
Jiang T, Guo C, Wang M et al. Isolation and complete genome sequence of a novel cyanophage, S-B05, infecting an estuarine Synechococcus strain: insights into environmental adaptation. Archives of Virology, 2020, 165(24). DOI:10.1007/s00705-020-04595-6 |
| [55] |
Yang F, Jin H, Wang XQ et al. Genomic analysis of Mic1 reveals a novel freshwater long-tailed cyanophage. Frontiers in Microbiology, 2020, 11: 484. DOI:10.3389/fmicb.2020.00484 |
| [56] |
Huang LY, Liu Q, Liu XX et al. Isolation and complete genome sequence of a novel cyanophage S-B68. Current Microbiology, 2020, 77(9): 2385-2390. DOI:10.1007/s00284-020-02045-9 |
| [57] |
Xue CL, Liu XX, Wang Q et al. The isolation and genome sequencing of a novel cyanophage S-H68 from the Bohai Sea, China. Marine Genomics, 2020, 53: 100739. DOI:10.1016/j.margen.2019.100739 |
| [58] |
Lin W, Li DF, Sun ZT et al. A novel freshwater cyanophage vB_MelS-Me-ZS1 infecting bloom-forming cyanobacterium Microcystis elabens. Molecular Biology Reports, 2020, 47(10): 7979-7989. DOI:10.1007/s11033-020-05876-8 |
| [59] |
Naknaen A, Suttinun O, Surachat K et al. A novel jumbo phage PhiMa05 inhibits harmful Microcystis sp. Frontiers in Microbiology, 2021, 12: 660351. DOI:10.3389/fmicb.2021.660351 |
| [60] |
Ortmann AC, Lawrence JE, Suttle CA. Lysogeny and lytic viral production during a bloom of the cyanobacterium Synechococcus spp. Microbial Ecology, 2002, 43(2): 225-231. DOI:10.1007/s00248-001-1058-9 |
| [61] |
Paul JH. Prophages in marine bacteria: Dangerous molecular time bombs or the key to survival in the seas?. The ISME Journal, 2008, 2(6): 579-589. DOI:10.1038/ismej.2008.35 |
| [62] |
Breitbart M, Bonnain C, Malki K et al. Phage puppet masters of the marine microbial realm. Nature Microbiology, 2018, 3(7): 754-766. DOI:10.1038/s41564-018-0166-y |
| [63] |
Clokie M, Mann NH. Marine cyanophages and light. Environmental Microbiology, 2010, 8(12): 2074-2082. DOI:10.1111/j.1462-2920.2006.01171.x |
| [64] |
Bailey S, Clokie MRJ, Millard A et al. Cyanophage infection and photoinhibition in marine cyanobacteria. Research in Microbiology, 2004, 155(9): 720-725. DOI:10.1016/j.resmic.2004.06.002 |
| [65] |
Liu R, Liu Y, Chen Y et al. Cyanobacterial viruses exhibit diurnal rhythms during infection. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(28). DOI:10.1073/pnas.1819689116 |
| [66] |
Cséke CS, Farkas GL. Effect of light on the attachment of cyanophage AS-1 to Anacystis nidulans. Journal of Bacteriology, 1979, 137(1): 667-669. DOI:10.1128/JB.137.1.667-669.1979 |
| [67] |
Cheng K, Frenken T, Brussaard CPD et al. Cyanophage propagation in the freshwater cyanobacterium Phormidium is constrained by phosphorus limitation and enhanced by elevated pCO2. Frontiers in Microbiology, 2019, 10: 617. DOI:10.3389/fmicb.2019.00617 |
| [68] |
Zhao YJ, Shi ZL, Huang GJ et al. Blue-green algal viruses (cyanophages). Virologica Sinica, 1999, 14(2): 100-105. [赵以军, 石正丽, 黄国锦等. 蓝藻病毒(噬藻体)的研究进展. 中国病毒学, 1999, 14(2): 100-105.] |
| [69] |
Puxty RJ, Millard AD, Evans DJ et al. Viruses inhibit CO2 fixation in the most abundant phototrophs on earth. Current Biology, 2016, 26(12): 1585-1589. DOI:10.1016/j.cub.2016.04.036 |
| [70] |
Cheng K, van de Waal DB, Niu XY et al. Combined effects of elevated pCO2 and warming facilitate cyanophage infections. Frontiers in Microbiology, 2017, 8: 1096. DOI:10.3389/fmicb.2017.01096 |
| [71] |
Niu XY, Cheng K, Rong QQ et al. The combined effects of elevated CO2 and elevated temperature on proliferation of cyanophage PP. Acta Ecologica Sinica, 2012, 32(22): 6917-6924. [牛晓莹, 程凯, 荣茜茜等. CO2浓度和温度升高对噬藻体PP增殖的联合作用. 生态学报, 2012, 32(22): 6917-6924. DOI:10.5846/stxb201110181547] |
| [72] |
Robb SM, Woods DR, Robb FT. Phage growth characteristics on stationary phase Achromobacter cells. The Journal of General Virology, 1978, 41(2): 265-272. DOI:10.1099/0022-1317-41-2-265 |
| [73] |
Kokjohn TA, Sayler GS. Attachment and replication of Pseudomonas aeruginosa bacteriophages under conditions simulating aquatic environments. Journal of General Microbiology, 1991, 137(3): 661-666. DOI:10.1099/00221287-137-3-661 |
| [74] |
Ogunseitan OA, Sayler GS, Miller RV. Dynamic interactions of Pseudomonas aeruginosa and bacteriophages in lake water. Microbial Ecology, 1990, 19(2): 171-185. DOI:10.1007/BF02012098 |
| [75] |
Suttle CA. Marine viruses—major players in the global ecosystem. Nature Reviews Microbiology, 2007, 5(10): 801-812. DOI:10.1038/nrmicro1750 |
| [76] |
Sandaa RA, Larsen A. Seasonal variations in virus-host populations in Norwegian coastal waters: Focusing on the cyanophage community infecting marine Synechococcus spp. Applied and Environmental Microbiology, 2006, 72(7): 4610-4618. DOI:10.1128/AEM.00168-06 |
| [77] |
Avrani S, Schwartz DA, Lindell D. Virus-host swinging party in the oceans: Incorporating biological complexity into paradigms of antagonistic coexistence. Mobile Genetic Elements, 2012, 2(2): 88-95. DOI:10.4161/mge.20031 |
| [78] |
Sun YT, Zhang S, Long LJ et al. Genetic diversity and cooccurrence patterns of marine cyanopodoviruses and picocyanobacteria. Applied and Environmental Microbiology, 2018, 84(16). DOI:10.1128/aem.00591-18 |
| [79] |
Avrani S, Wurtzel O, Sharon I et al. Genomic island variability facilitates Prochlorococcus-virus coexistence. Nature, 2011, 474(7353): 604-608. DOI:10.1038/nature10172 |
| [80] |
Zhang QY, Gui JF. Diversity, evolutionary contribution and ecological roles of aquatic viruses. Science China Life Sciences, 2018, 61(12): 1486-1502. DOI:10.1007/s11427-018-9414-7 |
| [81] |
Gregory AC, Solonenko SA, Ignacio-Espinoza JC et al. Genomic differentiation among wild cyanophages despite widespread horizontal gene transfer. BMC Genomics, 2016, 17(1): 1-13. DOI:10.1186/s12864-016-3286-x |
| [82] |
Zborowsky S, Lindell D. Resistance in marine cyanobacteria differs against specialist and generalist cyanophages. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(34): 16899-16908. DOI:10.1073/pnas.1906897116 |
| [83] |
Zhong KX, Suttle CA, Baudoux AC et al. A new freshwater cyanosiphovirus harboring integrase. Frontiers in Microbiology, 2018, 9: 2204. DOI:10.3389/fmicb.2018.02204 |
| [84] |
Keen EC, Bliskovsky VV, Malagon F et al. Novel "superspreader" bacteriophages promote horizontal gene transfer by transformation. mBio, 2017, 8(1). DOI:10.1128/mbio.02115-16 |
| [85] |
Ortmann AC, Wiedenheft B, Douglas T et al. Hot crenarchaeal viruses reveal deep evolutionary connections. Nature Reviews Microbiology, 2006, 4(7): 520-528. DOI:10.1038/nrmicro1444 |
| [86] |
Coello-Camba A, Diaz-Rua R, Duarte CM et al. Picocyanobacteria community and cyanophage infection responses to nutrient enrichment in a mesocosms experiment in oligotrophic waters. Frontiers in Microbiology, 2020, 11: 1153. DOI:10.3389/fmicb.2020.01153 |
| [87] |
Hewson I, Govil SR, Capone DG et al. Evidence of Trichodesmium viral lysis and potential significance for biogeochemical cycling in the oligotrophic ocean. Aquatic Microbial Ecology, 2004, 36: 1-8. DOI:10.3354/ame036001 |
| [88] |
Wang K, Wommack KE, Chen F. Abundance and distribution of Synechococcus spp. and cyanophages in the Chesapeake bay. Applied and Environmental Microbiology, 2011, 77(21): 7459-7468. DOI:10.1128/aem.00267-11 |
| [89] |
Wang JP, Bai P, Li Q et al. Interaction between cyanophage MaMV-DC and eight Microcystis strains, revealed by genetic defense systems. Harmful Algae, 2019, 85: 101699. DOI:10.1016/j.hal.2019.101699 |
| [90] |
Zhou QC, Wei N, Zheng LL et al. Host re-identification of cyanophage PP and its implications for host range and specificity. Virologica Sinica, 2013, 28(5): 306-308. DOI:10.1007/s12250-013-3356-z |
| [91] |
Liao MJ. Ecological effect in microcosm systems after the lysis of cyanobacteria host by cyanophage PP[Dissertation]. Wuhan: Central China Normal University, 2005. [廖明军. 微宇宙中噬藻体PP裂解蓝藻宿主的生态学效应研究[学位论文]. 武汉: 华中师范大学, 2005. ]
|
| [92] |
Šulčius S, Slavuckyté K, Paškauskas R. The predation paradox: Synergistic and antagonistic interactions between grazing by crustacean predator and infection by cyanophages promotes bloom formation in filamentous cyanobacteria. Limnology and Oceanography, 2017, 62(5): 2189-2199. DOI:10.1002/lno.10559 |
| [93] |
Mann NH. Phages of the marine cyanobacterial picophytoplankton. FEMS Microbiology Reviews, 2003, 27(1): 17-34. DOI:10.1016/S0168-6445(03)00016-0 |
| [94] |
Avrani S, Lindell D. Convergent evolution toward an improved growth rate and a reduced resistance range in Prochlorococcus strains resistant to phage. PNAS, 2015, 112(17): E2191-E2200. DOI:10.1073/pnas.1420347112 |
| [95] |
Manage PM, Kawabata Z, Nakano SI. Seasonal changes in densities of cyanophage infectious to Microcystis aeruginosa in a hypereutrophic pond. Hydrobiologia, 1999, 411: 211-216. DOI:10.1023/A:1003868803832 |
| [96] |
Yoshida M, Yoshida T, Kashima A et al. Ecological dynamics of the toxic bloom-forming cyanobacterium Microcystis aeruginosa and its cyanophages in freshwater. Applied and Environmental Microbiology, 2008, 74(10): 3269-3273. DOI:10.1128/AEM.02240-07 |
| [97] |
Peduzzi P, Gruber M, Gruber M et al. The virus's tooth: Cyanophages affect an African flamingo population in a bottom-up cascade. The ISME Journal, 2014, 8(6): 1346-1351. DOI:10.1038/ismej.2013.241 |
| [98] |
Ma XF, Coleman ML, Waldbauer JR. Distinct molecular signatures in dissolved organic matter produced by viral lysis of marine cyanobacteria. Environmental Microbiology, 2018, 20(8): 3001-3011. DOI:10.1111/1462-2920.14338 |
| [99] |
Biggs TEG, Huisman J, Brussaard CPD. Viral Lysis modifies seasonal phytoplankton dynamics and carbon flow in the Southern Ocean. The ISME Journal, 2021, 1-8. DOI:10.1038/s41396-021-01033-6 |
| [100] |
Wilhelm SW, Suttle CA. Viruses and nutrient cycles in the sea-viruses play critical roles in the structure and function of aquatic food webs. BioScience, 1999, 49(10): 781-788. DOI:10.2307/1313569 |
| [101] |
Zheng Q, Lin WX, Wang Y et al. Top-down controls on nutrient cycling and population dynamics in a model estuarine photoautotroph-heterotroph co-culture system. Molecular Ecology, 2021, 30(2): 592-607. DOI:10.1111/mec.15750 |
| [102] |
Bratbak G, Heldal M, Thingstad TF et al. Incorporation of viruses into the budget of microbial C-transfer A first approach. Marine Ecology Progress Series, 1992, 83: 273-280. DOI:10.3354/meps083273 |
| [103] |
Shelford EJ, Middelboe M, Møller EF et al. Virus-driven nitrogen cycling enhances phytoplankton growth. Aquatic Microbial Ecology, 2012, 66(1): 41-46. DOI:10.3354/ame01553 |
| [104] |
Kuznecova J, Šulčius S, Vogts A et al. Nitrogen flow in diazotrophic cyanobacterium Aphanizomenon flos-aquae is altered by cyanophage infection. Frontiers in Microbiology, 2020, 11: 2010. DOI:10.3389/fmicb.2020.02010 |
| [105] |
Wang HJ, Xu C, Liu Y et al. From unusual suspect to serial killer: Cyanotoxins boosted by climate change may jeopardize megafauna. The Innovation, 2021, 2(2): 100092. DOI:10.1016/j.xinn.2021.100092 |
| [106] |
Steffen MM, Davis TW, McKay RML et al. Ecophysiological examination of the Lake Erie Microcystis bloom in 2014: Linkages between biology and the water supply shutdown of Toledo, OH. Environmental Science & Technology, 2017, 51(12): 6745-6755. DOI:10.1021/acs.est.7b00856 |
 2022, Vol. 34
2022, Vol. 34