(2: 青海大学, 省部共建三江源生态与高原农牧业国家重点实验室, 西宁 810016)
(3: 青海大学, 黄河上游生态保护与高质量发展实验室, 西宁 810016)
(4: 青海大学, 水利部江河源区水生态治理与保护重点实验室, 西宁 810016)
(2: State Key laboratory of Plateau Ecology and Agriculture, Qinghai University, Xining 810016, P. R. China)
(3: Provincial Laboratory of Ecological Protection and High Quality Development in the Upper Yellow River, Qinghai University, Xining 810016, P. R. China)
(4: Key Laboratory of Water Ecological Remediation and Protection at Headwater Regions of Big Rivers, Ministry of Water Resources, Qinghai University, Xining 810016, P. R. China)
湖泊是陆地地表主要的蓄水载体之一,对区域内水资源的调度和生态系统的支撑具有重要作用,是生物与非生物因子共同作用下产生的自然综合体[1]。微生物参与并驱动湖泊生态系统中C、N、P等元素的循环和能量的流动[2]。湖泊微生物受到水体物理化学参数(如:温度、pH、电导率、营养物质浓度和离子)的影响,表现出显著的季节性,进而影响了湖泊生态系统的生物地球化学循环过程[3]。
微生物多样性、群落结构、功能和分布的形成机制是环境选择和扩散限制相互作用的结果[4]。中性理论和生态位理论是解析微生物群落构建的两个相互补充的重要机制[5]。中性理论认为自然界中微生物具有生态位等效性,主要包括随机的出生、死亡、迁移、灭绝、物种形成和有限扩散[6]。相反地,生态位理论认为微生物的扩散能力是无限的,而微生物群落中不同的物种有不同的生境偏好性,微生物群落的形成归因于环境因子的选择和各种生物之间的相互作用[5]。随机过程导致微生物群落发生不可预测的变化,而基于生态位理论的确定性过程会影响微生物群落的适应性,进而影响微生物群落的丰度和组成[6]。因此,微生物群落构建的确定性和随机性过程之间的相互补充是充分理解微生物多样性形成的关键。2006年,Sloan等建立了中性模型,拟合OTU的出现频率,量化中性过程在物种分布过程中所占的比例(R2)和评估物种的迁移率(m)[7]。目前,中性模型已被证实可以量化不同生境(比如河流和湖泊)中微生物群落物种丰度的分布情况[8-11]。Chen等基于Sloan中性模型,解析了随机性过程是亚热带河流在干燥和潮湿季节真核微生物群落的主要构建过程[4]。此外,Liu等使用Sloan中性模型量化研究长江浮游和沉积物细菌群落地理分布的结果表明,中性过程非常适合大型河流系统中细菌的群落分布[10]。随后,Stegen等基于Beta最近分类单元指数(Nearest Taxon Index-NTI)和Raup-Crick指数(RCbray)进一步量化了确定性和随机性过程对微生物群落构建的贡献比例[12-13]。例如,Li等使用零模型解析三峡水库沉积物细菌群落构建过程,发现同向选择是沉积物细菌群落的主要构建过程[14]。2020年,Gad等同时使用中性模型和零模型研究河流真核微生物的泛化种和特化种群落构建过程,结果表明确定性过程和随机性过程共同影响河流真核微生物群落构建过程[15]。同时,2022年Liu等使用零模型研究西藏区域23个湖泊在8月细菌和真核微生物群落的构建过程,发现确定性过程是细菌群落的主要构建过程,而随机性过程是真核微生物群落的主要构建过程[16]。
青藏高原东北部地处我国东部季风区、西北干旱区和青藏高原高寒区的交汇地带,是典型的干旱与半干旱气候区[17],自然发育了众多内陆咸水湖和盐湖群。近年来青藏高原的气候逐渐由暖干向暖湿化发展,导致湖泊面积快速扩增、水位上升和水体理化因子改变[18],进而影响了湖泊微生物多样性和群落结构,造成了湖泊微生物群落结构明显的季节差异[19]。前人研究西藏纳木错湖泊细菌群落结构,发现细菌丰度在7月最高,1月最低,并且放线菌、蓝细菌和Beta变形菌纲对季节变化的响应最为明显[20]。2013年,张红光等研究青海湖和纳木错湖水的可培养微生物季节的变化,发现在8月份微生物多样性到达最大值[21]。另外,李国强的研究结果表明青海湖在8和10月,湖水的微生物丰富度和多样性最高[22]。湖泊微生物群落结构的季节变化主要是水体理化因子驱动的结果,其中温度主要影响微生物的生长和代谢过程中各种酶的活性;盐度主要通过控制微生物细胞内外渗透压来影响微生物的功能代谢;营养盐主要为微生物的代谢提供营养底物[16]。目前,已有的研究主要集中在对青藏高原湖泊群单一季节和单一湖泊多季节的研究中[23-24],针对青藏高原东北部湖泊群微生物特征季节差异研究的报道较少,对微生物群落构建过程的研究更少。
因此,本文选择青藏高原东北部湖泊群为研究对象,通过现场采集水样并对其进行高通量测序分析和统计分析,研究湖泊群细菌群落特征及其季节变化的驱动因素。同时,使用中性模型和零模型分析,解析中性过程、确定性过程和随机性过程分别对不同季节细菌群落构建的贡献比例,系统地解析了微生物群落的季节分布特征。开展的研究一方面能够补充高海拔湖泊生态系统细菌群落特征的本底数据,另一方面揭示不同季节湖泊细菌群落变化的驱动因素、构建过程及其差异,对于深入理解高海拔湖泊细菌群落构建和湖泊水资源管理具有重要的支撑意义。
1 材料与方法 1.1 采样点描述青藏高原暖季为5—10月,冷季为11—4月[25-26],本研究选择采样时间为2020年6和12月,对应的季节在本文中表述为非冰封期和冰封期,在两个时期对青藏高原东北部青海湖(QHL,非冰封期N=6,冰封期N=5)、尕海(GHL,N=3)、托素湖(TSL,非冰封期N=4,冰封期N=3)、可鲁克湖(KLKL,N =3)和小柴旦湖(XCDL,N =4)5个湖泊进行表层水样的采集。考虑到面积的大小不同,不同湖泊设置不同的样点数(N),样点主要分布在入/出湖口或者湖边/湖心以及两者之间的中点上,具体分布如图 1所示。
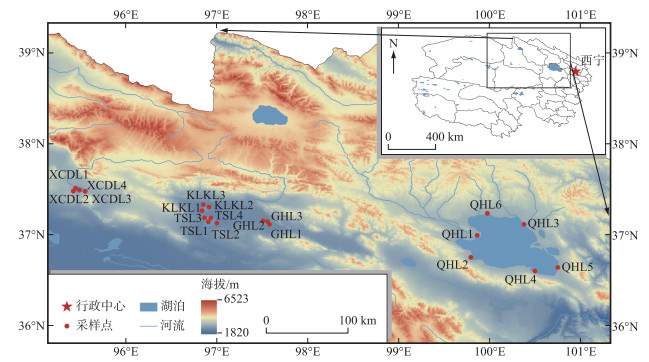
|
图 1 研究湖泊分布及各自采样点分布 Fig.1 Distribution of the investigated lakes as well as corresponding sampling sites |
在每个样点水深约为0.5 m的位置进行水样的采集,每个样点采集水样5份,每份约1 L,装入已灭菌的窄口玻璃瓶,4℃保存运往实验室。采集的水样中3份用0.22 μm孔隙的滤膜(Millipore S-Pak Germany)进行抽真空过滤预处理,过滤后将微生物富集的滤膜放置在无菌的离心管中,放置在-80℃保存,用于后续细菌总DNA提取。同时将剩余的水样低温黑暗保存,用于实验室水体营养盐和溶解有机碳浓度检测。
1.3 湖泊水体物理化学参数检测采用便携式水质仪(EXOTM2 Sonde, SY1, USA)在现场采样的同时测定水温(Temp)、pH、溶解氧(DO)、电导率(EC)、氧化还原电位(ORP)、盐度(SAL)、总溶解固体(TDS)、浊度(Turb)、叶绿素a(Chl.a)。另外,总氮(TN)、总磷(TP)、氨氮(NH3-N)、硝态氮(NO3--N)、亚硝态氮(NO2--N)和溶解有机碳(DOC)的浓度检测分别采用如下标准进行:Q/QHSHJ 09—2012、Q/QHSHJ08—2012、HJ535—2009、SL84—1994、GB/T5750.5—2006和HJ501—2009。
1.4 总DNA提取和PCR过程将冷冻保存的细菌滤膜采用CTAB法提取总DNA,所用试剂和操作流程根据参考文献[27]。用1%(w/v)浓度的琼脂糖凝胶电泳检测总DNA纯度。用引物515F(5′-GTGCCAGCMGCCGCGGTTA-3′)/806R(5′-GGACTACHVGGGTWTCTAAT-3′)对细菌16S r RNA V4区进行聚合酶链式反应[28]。反应体系及其流程依据我们先前研究中的PCR扩增体系和流程[29]。
1.5 生物信息学分析质检合格的PCR产物送样进行高通量测序(诺禾致源生物信息科技有限公司,北京)。将原始下机的测序数据进行如下处理:(1)按照条码序列和引物序列对原始下机的数据进行各样本数据的拆分,并使用FLASH截取掉条码序列后进行每个样本序列标签的拼接得到每个样本的原始序列数据;(2)序列数据截取和过滤的具体操作如下:从连续低质量值的碱基数到设定长度的第一个低质量的碱基位截断;再将截取后的序列数据集中碱基长度小于总序列长度的75%的序列过滤掉;得到的序列在去除嵌合体后进一步在97%的一致性水平上进行OTU的聚类;最后使用QIIME(1.9.1)进行alpha多样性指数的计算[30-31]。相关原始测序数据已经上传到NCBI(http://www.ncbi.nlm.nih.gov),序列登记编号为PRJNA728144和PRJNA837682。
1.6 细菌群落多样性特征统计分析进行方差齐性检验后,使用单因素方差分析(One-way ANOVA)检验非冰封期和冰封期水体理化参数的差异显著性[32]。非冰封期与冰封期细菌alpha多样性指数的差异使用非参数Wilcoxon检验。另外,计算环境因子和不同时期alpha多样性指数的Spearman相关关系,确定不同时期细菌群落alpha多样性指数的影响因子。基于Bray-Curtis距离的主坐标分析(principal coordinate analysis)来解析湖泊细菌群落结构在不同时期的差异,并使用基于Bray-Curtis相似性指数的相似性分析(analysis of similarity)检验细菌群落结构在非冰封期和冰封期之间差异的显著性[33]。基于DCA(detrended correspondence analysis)的分析结果,当DCA第1轴的特征向量值(DCA1)小于3时,选择RDA(redundancy analysis)分析;当DCA1值介于3~4之间时,选择RDA和CCA(canonical correspondence analysis)均可;当DCA1大于4时,选择CCA分析。基于OTU相对丰度数据和VIF(variance inflation factor)小于10的环境因子进行RDA和CCA分析[33]。使用vegan包中“envfit”函数对影响细菌群落结构的环境因子进行显著性检验。使用环境因子的欧式矩阵和细菌群落结构的Bray-Curtis相似指数矩阵进行Mantel和偏Mantel检验,分析水体理化因子和地理因子对不同时期湖泊细菌群落结构的影响。基于OTU丰度表,构建中性模型分析中性过程对不同时期湖泊细菌群落建构的贡献[5]。另外,基于OTU丰度表及其系统发育树,使用Picante包进一步量化确定性过程(异向选择和同向选择)和随机性过程(扩散限制和同向扩散)对不同时期细菌群落构建的贡献比例[3, 34-35]。零模型是基于β-NTI和RCbray指数来量化确定性过程和随机性过程对细菌群落构建的贡献,当β-NTI小于-2时,认为细菌群落构建过程是同向选择;当β-NTI大于2时,认为细菌群落构建过程是异向选择。另外,当β-NTI介于-2和2之间时,认为细菌群落构建过程为随机性过程并根据RCbray指数的大小,来判断扩散限制(RCbray>0.95)、同向扩散(RCbray < -0.95)和不确定过程(-0.95 < RCbray < 0.95)(如:低扩散和低选择)对细菌群落构建过程的贡献[12, 14]。上述所有的分析过程及可视化均在R(v.4.0.5)中进行。
2 结果分析 2.1 湖泊水体理化参数的变化高原湖泊在非冰封期和冰封期15个水体的理化因子特征见附表Ⅰ。托素湖水温在不同时期的均值最大(8.05℃,非冰封期=12.15,冰封期=3.95)。可鲁克湖在非冰封期和冰封期溶解氧浓度的均值最高(9.76 mg/L,非冰封期=6.79,冰封期=12.73)。5个湖泊的水体非冰封期pH值均小于冰封期,均表现出碱性。因为补给水源、湖泊面积大小和水文条件的差异,不同湖泊的盐度差异较大,其中尕海的平均盐度最高(69 g/L),可鲁克湖最低(3.17 g/L)。受到周围旅游业和畜牧业的影响,青海湖不同时期叶绿素a和总磷的平均浓度明显高于其他湖泊。在营养盐方面,尕海总氮和硝态氮的浓度在非冰封期和冰封期的变化量最大,托素湖最小。此外,非冰封期5个采样湖泊的平均水温、pH、溶解氧、氧化还原电位、浊度、叶绿素a和总磷与冰封期有显著的差异,而电导率、总溶解固体、盐度、溶解有机碳、硝态氮、亚硝态氮、氨氮在不同时期水体中无明显差异。
2.2 细菌群落特征的变化 2.2.1 不同时期alpha多样性指数的比较采样湖泊所有样点水体在非冰封期和冰封期共检测出的OTUs数目分别为46147和55209。进一步计算并分析不同时期细菌群落的alpha多样性指数(Shannon和Simpson多样性指数、Chao1丰度指数以及PD系统发育指数)的差异性及其与环境因子的Spearman相关性,结果如附表Ⅱ、图 2和图 3所示。非冰封期和冰封期细菌群落的平均Chao1丰度指数分别为2342.69和3026.69,非冰封期细菌Chao1丰度指数显著低于冰封期(Wilcoxon,P < 0.01)。非冰封期细菌群落的平均Simpson指数(0.92)显著低于冰封期(0.94)。非冰封期细菌群落Shannon指数为6.29,冰封期为6.81,非冰封期和冰封期并无显著差异。非冰封期细菌群落平均PD指数略大于冰封期,与冰封期细菌群落PD指数并无显著差异。非冰封期细菌Chao1丰度指数和Simpson多样性显著低于冰封期,表明高原湖泊细菌群落丰度和多样性存在显著的季节分布特征。另外,环境因子与不同时期alpha多样性指数的相关关系结果表明,非冰封期细菌多样性随着水温、氧化还原电位、溶解有机碳和总氮的增加而显著增加(图 3,P < 0.05),而冰封期细菌多样性随着溶解有机碳和总氮的增加显著减少。非冰封期细菌Chao1丰度指数与经度、海拔、pH、氧化还原电位、叶绿素a和氨氮呈显著正相关,与海拔呈显著负相关。冰封期细菌Chao1丰度指数与水温和溶解氧呈显著正相关,与电导率、总溶解固体、盐度、总氮和总磷呈显著负相关。
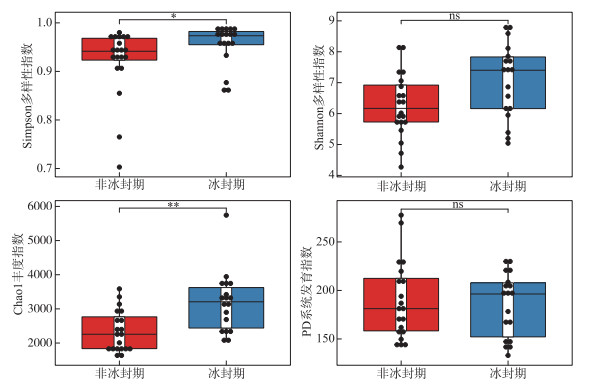
|
图 2 非冰封期和冰封期细菌群落alpha多样性指数对比分析(*代表非冰封期和冰封期细菌alpha多样性指数的Wilcoxon检验P < 0.05,**代表P < 0.01,ns代表差异不显著) Fig.2 Comparative analysis of alpha diversity indices in bacterial communities between non-freezing and freezing periods |
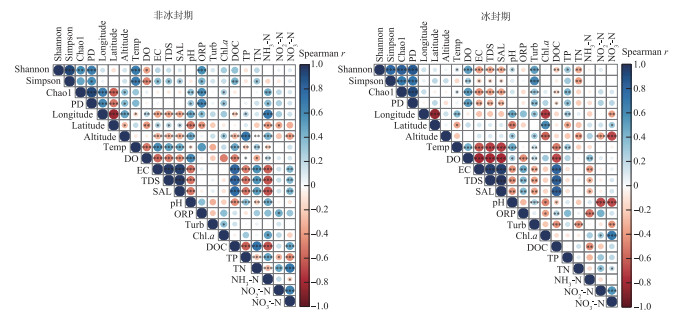
|
图 3 非冰封期和冰封期细菌alpha多样性指数与环境因子之间Spearman相关系数(*代表环境因子与细菌alpha多样性指数的Spearman相关系数的显著性P < 0.05,**代表P < 0.01,***代表P < 0.001) Fig.3 Spearman coefficient between alpha diversity indices of bacterial in non-freezing and freezing periods and environmental factors |
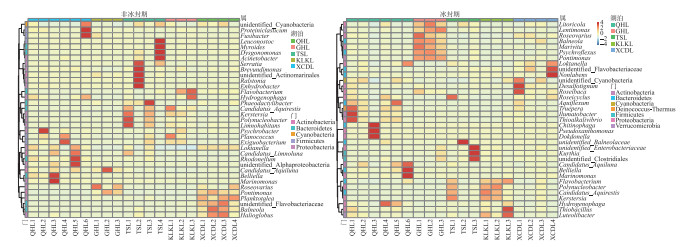
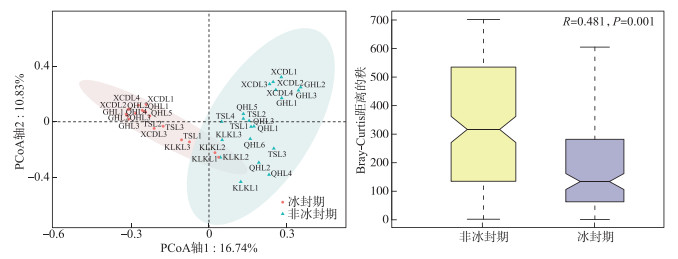
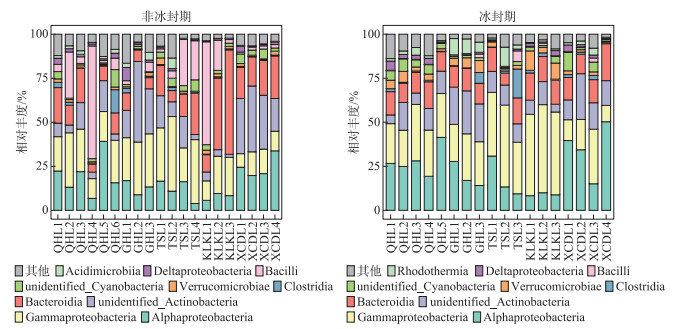
在不同的分类水平上(门和属),高原湖泊非冰封期水体细菌物种组成及其相对丰度存在明显差异。非冰封期和冰封期的优势菌均为变形菌门(Proteobacteria,非冰封期=40.06%,冰封期=56.31%),但在冰封期其平均相对丰度比例更高。然而,非冰封期细菌群落中的拟杆菌门(Bacteroidetes-18.51%)、放线菌门(Actinobacteria-17.71%)和厚壁菌门(Firmicutes-14.21%)的相对丰度均明显的高于冰封期Bacteroidetes(15.81%)、Actinobacteria(14.15%)和Firmicutes(3.03%)(图 4)。这表明了在门分类水平上,细菌群落组成具有季节分布特征。在属水平上,非冰冻期和冰冻期湖泊水体中优势细菌也表现出极为明显的差异。其中非冰封期细菌属主要为Proteiniclasticum、Fusibacter、Leuconostoc、Myroides、Dysgonomonas、Flavobacterium等,冰封期的主要为Nonlabens、Desulfotignum、Roseibaca、Chitinophagadunha、Pseudoxanthomonas等(图 5),此外,基于Bray-Curtis距离的PCoA结果表明,前两个轴对不同时期细菌群落变化的解释率为27.57%(图 6)。来自同一时期的细菌群落基本聚集在一起,而非冰封期和冰封期细菌群落出现了明显的分离,表明不同时期细菌的群落结构不同。同时,不同时期细菌群落结构相似性分析结果表明非冰封期和冰封期湖泊细菌群落组成差异极显著(R=0.481,P=0.001)(图 6)。
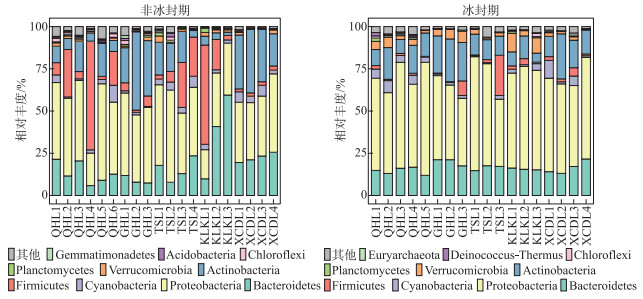
|
图 4 不同样点非冰封期和冰封期前10门相对丰度堆积图 Fig.4 Plot of relative abundance of top 10 phyla in different sampling sites in non-freezing and freezing periods |
|
图 5 不同样点非冰封期和冰封期细菌前35个属相对丰度聚类热图 Fig.5 Top 35 genera heatmaps of bacterial community in different sampling sites in non-freezing and freezing periods |
|
图 6 基于Bray-Curtis距离的不同时期细菌群落结构的主坐标分析(椭圆代表 95%的置信圈)和细菌群落结构的相似性分析 Fig.6 Principal coordinate analysis of bacterial community based on the Bray-Curtis distance (ellipse represent 95% confidence internal) and analysis of similarity of bacterial community in different periods |
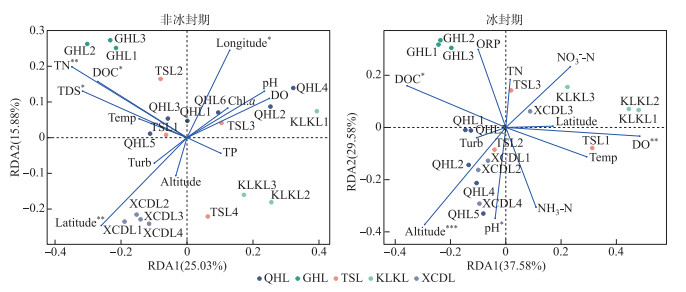
DCA结果(非冰封期DCA1=1.82,冰封期DCA1=1.64)表明RDA分析可以合理解析不同时期细菌群落的驱动因子。非冰封期和冰封期细菌群落结构与环境因子的RDA的结果如图 7所示。从图中可以看出,对采样湖泊非冰封期细菌群落结构影响显著(P < 0.05)的环境因子分别为:总氮>纬度>总溶解固体>经度>溶解有机碳,而显著影响冰封期细菌群落结构的环境因子分别为:海拔>溶解氧>溶解有机碳>pH。非冰封期和冰封期的RDA前两轴分别解释了细菌群落结构总变化的40.91%和67.16%。Mantel和偏Mantel检验结果显示高原湖泊非冰封期和冰封期细菌群落结构相似性均与环境因子显著相关,且水体理化因子对非冰封期细菌群落结构的影响比冰封期更明显。然而,地理因子仅对冰封期细菌群落结构有显著影响(表 1)。
|
图 7 非冰封期和冰封期细菌群落结构的冗余分析(带*的环境因子代表“envfit”函数检验P < 0.05) Fig.7 Redundancy analysis of bacterial community structure in non-freezing and freezing periods (environmental factors with * represent P < 0.05 using function of "envfit"test) |
| 表 1 不同时期细菌群落结构(Bray-Curtis distance)与环境因子(Euclidean distance)之间关系的相关和偏相关检验 Tab. 1 Mantel and partial Mantel tests for relationship between bacterial community structures (Bray-Curtis distance) and environmental factors (Euclidean distance) in different periods |
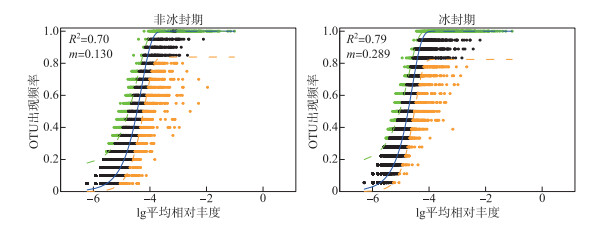
使用基于中性理论的中性模型来拟合不同OTUs的出现频率,探究采样湖泊水体细菌群落在不同时期分布特征差异的驱动机制。中性模型评估结果如图 8所示,非冰封期和冰封期细菌OTUs的出现频率和它们相对丰度变化之间关系的70%和79%可以被中性过程所评估,表明中性过程对冰封期湖泊细菌的构建过程贡献更大。结果也表明在不同时期,与确定性过程相比,细菌种类的损失和增加之间的平衡过程在高海拔湖泊中占据了重要的地位。另外,非冰封期和冰封期细菌的迁移率m分别为0.130和0.289,这表明非冰封期湖泊细菌物种扩散能力低于冰封期。进一步基于零模型,量化了确定性过程和随机性过程对采样湖泊细菌群落结构的相对贡献比例,结果如图 9所示。确定性和随机性过程共同驱动青藏高原湖泊细菌的季节变化。非冰封期细菌群落的构建过程有同向选择(贡献率=32.63%)、异向选择(贡献率=0.53%)、同向扩散(贡献率=1.58%)、扩散限制(贡献率=54.74%)、不确定的过程(贡献率=10.53%)。冰封期细菌群落的构建过程主要由随机性的扩散限制过程(贡献率=55.56%)所主导,确定性过程(同向选择和异向选择)对冰封期细菌群落构建的贡献比例为34.50%。
|
图 8 基于中性模型的非冰封期和冰封期OTU出现频率图(蓝色实线表示中性模型的拟合结果,绿色和橘色的虚线分别表示在95%的置信区间上和下的预测结果,绿色和橙色的点代表OTU的出现频率大于或者小于模型预测的频率) Fig.8 The occurrence frequency of the OTU in non-freezing and freezing periods based on the neutral model (blue solid lines denote the fitting results of neutral model, green and orange dash lines denote the results of the model prediction of the upper and lower of the 95% confidence interval, respectively. Green and orange dots represent the occurrence frequency of OTU with more or less than that in the prediction of neutral model) |
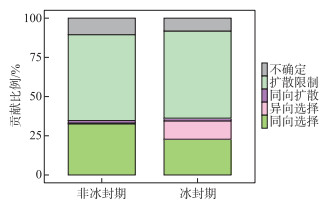
|
图 9 不同时期细菌群落的构建过程 Fig.9 Processes in assembly of bacterial communities in different periods |
本研究结果表明不同采样湖泊和同一湖泊不同样点的水体理化因子差异较大。青海湖、尕海、托素湖和小柴旦湖是咸水湖,可鲁克湖是淡水湖。值得注意的是,可鲁克湖KLKL1样点的盐度与KLKL2和KLKL3样点存在较大差异。这主要是由于KLKL1样点最接近尾闾咸水湖(托素湖)。青海湖QHL2、QHL5和QHL6样点水体叶绿素a和总氮浓度明显高于其他3个样点,青海湖水体理化参数的空间分异主要是由于入湖补给水源的差异[36]。根据前人研究结果,QHL2和QHL6样点处的入湖河泉吉河和黑马河总氮浓度超标,QHL5的入湖河倒淌河化学需氧量超标[36],这与湖边污泥中有机氮浓度较高和当地牧民生活废水入湖有关[37]。
微生物alpha多样性指数是反映微生物数量和群落结构特征的重要指标[21]。本研究中发现青藏高原北部湖泊细菌Chao1丰度指数和Simpson多样性指数在非冰封期和冰封期表现出显著不同,并且非冰封期低于冰封期,说明冰封期采样湖泊水体中细菌的物种较为丰富,群落结构更为复杂。这与先前报道的暖季细菌群落高于冷季的结论不一致[38-39]。根据一般生存理论,在更极端(寒冷)的环境条件下湖泊水体细菌的数量一般会偏少,群落结构趋于简单,而本研究结果表明非冰封期细菌群落丰度和多样性低于冰封期,可能存在如下解释:(1)高原湖泊细菌本身具备抵抗极端寒冷生境的能力,随着冰封期温度的降低,耐低温细菌的生态位可用性增加[40];(2)冰封期更均匀的细菌分布(非冰封期和冰封期Pielou's均匀度指数分别为0.56和0.61),使得冰封期细菌多样性高于非冰封期;(3)可能是湖泊冰封期间人类和动物活动影响相对较小所致[41]。一般而言,人类活动的影响会导致湖水中营养物浓度增加和藻类的大量繁殖,导致生境中溶解氧浓度降低,使得好氧细菌丰度减少。2020年,Wang等研究人类活动影响白洋淀浅湖中微生物群落,发现了强烈的人类活动使湖泊微生物的Shannon和Simpson指数显著降低[40]。Wang等研究发现旅游业发展能够减少国家湿地公园细菌群落结构的alpha多样性但是增加beta多样性[32]。另外,已有研究结果发现青海湖入湖河流中微生物alpha多样性指数显著高于湖泊水体本身,因此,非冰封期采样湖泊水体细菌多样性低于冰封期,并非是非冰封期入湖地表水的稀释所致[41]。
在非冰封期和冰封期采样湖泊水体中细菌的优势门都为Proteobacteria,与杨渐[42]得出的青藏高原湖泊微生物群落多样性中Proteobacteria为优势门的结论一致。研究表明,Proteobacteria在水环境中广泛分布,主要有Alpha-, Beta-, Gamma-, Delta-, Epsilon-proteobacteria纲[24]。然而,Proteobacteria在非冰封期湖泊中平均相对丰度明显低于冰封期,主要是由于Alpha-和Gamma-变形菌纲在非冰封期明显低于冰封期(附图Ⅰ),这与Jiang等研究水库Proteobacteria在秋季和冬季高于春季和夏季的结果一致[43]。同样地,Firmicutes在非冰封期相对丰度为14.21%,而在冰封期为3.03%。已有研究表明Firmicutes可以作为粪便污染指标[39],因此其差异性可能与非冰封期湖泊周边动物活动频繁有关。另外,PCoA结果说明高原湖泊非冰封期和冰封期细菌群落结构具有显著差异。这种差异可能是由于两个时期环境因素和水文条件不同造成的。如非冰封期和冰封期水温、溶解氧等理化参数存在显著差异,而水温和溶解氧被证实是湖泊细菌群落变化的显著驱动因子[32, 35]。
大量研究结果表明湖泊水体盐度、pH、水温、总氮和海拔,是湖泊细菌群落特征差异的显著驱动因子[3, 44-45]。Ji等[46]研究西藏区域25个湖泊水体的细菌多样性,结果显示盐度是解释盐湖细菌群落变化的主要因素,但是在淡水湖细菌群落特征受水温和空间因子的影响显著,总氮对湖泊细菌群落变化的影响不显著,仅解释了细菌群落变化的1.2%。本文的研究结果与其存在一定的差异。一般而言,总氮为细菌生长和代谢过程提供必须的营养和能量,影响了细菌的丰度和群落结构。出现这种差异一方面可能与本研究中与总氮正相关的Actinobacteria相对丰度较大有关[39];另一方面,是研究区域尺度大小不同所致。当研究区域尺度较大,湖泊之间环境因子梯度(如:盐度和水温)和空间因子梯度差异过大,会影响细菌群落结构。海拔是冰封期高原湖泊细菌群落的最显著驱动因子,海拔与冰冻期湖泊水体中叶绿素a和营养盐(总氮、硝态氮和亚硝态氮)呈显著负相关(图 3,P < 0.05)。因此,我们推测海拔对冰冻期湖泊水体细菌群落结构的影响是通过改变细菌限制营养元素来驱动细菌群落的变化。另外,溶解氧同样对冰封期高原湖泊细菌有显著的驱动作用,这可能是由于其驱动好氧细菌、兼性厌氧细菌和厌氧细菌比例的变化,从而改变细菌群落结构。冰封期水体中溶解氧浓度的变化与水温呈显著正相关(图 3,P < 0.05),说明当水温降低,水体中溶解氧浓度随之减少,致使偏好厌氧生境的Gamma-proteobacteria相对丰度增加[23],这也在一定程度上说明了Gamma-proteobacteria纲相对丰度在不同时期的差异。
除了环境因子的变化对采样湖泊水体浮游细菌群落特征产生影响外,中性模型模拟其他过程对细菌群落分布的影响程度,结果表明采样湖泊非冰封期细菌群落组成变化的70%可以被中性过程所解释,而冰封期79%可以被中性过程解释,指示了随机性过程在塑造高原湖泊细菌群落构建中的重要作用。然而冰封期比非冰封期拟合程度高,表明冰封期细菌群落的中性过程更强或者扩散潜力更大,虽然冰封期水体的连通性降低,可能会使冰封期细菌迁移率降低,但在相似的元群落结构下,微生物高扩散潜力不仅与连通性有关,还与生境的同质性有关[5]。本研究中高原湖泊冰封期水体更均匀的物理化学条件更有利于中性过程,较大的环境梯度使得环境因子对微生物群落的影响更大[5]。这一点也可以被偏Mantel检验水体理化因子对非冰封期细菌群落的影响更大的结果所证实(表 1)。因此,采样湖泊生境的同质性对细菌群落的迁移率要大于水体的连通性,因而表现出冰封期细菌群落中性过程更强(扩散潜力更大)。另外,中性理论认为物种的丰度越高,其扩散潜力越大[4],这与本研究采样湖泊冰封期细菌群落Chao1丰度指数高于非冰封期的结论一致。然而,随机模型并不能100%的解释采样湖泊细菌非冰封期和冰封期的群落构建机制,这表明采样湖泊细菌群落的构建在整体上受到多种过程(如同向选择、异向选择、低扩散以及低环境选择)的驱动[10]。
4 附录附表Ⅰ、Ⅱ和附图Ⅰ见电子版(DOI: 10.18307/2023.0121)。
| 附表Ⅰ 湖泊非冰封期和冰封期不同样点环境因子统计 Appendix Ⅰ Environmental factors in different sampling sites of each lake in non-freezing and freezing periods |
| 附表Ⅱ 湖泊非冰封期和冰封期不同样点细菌多样性指数 Appendix Ⅱ The diversity indices of lake bacterial in sampling sites of each lake in non-freezing and freezing periods |
|
附图Ⅰ 不同样点非冰封期和冰封期前10纲相对丰度堆积图 AttachedFigI Plot of relative abundance of top 10 classes in different sampling sites in non-freezing and freezing periods |
| [1] |
杜丁丁. 中国西部地区湖泊碳库效应的影响因素及评价[学位论文]. 兰州: 兰州大学, 2018.
|
| [2] |
Xie GJ, Tang XM, Shao KQ et al. Spatiotemporal patterns and environmental drivers of total and active bacterial abundances in Lake Taihu, China. Ecological Indicators, 2020, 114: 106335. DOI:10.1016/j.ecolind.2020.106335 |
| [3] |
Gu ZQ, Liu KS, Pedersen MW et al. Community assembly processes underlying the temporal dynamics of glacial stream and lake bacterial communities. Science of the Total Environment, 2021, 761: 143178. DOI:10.1016/j.scitotenv.2020.143178 |
| [4] |
Roguet A, Laigle GS, Therial C et al. Neutral community model explains the bacterial community assembly in freshwater lakes. FEMS Microbiology Ecology, 2015, 91(11): fiv125. DOI:10.1093/femsec/fiv125 |
| [5] |
Chen WD, Ren KX, Isabwe A et al. Correction to: Stochastic processes shape microeukaryotic community assembly in a subtropical river across wet and dry seasons. Microbiome, 2019, 7: 148. DOI:10.1186/s40168-019-0749-8 |
| [6] |
Mo YY, Peng F, Gao XF et al. Low shifts in salinity determined assembly processes and network stability of microeukaryotic plankton communities in a subtropical urban reservoir. Microbiome, 2021, 9(1): 128. DOI:10.1186/s40168-021-01079-w |
| [7] |
Sloan WT, Lunn M, Woodcock S et al. Quantifying the roles of immigration and chance in shaping prokaryote community structure. Environmental Microbiology, 2006, 8(4): 732-740. DOI:10.1111/j.1462-2920.2005.00956.x |
| [8] |
Hubbell SP. The unified neutral theory of biodiversity and biogeography. Princeton: Princeton University Press, 2001: 448.
|
| [9] |
Woodcock S, van der Gast CJ, Bell T et al. Neutral assembly of bacterial communities. FEMS Microbiology Ecology, 2007, 62(2): 171-180. DOI:10.1111/j.1574-6941.2007.00379.x |
| [10] |
Liu T, Zhang AN, Wang JW et al. Integrated biogeography of planktonic and sedimentary bacterial communities in the Yangtze River. Microbiome, 2018, 6(1): 16. DOI:10.1186/s40168-017-0388-x |
| [11] |
Logares R, Lindström ES, Langenheder S et al. Biogeography of bacterial communities exposed to progressive long-term environmental change. The ISME Journal, 2013, 7(5): 937-948. DOI:10.1038/ismej.2012.168 |
| [12] |
Stegen JC, Lin XJ, Fredrickson JK et al. Estimating and mapping ecological processes influencing microbial community assembly. Frontiers in Microbiology, 2015, 6: 370. DOI:10.3389/fmicb.2015.00370 |
| [13] |
Wang JJ, Shen J, Wu YC et al. Phylogenetic beta diversity in bacterial assemblages across ecosystems: Deterministic versus stochastic processes. The ISME Journal, 2013, 7(7): 1310-1321. DOI:10.1038/ismej.2013.30 |
| [14] |
Li Y, Gao Y, Zhang WL et al. Homogeneous selection dominates the microbial community assembly in the sediment of the Three Gorges Reservoir. Science of the Total Environment, 2019, 690: 50-60. DOI:10.1016/j.scitotenv.2019.07.014 |
| [15] |
Gad M, Hou LY, Li JW et al. Distinct mechanisms underlying the assembly of microeukaryotic generalists and specialists in an anthropogenically impacted river. Science of the Total Environment, 2020, 748: 141434. DOI:10.1016/j.scitotenv.2020.141434 |
| [16] |
Liu KS, Liu YQ, Hu AY et al. Different community assembly mechanisms underlie similar biogeography of bacteria and microeukaryotes in Tibetan lakes. FEMS Microbiology Ecology, 2020, 96(6): fiaa071. DOI:10.1093/femsec/fiaa071 |
| [17] |
Lv L, Zhang TB, Yi GH et al. Changes of lake areas and its response to the climatic factors in Tibetan Plateau since 2000. J Lake Sci, 2019, 31(2): 573-589. [闾利, 张廷斌, 易桂花等. 2000年以来青藏高原湖泊面积变化与气候要素的响应关系. 湖泊科学, 2019, 31(2): 573-589. DOI:10.18307/2019.0225] |
| [18] |
刘文. 青藏高原北部湖泊有机质构成与微生物降解过程及其对盐度响应的研究[学位论文]. 武汉: 中国地质大学, 2020.
|
| [19] |
Li M, Guo J, Shi ZG et al. Bacterial community structure in lakes on the northeastern Qinghai-Tibetan Plateau. Chinese Journal Applied Environmental Biology, 2013, 19(5): 750-758. [李明, 郭嘉, 石正国等. 春季青藏高原东北部湖泊细菌种类组成. 应用与环境生物学报, 2013, 19(5): 750-758. DOI:10.3724/SP.J.1145.2013.00750] |
| [20] |
Liu XB, Yao TD, Kang SC et al. Bacterial community of the largest oligosaline lake, Namco on the Tibetan Plateau. Geomicrobiology Journal, 2010, 27(8): 669-682. DOI:10.1080/01490450903528000 |
| [21] |
Zhang HG, Li L, Zhao Y et al. Seasonal changes of culturable microbes in different altitude lakes in Qinghai-Tibet Plateau. Journal of Traditional Chinese Veterinary Medicine, 2013, 32(3): 49-55. [张红光, 李琳, 赵燕等. 青藏高原不同海拔湖水中可培养微生物的季节性变化. 中兽医医药杂志, 2013, 32(3): 49-55.] |
| [22] |
李国强. 应用高通量测序技术分析青藏高原湖水中微生物多样性[学位论文]. 兰州: 兰州交通大学, 2016.
|
| [23] |
Wu QL, Zwart G, Schauer M et al. Bacterioplankton community composition along a salinity gradient of sixteen high-mountain lakes located on the Tibetan Plateau, China. Applied and Environmental Microbiology, 2006, 72(8): 5478-5485. DOI:10.1128/AEM.00767-06 |
| [24] |
Xing P, Hahn MW, Wu QL. Low taxon richness of bacterioplankton in high-altitude lakes of the eastern Tibetan Plateau, with a predominance of Bacteroidetes and Synechococcus spp. Applied and Environmental Microbiology, 2009, 75(22): 7017-7025. DOI:10.1128/AEM.01544-09 |
| [25] |
Dietze E, Hartmann K, Diekmann B et al. An end-member algorithm for deciphering modern detrital processes from lake sediments of Lake Donggi Cona, NE Tibetan Plateau, China. Sedimentary Geology, 2012, 243/244: 169-180. DOI:10.1016/j.sedgeo.2011.09.014 |
| [26] |
Yang YF, Jiang H, Niu FJ et al. The space-time variation analyses of air temperature over the Qinghai-Xizang Plateau in warm and cold seasons. Plateau Meteorology, 2007, 26(3): 496-502. [杨瑜峰, 江灏, 牛富俊等. 青藏高原暖季与冷季气温的时空演变分析. 高原气象, 2007, 26(3): 496-502. DOI:10.3321/j.issn:1000-0534.2007.03.009] |
| [27] |
Shen L, Yao TD, Xu BQ et al. Variation of culturable bacteria along depth in the East Rongbuk ice core, Mt. Everest. Geoscience Frontiers, 2012, 3(3): 327-334. DOI:10.1016/j.gsf.2011.12.013 |
| [28] |
Walters W, Hyde ER, Berg-Lyons D et al. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys. mSystems, 2015, 1(1): e00009-e00015. DOI:10.1128/mSystems.00009-15 |
| [29] |
Wang DD, Lin Q, Mahmut O et al. Diversity of airborne bacterial communities in sandstorm area of Taklimakan. Acta Microbiologica Sinica, 2018, 58(8): 1420-1430. [王丹丹, 林青, 欧提库尔·玛合木提等. 塔克拉玛干沙尘暴源区空气细菌群落多样性. 微生物学报, 2018, 58(8): 1420-1430.] |
| [30] |
Caporaso JG, Kuczynski J, Stombaugh J et al. QⅡME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [31] |
Caporaso JG, Lauber CL, Walters WA et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. The ISME Journal, 2012, 6(8): 1621-1624. DOI:10.1038/ismej.2012.8 |
| [32] |
Wang BH, Zheng XF, Zhang HJ et al. Bacterial community responses to tourism development in the Xixi National Wetland Park, China. Science of the Total Environment, 2020, 720: 137570. DOI:10.1016/j.scitotenv.2020.137570 |
| [33] |
Zeng J, Jiao CC, Zhao DY et al. Patterns and assembly processes of planktonic and sedimentary bacterial community differ along a trophic gradient in freshwater lakes. Ecological Indicators, 2019, 106: 105491. DOI:10.1016/j.ecolind.2019.105491 |
| [34] |
Stegen JC, Lin XJ, Konopka AE et al. Stochastic and deterministic assembly processes in subsurface microbial communities. The ISME Journal, 2012, 6(9): 1653-1664. DOI:10.1038/ismej.2012.22 |
| [35] |
He Q, Wang S, Hou WG et al. Temperature and microbial interactions drive the deterministic assembly processes in sediments of hot springs. Science of the Total Environment, 2021, 772: 145465. DOI:10.1016/j.scitotenv.2021.145465 |
| [36] |
Zhang XJ, Xu ZM, Song XY et al. Application and study of several water quality evaluation method in rivers flowing into Qinghai Lake. Environmental Engineering, 2013, 31(1): 117-121. [张小君, 徐中民, 宋晓谕等. 几种水环境质量评价方法在青海湖入湖河流中的应用. 环境工程, 2013, 31(1): 117-121.] |
| [37] |
Zhang K, Lan JH, Shen ZX et al. The chemical composition and quality evaluation of surface water in Qinghai Lake areas. Journal of Earth Environment, 2010, 1(3): 162-168. [张琨, 蓝江湖, 沈振兴等. 青海湖流域水化学分析及水质初步评价. 地球环境学报, 2010, 1(3): 162-168.] |
| [38] |
Shang YQ, Wu XY, Wang XB et al. Factors affecting seasonal variation of microbial community structure in Hulun Lake, China. Science of the Total Environment, 2022, 805: 150294. DOI:10.1016/j.scitotenv.2021.150294 |
| [39] |
Zhang L, Fang WK, Li XC et al. Linking bacterial community shifts with changes in the dissolved organic matter pool in a eutrophic lake. Science of the Total Environment, 2020, 719: 137387. DOI:10.1016/j.scitotenv.2020.137387 |
| [40] |
Wang WJ, Yi YJ, Yang YF et al. Impact of anthropogenic activities on the sediment microbial communities of Baiyangdian shallow lake. International Journal of Sediment Research, 2020, 35(2): 180-192. DOI:10.1016/j.ijsrc.2019.10.006 |
| [41] |
Ren Z, Wang F, Qu XD et al. Taxonomic and functional differences between microbial communities in Qinghai Lake and its input streams. Frontiers in Microbiology, 2017, 8: 2319. DOI:10.3389/fmicb.2017.02319 |
| [42] |
杨渐. 青藏高原湖泊微生物群落演替及其环境指示意义[学位论文]. 武汉: 中国地质大学, 2015.
|
| [43] |
Jiang TT, Sun SN, Chen YN et al. Microbial diversity characteristics and the influence of environmental factors in a large drinking-water source. Science of the Total Environment, 2021, 769: 144698. DOI:10.1016/j.scitotenv.2020.144698 |
| [44] |
Yang J, Ma LA, Jiang HC et al. Salinity shapes microbial diversity and community structure in surface sediments of the Qinghai-Tibetan Lakes. Scientific Reports, 2016, 6: 25078. DOI:10.1038/srep25078 |
| [45] |
Zhong ZP, Liu Y, Miao LL et al. Prokaryotic community structure driven by salinity and ionic concentrations in plateau lakes of the Tibetan Plateau. Applied and Environmental Microbiology, 2016, 82(6): 1846-1858. DOI:10.1128/AEM.03332-15 |
| [46] |
Ji MK, Kong WD, Yue LY et al. Salinity reduces bacterial diversity, but increases network complexity in Tibetan Plateau lakes. FEMS Microbiology Ecology, 2019, 95(12): fiz190. DOI:10.1093/femsec/fiz190 |
 2023, Vol. 35
2023, Vol. 35 


