(2: 中国科学院南京地理与湖泊研究所, 湖泊与环境国家重点实验室, 南京 210008)
(3: 襄阳职业技术学院, 襄阳 441050)
(4: 滨州市水文中心, 滨州 256600)
(2: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
(3: Xiangyang Polytechnic, Xiangyang 441050, P. R. China)
(4: Binzhou Hydrological Center, Binzhou 256600, P. R. China)
浮游细菌(Bacterioplankton)是湖泊生态系统中的重要组成部分,具有极高的个体数量和复杂的物种、遗传多样性[1]。其不仅在湖泊有机质矿化、营养盐循环及能量流动中发挥重要作用,而且对湖泊富营养化等环境问题具有指示性[2-4]。揭示湖泊中浮游细菌的空间格局及其驱动机制,对深刻认识湖泊中的生物地球化学循环过程具有重要意义[5],同时也能为湖泊生态系统健康的维持提供理论参考[6]。
早期学者认为,自然生境中细菌等微生物因其巨大的个体数量与频繁的扩散作用,因此在空间上具有全球分布的特征[7-8],并提出了“Everything is everywhere, but, the environment selects”的经典假说[9]。尽管,该假说中环境对微生物分布进行选择的论断已被普遍接受[10],但前半句,即微生物呈现全球性分布的论断正遭到越来越多的挑战[1, 11-12]。事实上,很多研究发现湖泊等生境中的微生物与大型动植物一样,其组成及相对丰度等普遍呈现显著的空间差异性[1, 5, 13]。但是,也有学者通过模拟试验发现,相互连通的网格状水体景观中,微生物群落结构趋于均质化[14]。而存在无序水文连通的湖泊之间,浮游细菌群落的空间差异性也较低[6]。浅水湖泊即具备水文连通的特性,又具有水动力扰动强烈的生境特点[15-16],但其中浮游细菌是否具有空间差异仍然没有定论。
湖泊中浮游细菌空间格局的形成机制是微生物生物地理学关注的核心问题之一[12]。微生物的空间格局受到多种因素控制,除了局部环境因素外,历史进化因素(如地理距离、扩散历史等)也对微生物的空间分布产生重要影响[10, 13]。基于此,Martiny等[13]提出主导微生物空间格局的四种生态理论假设,用以探讨环境因子与地理距离对微生物空间格局的相对贡献。近年来,有学者发现经典的生态位理论与中性理论同样适用于微生物空间格局形成机制的研究[6, 17],并以此提出了影响微生物空间格局的两个生态学过程,即确定性过程(deterministic processes)与随机过程(stochastic processes)[10]。确定性过程是基于生态位理论(niche theory)而提出的,该过程强调生物群落构建是由物种自身特性、种间关系以及环境状况控制的[18]。随机过程则是以中性理论(neutral theory)为基础的生态学概念,反映的是历史偶然性、生态漂移和群落随机形成与灭绝等在生物群落构建过程中的主导作用[10]。研究表明,不同湖泊中浮游细菌的群落构建过程因地而异[6, 19],这与水体扰动、栖息地的连通性、生境大小、空间尺度,甚至采样布点方式等多种因素有关[5, 20]。浅水湖泊中强烈的风浪扰动不但会影响微生物的空间扩散,而且还会影响环境变量的空间异质性[16]。此生境条件下,浮游细菌空间格局的驱动机制尚不明确[1, 21]。
太湖是世界上典型的大型浅水湖泊,其浮游细菌群落的空间分布规律及其影响因素也得到很多学者的关注。以往的研究通过在太湖不同湖区或者沿入湖河流进行针对性采样,普遍发现太湖中水体细菌丰度、群落结构在空间上存在显著的空间差异性,且受到水环境因子的显著影响[3, 22]。也有学者针对太湖个别湖区研究水体细菌的群落结构及其影响因素[23-24],发现随机过程在东太湖浮游细菌的群落构建中发挥关键作用。但总体而言,太湖全湖浮游细菌群落的空间格局及其驱动机制仍未明晰。本研究通过对太湖全湖进行均匀布点,系统调查太湖浮游细菌的空间分布格局,分析环境因子与空间因子对浮游细菌群落的影响,定量评估确定性过程与随机过程对浮游细菌空间格局的各自贡献率,为深刻认识浅水湖泊中微生物空间格局的形成过程提供理论依据。
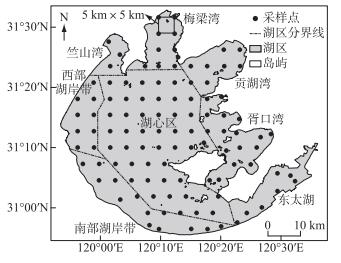
1 材料和方法 1.1 采样点布设、样品采集及预处理参照水利部太湖流域管理局发布的《太湖健康状况报告》(以2018年为例)[25],将太湖划分为8个湖区(图 1)。利用地理信息系统(ArcGIS 10.5),将整个太湖按照每25 km2划分一个网格,在每个网格的四个节点设置采样点,共布设111个样点(图 1)。样品采集于2013年9月4-6日进行,采样过程中通过便携式GPS进行样点定位,在每个样点用采水器采集表层(0~50 cm)水样约5 L,置于事先用酒精灭菌的聚丙烯采样瓶中,带回至太湖湖泊生态系统研究站实验室进行样品处理。
|
图 1 太湖采样点分布 Fig.1 Location of sampling sites in Lake Taihu |
首先,将各水样摇匀后用GF/F滤膜(直径为47 mm,孔径0.45 μm)过滤,滤膜上的样品用于分析水体中的叶绿素a (Chl.a),滤后水用于分析可溶性有机碳(DOC);用烧过的GF/C滤膜过滤一定体积的水样用于分析其中的总悬浮物浓度(TSS)。然后,将各个水样充分摇匀后取300 mL加入到用酒精灭菌的滤瓶中,用0.22 μm孔径的聚碳酸酯滤膜(直径为47 mm,Millipore, Billerica, MA, USA) 过滤;将各滤膜置于1.5 mL的灭菌离心管中,存放至-80℃超低温冰箱中,用于后续进行浮游细菌的16S rRNA基因分析。
1.2 水体理化性质分析及浮游细菌DNA测序在采样过程中,现场用透明度盘测量水体透明度(SDD), 用声呐测深仪测量各样点水体深度(WD), 用多参数水质分析仪(YSI 6600 V2, Yellow Springs Instruments, USA)测量水体温度(WT)、pH、溶解氧(DO)、电导率(EC)和矿化度(TDS)。水体总氮(TN)、总磷(TP)、总悬浮物(TSS)、叶绿素a(Chl.a)、溶解性有机碳(DOC)、氨氮(NH3-N)、硝态氮(NO3--N)及磷酸盐(PO43--P)参照《湖泊富营养化调查规范》进行分析[26]。样品中的总DNA用试剂盒(FastDNAⓇ Spin Kit for Soil)提取,然后用微量分光光度计(NanoDrop ND-1000 UV/Vis spectral photometer)检验DNA提取的纯度。将符合扩增、测序标准的样品做好标记后各取20 μL,选择细菌核糖体16S rRNA基因V4区特异性引物515F (5′-GTGYCAGCMGCCGCGGTAA-3′) 和806R (5′-GGACTACNVGGGTWTCTAAT-3′) 进行扩增[6]。最后,将扩增产物(扩增子)送至BGI公司(中国武汉)在Illumina MiSeq测序平台进行测序。
1.3 数据分析与统计测得的序列通过生态学定量研究工具QIIME2(版本:Core 2020.8)的分析流程进行分析[27],基于SILVA数据库(版本:138)进行物种注释[28],详细分析流程见Tang等[19]的文献所述。在此基础上,对细菌ASV数据进行Hellinger转化降低数据不均产生的影响[29],采用基于Bray-Curtis距离的非度量多维尺度排序分析方法(NMDS),对太湖浮游细菌的空间格局进行分析,各湖区间浮游细菌群落结构的差异性通过R语言(版本:3.6.4)[30] “vegan”包[31]中的置换多因素方差分析(Adonis)功能进行检验。
使用R语言“geosphere”包中的方法根据各样点的经纬度计算两两样点之间的地理距离。然后通过“vegan”包中的vegdist函数计算样点之间细菌群落间物种组成的相异度Bray-curtis距离指数以及环境因子的差异性欧氏距离(Euclidean)指数。利用斯皮尔曼等级相关(Spearman Rank Correlation)分析细菌群落相似指数(相似指数=100-相异度指数)、环境因子相异度指数与地理距离的关系。此外,通过Mantel检验分析细菌群落、环境因子与地理距离之间相关性的显著性,并分析细菌群落相似指数与各个环境因子之间相关性的显著性。
通过邻体矩阵主坐标分析(Principal coordinates of neighbor matrices, PCNMs)的方法得到空间变量的不同组分(PCNM)及线性趋势(Linear trend)[32]。在此基础上通过前向选择对浮游细菌群落结构具有显著影响的环境与空间变量[33]。由于去趋势对应分析(detrended correspondence analysis, DCA)发现浮游细菌群落排序的最长轴 < 3.0(2.7),故选用冗余分析(redundancy analysis, RDA)分析环境与空间变量对浮游细菌群落变化的贡献率[34]。通过变差分析(variation partitioning approach, VPA)检验环境变量和空间因子整体在构建浮游细菌群落中的相对重要性。
浮游细菌空间格局的驱动机制利用Sloan等[35]开发的中性群落模型(Neutral community model, NCM)和Stegen等[36-37]开发的基于零模型(Null-model-based model)的计算方法进行分析。通过NCM评估随机过程对细菌群落空间格局形成的潜在影响。而零模型则用于定量评估确定性过程与随机过程对浮游细菌空间格局的贡献率,并且通过模型中的beta最邻近指数(beta-nearest taxon index, βNTI)和基于Bray-Curtis的Raup Crick距离(Bray-Curtis based Raup-Crick, RCbray)进一步确定控制细菌空间分布的生态学过程。当|βNTI| > 2时表明细菌群落构建受到选择作用控制,其中βNTI > 2时由异质化选择(Heterogeneous selection)控制,βNTI < -2时由同质化选择(Homogeneous selection)控制;当|βNTI| < 2时表明细菌群落构建不受选择作用控制,此时与RCbray距离指数结合判断细菌群落的构建机制。其中,|βNTI| < 2且RCbray > 0.95时群落构建受到扩散限制(Dispersal limitation)主导;|βNTI| < 2且RCbray < -0.95时受到同质化扩散(Homogenizing dispersal)主导;|βNTI| < 2且|RCbray| < 0.95时则受到非主导过程(Undominated)的控制[36]。此外,本研究用R软件进行数据统计分析及研究结果可视化[30],研究中的图件用Adobe illustrator CC 2019软件进行优化。
1.4 基因序列上传信息本研究中涉及到的细菌原始序列已上传到国际生物技术信息中心(NCBI)基因数据库(https://www.ncbi.nlm.nih.gov/),上传的序列号为:SRR17477860。
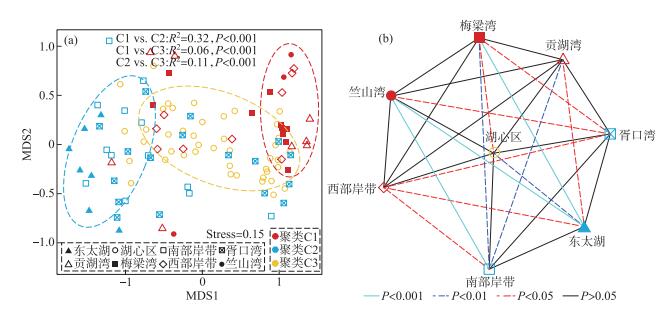
2 结果与分析 2.1 太湖浮游细菌的空间格局NMDS分析表明,太湖西北部湖区(C1:梅梁湾、竺山湾、贡湖湾及西部岸带)与东南湖区(C2:东太湖、胥口湾及南部岸带)之间浮游细菌群落结构存在显著差异(图 2a, R2=0.36, P < 0.001),尤其是东太湖与梅梁湾、竺山湾之间及竺山湾与南部岸带之间差异性最为显著(图 2b, P < 0.001);此外,湖心区(C3)的浮游细菌群落结构与西北部湖区和东南部湖区在整体上存在显著差异,但相对差异性较弱(图 2a, C1 vs. C3, R2=0.06, C2 vs. C3, R2=0.11),湖心区浮游细菌群落结构与贡湖湾、西部岸带、南部岸带等多数湖区间的差异性不显著(图 2b, P>0.05)。
|
图 2 太湖浮游细菌的空间格局(a)及湖区间群落差异性检验(b) Fig.2 Geographic patterns of bacterioplankton in Lake Taihu (a) and the significance test of difference of bacterioplankton community among each lake region(b) |
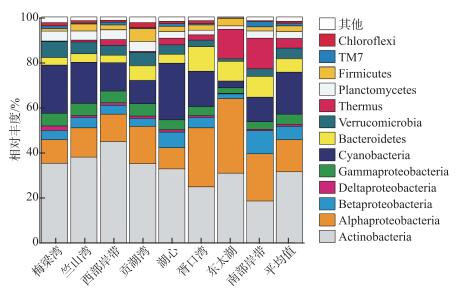
太湖浮游细菌优势门类(平均相对丰度>1 %)包括放线菌(Actinobacteria)、变形菌(Proteobacteria)、蓝细菌(Cyanobacteria)、拟杆菌(Bacteroidetes)、疣微菌(Verrucomicrobia)、栖热菌(Thermus)、浮霉菌(Planctomycetes)、厚壁菌(Firmicutes)等,其各自平均相对丰度分别为:31.56 %、25.85 %、18.63 %、5.92 %、4.80 %、4.16 %、3.18 %、2.41 %。而Proteobacteria中Alphaproteobacteria(14.11 %)、Betaproteobacteria(5.84 %)、Gammaproteobacteria(4.41 %)和Deltaproteobacteria(1.00 %)为主要类群。不同湖区间浮游细菌主要门类的平均相对丰度存在一定差异,Actinobacteria、Cyanobacteria和Verrucomicrobia在西部岸带、竺山湾、梅梁湾平均丰度较高,而在南部岸带、胥口湾和东太湖平均丰度相对较低;Alphaproteobacteria、Thermus和Bacteroidetes则呈现相反的趋势(图 3)。
|
图 3 太湖各湖区主要浮游细菌门类的相对丰度对比 Fig.3 Comparison of the major bacterioplankton phyla across different lake regions in Lake Taihu |
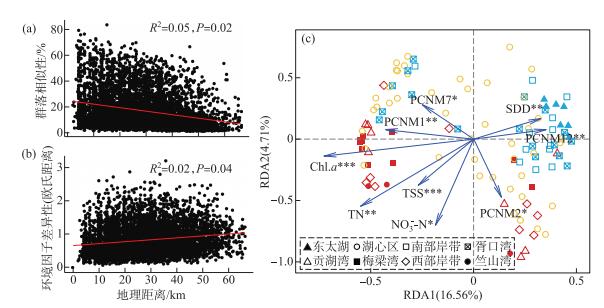
回归分析显示,尽管浮游细菌群落相似性随着地理距离的增加显著降低(P=0.02),但是两者的相关性较弱(R2=0.05),说明太湖中浮游细菌群落结构呈现微弱的距离衰减规律(图 4a),地理距离对太湖浮游细菌空间分布格局的影响可能较小。此外,调查期间太湖表层水体的平均WT、pH、EC、TDS、DO和SDD分别为(26.05±1.58)℃、(8.72±0.36)、(580.90±53.37) μS/cm、(0.37±0.03) mg/L、(8.72±1.25) mg/L和(0.40±0.24) m;而全湖氮、磷营养盐浓度的分析显示,TN、NH3-N及NO3--N的平均浓度分别为(1.47±1.20)、(0.07±0.08)和(0.16±0.27) mg/L,TP和PO43--P的平均浓度分别为(0.11±0.10)和(0.02±0.04) mg/L;此外,TSS、Chl.a及DOC的平均浓度分别为(56.19±36.55)、(44.85±80.58)、(5.13±0.74) mg/L。空间上,太湖表层水体中环境因子的空间差异性较小(图 4b, R2=0.02, P=0.04)。冗余分析显示,太湖中浮游细菌群落结构与5个环境因子(TSS、SDD、Chl.a、TN及NO3--N)、4个空间变量(PCNM1、PCNM2、PCNM7及PCNM12)显著相关。而且,Chl.a与西北部几个湖区(C1,梅梁湾、竺山湾、西部岸带)的浮游细菌群落结构显著正相关(P < 0.001);SDD与东南部几个湖区(C2,东太湖、胥口湾、南部岸带) 的浮游细菌群落结构显著正相关(P < 0.001);而TSS则与湖心区(C3) 的浮游细菌群落结构显著正相关(P < 0.001)(图 4c)。
|
图 4 浮游细菌群落相似性和环境因子差异性与地理距离之间的相关性(a, b),浮游细菌群落组成与对其产生显著影响的环境变量和空间变量之间的关系(c)(*表示P < 0.05、* *表示P < 0.01、* * *表示P < 0.001) Fig.4 The distance-decay relationship of bacterioplankton community similarity(a) and environmental dissimilarity (b), the bacterial community composition in relation to significant local environmental variables and regional geographical factors (c)(*: P < 0.05, * *: P < 0.01, * * *: P < 0.001) |
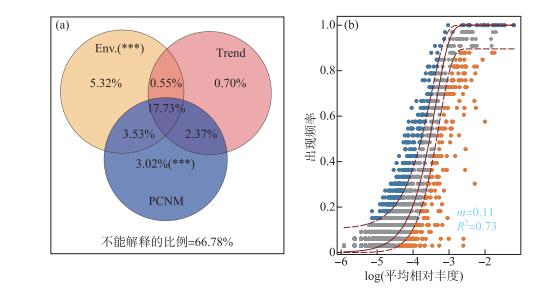
研究发现,环境变量(Evn.)和空间变量(包括线性趋势(Linear trend)和PCNM变量)与浮游细菌的空间格局显著相关。而环境变量及空间变量可以解释33.22 % 的浮游细菌群落的变化(图 5a)。其中,纯环境变量和纯空间变量的解释率分别为5.32 %、3.02 %,而这两者共同解释的比率为17.73 %,说明二者之间存在较强的交互作用。另外,有高达66.78 % 的细菌群落的变化不能被以上三者解释,说明太湖中浮游细菌的空间格局受到更为复杂的因素影响(图 5a)。此外,中性群落模型对太湖浮游细菌群落的整体拟合优度较高(R2=0.73),表明太湖浮游细菌的空间格局受随机性过程的影响较大(图 5b)。
|
图 5 太湖浮游细菌群落格局的驱动因素(a)及中性群落模型拟合结果(b) (*表示P < 0.001) Fig.5 Drivers of bacterial community composition in Lake Taihu (a) and the fit of the neutral community model (NCM) of community assembly (b)(*: P < 0.001) |
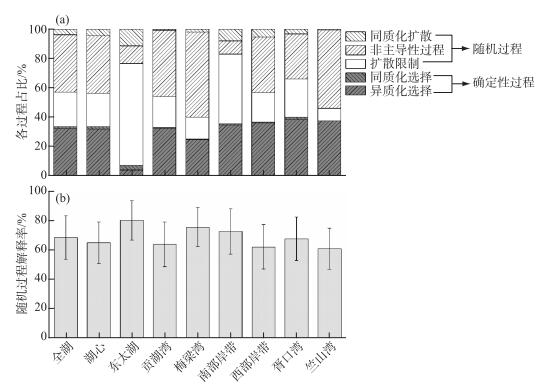
零模型的分析结果显示,太湖浮游细菌的空间格局受到多个生态学过程的共同影响。其中,异质化选择、非主导性过程及扩散限制这3种作用的影响较大,且总体上受到随机过程主导(图 6a),其总解释率为68.52 % ±14.84 % (图 6b)。各湖区间,控制浮游细菌空间分布的生态学过程也存在差异,例如:东太湖与南部岸带水体浮游细菌空间分布受到扩散限制主导,而异质化选择作用对胥口湾浮游细菌的空间分布影响较大,梅梁湾和竺山湾中浮游细菌空间分布则受非主导性过程的影响较大(图 6a)。此外,随机过程对各个湖区浮游细菌群落结构空间变化的解释率均超过50 % (图 6b)。这一发现与中性群落模型的分析结果一致,表明太湖表层水体中浮游细菌的空间格局主要受到随机过程主导,且随机过程中的非主导性过程是主导因素。
|
图 6 太湖浮游细菌群落空间格局形成的驱动机制(a)及随机过程的相对贡献率(b) Fig.6 The driving mechanisms of spatial pattern formation of bacterioplankton communities (a) and relative importance of stochastic processes on spatial pattern formation of bacterioplankton communities in Lake Taihu (b) |
有研究表明,在较大空间跨度[1, 6]和存在地理隔离的湖泊间[11-12],以及在即使较小空间的静水池塘内[38],浮游细菌的群落结构均普遍呈现出显著的空间差异性。本研究发现,太湖西北部几个湖区与东南部的几个湖区之间浮游细菌群落结构也存在显著差异(图 2a),而这两个区域内各湖区间的浮游细菌群落结构差异并不显著(图 2b),且湖心区与周边湖区间之间浮游细菌群落结构差异也不显著(图 2b)。Tang等[3]也发现,太湖水体细菌中的颗粒有机物附着细菌和游离细菌群落结构在湖心区与梅梁湾之间及湖心区与东太湖之间的空间差异性较小。太湖浮游细菌的空间格局受到多种因素的影响。
首先,环境因子在太湖浮游细菌群落空间格局的形成中发挥重要作用。本研究发现,环境变量对浮游细菌群落变化的贡献率比空间变量更高(图 5a),且选择过程能够解释较大比例的浮游细菌群落结构的变化(图 6a),这可能主要是受到环境选择作用的影响。结果显示,不同湖区中生境环境存在差异,其中的浮游细菌群落组成也与不同的环境因子密切相关(图 4b)。太湖同时存在草、藻及两者过渡的(湖心)3种生境类型,而草、藻型湖区间浮游细菌群落结构差异性最大,且两者与湖心区浮游细菌的群落结构差异性相对较小(图 2a,图 3),这一发现与滇池的情况一致[39]。这主要是由于草、藻的分解转化速率、过程不同,因此主导这些过程的微生物群落势必存在差异[39-40];而且草、藻分解产物的差异,加之草、藻在代谢物分泌、抵御风浪扰动、物质吸附等方面不同,导致不同生境中水环境因子的差异[41]。太湖西北部湖区是典型的浊水、藻型生境,浮游藻类为主要生产者,Cyanobacteria的相对丰度也较其它湖区更高(图 3)。该湖区中的浮游细菌群落与Chl.a及TN显著相关(图 4b),说明该区域中浮游细菌在藻类分解及氮素循环中发挥重要作用。而东南部湖区则是典型的清水、草型生境,高等水生植被为其中主要的生产者,其中的浮游细菌群落更适应在清水环境下生存,因此与SDD显著相关(图 4b)。此外,太湖中间湖心区为过渡区域,风浪扰动最为强烈[42]。有研究表明,扰动会引起沉积物再悬浮将其中的细菌扩散至水体中,改变浮游细菌的群落结构[43],这也是湖心区浮游细菌群落与TSS显著相关的原因(图 4b)。
其次,太湖浮游细菌群落的空间格局与频繁的水动力扰动有关。有学者通过模拟实验发现,在没有水流方向偏向的连通水体生境中,局部扩散作用会使其中的微生物群落结构趋于均质化[14]。Yuan等[44]也发现,无序的水力扰动能够增加互通水体之间浮游植物群落的相似性。前期的研究也表明,互相连通的邻近湖泊间,频繁无序的水动力扰动也会使其中的浮游细菌群落结构趋于均质化[6]。太湖水深较浅、湖面广阔且处于亚热带季风气候区,在高频的风力推动下各湖区间水动力扰动作用剧烈[15-16, 42]。本研究发现,太湖浮游细菌群落结构呈现出较弱的地理衰减规律(图 4a),说明大部分湖区间浮游细菌受到的扩散限制较弱。这是由于太湖无序的风浪扰动使得水体微生物随着水力混合作用在各个湖区间扩散,从而使邻近湖区间浮游细菌群落结构的差异性减弱。本研究表明,尽管在湖泊微生物地理格局的研究中,“Everything is everywhere, but, the environment selects”这一假设的前半句正不断受到质疑[11, 38],但以太湖为代表的大型浅水湖泊中,强烈风浪扰动下无序的水力混合作用能够促进浮游细菌的空间扩散,从而削弱湖泊中浮游细菌群落结构的空间差异性。本研究表明,在一些相互联通的水体中,若存在充分的水力混合作用,那么整个水体中浮游细菌的群落结构在空间上可能会趋于均匀分布。
此外,太湖浮游细菌的空间格局受到随机过程的强烈影响。本研究发现,环境变量与空间变量对太湖浮游细菌群落的变化解释率并不高,大部分细菌群落的变化受到其他因素的影响(图 5a)。这一现象与一些具有水动力扰动的水体生境中的研究发现类似[6, 17],但与静水生境中的研究发现截然不同[38]。进一步分析显示,太湖浮游细菌的空间格局总体上受到随机过程主导(图 5b)。有研究表明,存在水文连通的水体生境之间,无序的水动力扰动会导致其中的浮游细菌随机聚集[6, 17],这与静水环境截然不同[38]。太湖中的水力混合作用是由风浪扰动引起的,无序的水力混合能够促进浮游细菌的随机扩散[6-17]。本研究结合零模型对驱动太湖浮游细菌群落聚集的生态学过程进行分析。结果显示,确定性过程中的异质化选择(32.20 %)和随机过程中的非主导性过程及扩散限制(62.56 %)是影响太湖浮游细菌空间分布的主要过程。这一结果与中性群落模型的分析结果一致(图 5b),同时也表明局部环境对浮游细菌的空间分布也具有深刻影响。此外,本研究发现不同湖区主导浮游细菌群落聚集的生态学过程也存在差异,如:东太湖与南部岸带中浮游细菌受到更强的扩散限制(图 6a),这可能是由于该区风浪扰动相对较弱[39],导致细菌扩散也较弱。而藻型湖区(梅梁湾和竺山湾等)浮游细菌的群落聚集受到非主导性过程主导,这可能与该区大量的藻类活动密切相关。有研究表明,太湖蓝藻与其他异养细菌之间具有密切的生态关联性[45],而藻类消长周转较快[40],导致与之相关的浮游细菌也具有较快的周转速率[46],这种情况下可能会增加细菌的漂变。而且藻类的空间分布受到风浪扰动的强烈影响,在太湖多变且不稳定的风场下蓝藻在水体中会呈现多变的动态特征[47]。受到藻类空间运移的影响,藻型湖区中浮游细菌的群落聚集也会呈现无序多变的特征。
4 结论1) 太湖中,浮游细菌的群落结构在西北部湖区(藻型湖区:梅梁湾、竺山湾、西部岸带及贡湖湾)和东南部湖区(草型湖区:东太湖、胥口湾及南部岸带)之间存在较大空间差异性,但这两个区域内的湖区之间及湖心区与各邻近湖区之间的差异性并不显著。
2) 局部环境因子与随机过程共同影响了太湖浮游细菌群落的空间格局,而且随机过程起到了主导作用。
3) 太湖多变且不稳定的风浪扰动会促进浮游细菌的随机扩散,导致局部浮游细菌群落的空间差异性降低,也是造成其中的浮游细菌空间格局受随机过程控制的主要原因。
致谢: 感谢薛静琛、龚伊、张磊等老师在野外采样及样品分析过程中给予的帮助。
| [1] |
Liu LM, Yang J, Yu Z et al. The biogeography of abundant and rare bacterioplankton in the lakes and reservoirs of China. The ISME Journal, 2015, 9(9): 2068-2077. DOI:10.1038/ismej.2015.29 |
| [2] |
Hu Y, Xie GJ, Jiang XY et al. The relationships between the free-living and particle-attached bacterial communities in response to elevated eutrophication. Frontiers in Microbiology, 2020, 11: 423. DOI:10.3389/fmicb.2020.00423 |
| [3] |
Tang XM, Chao JY, Gong Y et al. Spatiotemporal dynamics of bacterial community composition in large shallow eutrophic Lake Taihu: High overlap between free-living and particle-attached assemblages. Limnology and Oceanography, 2017, 62(4): 1366-1382. DOI:10.1002/lno.10502 |
| [4] |
Ren LJ, He D, Xing P et al. Bacterial diversity and ecological function in lake water bodies. Biodiversity Science, 2013, 21(4): 421-432. [任丽娟, 何聃, 邢鹏等. 湖泊水体细菌多样性及其生态功能研究进展. 生物多样性, 2013, 21(4): 421-432. DOI:10.3724/SP.J.1003.2013.12052] |
| [5] |
Hanson CA, Fuhrman JA, Horner-Devine MC et al. Beyond biogeographic patterns: Processes shaping the microbial landscape. Nature Reviews Microbiology, 2012, 10(7): 497-506. DOI:10.1038/nrmicro2795 |
| [6] |
Bai CR, Cai J, Zhou L et al. Geographic patterns of bacterioplankton among lakes of the middle and lower reaches of the Yangtze River Basin, China. Applied and Environmental Microbiology, 2020, 86(6): e02423-e02419. DOI:10.1128/AEM.02423-19 |
| [7] |
Finlay BJ, Clarke KJ. Ubiquitous dispersal of microbial species. Nature, 1999, 400(6747): 828. DOI:10.1038/23616 |
| [8] |
Finlay BJ. Global dispersal of free-living microbial eukaryote species. Science, 2002, 296(5570): 1061-1063. DOI:10.1126/science.1070710 |
| [9] |
Baas-Becking LGM. Geobiologie of inleiding tot de milieukunde. Van Stockum & Zoon: The Hague, The Netherlands, 1934.
|
| [10] |
Zhou JZ, Ning DL. Stochastic community assembly: Does it matter in microbial ecology?. Microbiology and Molecular Biology Reviews: MMBR, 2017, 81(4): e00002-e00017. DOI:10.1128/MMBR.00002-17 |
| [11] |
Li HB, Zeng J, Ren LJ et al. Contrasting patterns of diversity of abundant and rare bacterioplankton in freshwater lakes along an elevation gradient. Limnology and Oceanography, 2017, 62(4): 1570-1585. DOI:10.1002/lno.10518 |
| [12] |
Liao JQ, Cao XF, Wang J et al. Similar community assembly mechanisms underlie similar biogeography of rare and abundant bacteria in lakes on Yungui Plateau, China. Limnology and Oceanography, 2017, 62(2): 723-735. DOI:10.1002/lno.10455 |
| [13] |
Martiny JBH, Bohannan BJM, Brown JH et al. Microbial biogeography: Putting microorganisms on the map. Nature Reviews Microbiology, 2006, 4(2): 102-112. DOI:10.1038/nrmicro1341 |
| [14] |
Carrara F, Altermatt F, Rodriguez-Iturbe I et al. Dendritic connectivity controls biodiversity patterns in experimental metacommunities. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(15): 5761-5766. DOI:10.1073/pnas.1119651109 |
| [15] |
Wu TF, Qin BQ, Ma JR et al. Movement of cyanobacterial colonies in a large, shallow and eutrophic lake: A review. Chinese Science Bulletin, 2019, 64(36): 3833-3843. [吴挺峰, 秦伯强, 马健荣等. 浅水富营养化湖泊中蓝藻群体运动研究述评. 科学通报, 2019, 64(36): 3833-3843. DOI:10.1360/TB-2019-0033] |
| [16] |
Qin BQ. Shallow Lake limnology and control of eutrophication in Lake Taihu. J Lake Sci, 2020, 32(5): 1229-1243. [秦伯强. 浅水湖泊湖沼学与太湖富营养化控制研究. 湖泊科学, 2020, 32(5): 1229-1243. DOI:10.18307/2020.0501] |
| [17] |
Mo YY, Zhang WJ, Yang J et al. Biogeographic patterns of abundant and rare bacterioplankton in three subtropical bays resulting from selective and neutral processes. The ISME Journal, 2018, 12(9): 2198-2210. DOI:10.1038/s41396-018-0153-6 |
| [18] |
Fargione JE, Tilman D. Diversity decreases invasion via both sampling and complementarity effects. Ecology Letters, 2005, 8(6): 604-611. DOI:10.1111/j.1461-0248.2005.00753.x |
| [19] |
Tang XM, Xie GJ, Shao KQ et al. Contrast diversity patterns and processes of microbial community assembly in a river-lake continuum across a catchment scale in northwestern China. Environmental Microbiome, 2020, 15(1): 10. DOI:10.1186/s40793-020-00356-9 |
| [20] |
Zhou JZ, Deng Y, Zhang P et al. Stochasticity, succession, and environmental perturbations in a fluidic ecosystem. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(9): 836-845. DOI:10.1073/pnas.1324044111 |
| [21] |
Ofiteru ID, Lunn M, Curtis TP et al. Combined niche and neutral effects in a microbial wastewater treatment community. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(35): 15345-15350. DOI:10.1073/pnas.1000604107 |
| [22] |
Feng S, Qin BQ, Gao G. Response of bacterial communities to eutrophic water in Lake Taihu. Acta Scientiae Circumstantia, 2007, 27(11): 1823-1929. [冯胜, 秦伯强, 高光. 细菌群落结构对水体富营养化的响应. 环境科学学报, 2007, 27(11): 1823-1929.] |
| [23] |
Xue YG, Liu F, Sun M et al. Community structure and influencing factors of bacterioplankton in spring in Zhushan Bay, Lake Taihu. Environmental Science, 2018, 39(3): 1151-1158. [薛银刚, 刘菲, 孙萌等. 太湖竺山湾春季浮游细菌群落结构及影响因素. 环境科学, 2018, 39(3): 1151-1158.] |
| [24] |
Zeng J, Lin YQ, Zhao DY et al. Seasonality overwhelms aquacultural activity in determining the composition and assembly of the bacterial community in Lake Taihu, China. Science of the Total Environment, 2019, 683: 427-435. DOI:10.1016/j.scitotenv.2019.05.256 |
| [25] |
水利部太湖流域管理局. 太湖健康状况报告(2018). 上海: 太湖流域管理, 2019.
|
| [26] |
金相灿, 屠清瑛. 湖泊富营养化调查规范: 第2版. 北京: 中国环境科学出版社, 1990.
|
| [27] |
Bolyen E, Rideout JR, Dillon MR et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nature Biotechnology, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 |
| [28] |
Quast C, Pruesse E, Yilmaz P et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research, 2013, 41(D1): D590-D596. DOI:10.1093/nar/gks1219 |
| [29] |
Legendre P, Gallagher ED. Ecologically meaningful transformations for ordination of species data. Oecologia, 2001, 129(2): 271-280. DOI:10.1007/s004420100716 |
| [30] |
Core R Team. R: A Language and Environment for Statistical Computing. R Foundation for Statistical Computing, 2019.
|
| [31] |
Dixon P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science, 2003, 14(6): 927-930. DOI:10.1111/j.1654-1103.2003.tb02228.x |
| [32] |
Borcard D, Legendre P. All-scale spatial analysis of ecological data by means of principal coordinates of neighbour matrices. Ecological Modelling, 2002, 153(1): 51-68. DOI:10.1016/S0304-3800(01)00501-4 |
| [33] |
Blanchet FG, Legendre P, Borcard D. Forward selection of explanatory variables. Ecology, 2008, 89(9): 2623-2632. DOI:10.1890/07-0986.1 |
| [34] |
Lepš J, Šmilauer P. Multivariate analysis of ecological data using CANOCO. Cambridge: Cambridge University Press, 2003.
|
| [35] |
Sloan WT, Lunn M, Woodcock S et al. Quantifying the roles of immigration and chance in shaping prokaryote community structure. Environmental Microbiology, 2006, 8(4): 732-740. DOI:10.1111/j.1462-2920.2005.00956.x |
| [36] |
Stegen JC, Lin XJ, Konopka AE et al. Stochastic and deterministic assembly processes in subsurface microbial communities. The ISME Journal, 2012, 6(9): 1653-1664. DOI:10.1038/ismej.2012.22 |
| [37] |
Stegen JC, Lin XJ, Fredrickson JK et al. Quantifying community assembly processes and identifying features that impose them. The ISME Journal, 2013, 7(11): 2069-2079. DOI:10.1038/ismej.2013.93 |
| [38] |
Lear G, Bellamy J, Case BS et al. Fine-scale spatial patterns in bacterial community composition and function within freshwater ponds. The ISME Journal, 2014, 8(8): 1715-1726. DOI:10.1038/ismej.2014.21 |
| [39] |
Wang YJ, Li HB, Xing P et al. Contrasting patterns of free-living bacterioplankton diversity in macrophyte-dominated versus phytoplankton blooming regimes in Dianchi Lake, a shallow lake in China. Chinese Journal of Oceanology and Limnology, 2017, 35(2): 336-349. DOI:10.1007/s00343-016-5277-9 |
| [40] |
Wu QL, Xing P, Li HB et al. Impacts of regime shift between phytoplankton and macrophyte on the microbial community structure and its carbon cycling in lakes. Microbiology China, 2013, 40(1): 87-97. [吴庆龙, 邢鹏, 李化炳等. 草藻型稳态转换对湖泊微生物结构及其碳循环功能的影响. 微生物学通报, 2013, 40(1): 87-97.] |
| [41] |
Crump BC, Amaral-Zettler LA, Kling GW. Microbial diversity in Arctic freshwaters is structured by inoculation of microbes from soils. The ISME Journal, 2012, 6(9): 1629-1639. DOI:10.1038/ismej.2012.9 |
| [42] |
Wang Z, Wu TF, Zou H et al. Changes in seasonal characteristics of wind and wave in different regions of Lake Taihu. J Lake Sci, 2016, 28(1): 217-224. [王震, 吴挺峰, 邹华等. 太湖不同湖区风浪的季节变化特征. 湖泊科学, 2016, 28(1): 217-224. DOI:10.18307/2016.0125] |
| [43] |
Shao KQ, Gao G, Tang XM et al. Low resilience of the particle-attached bacterial community in response to frequent wind-wave disturbance in freshwater mesocosms. Microbes and Environments, 2013, 28(4): 450-456. DOI:10.1264/jsme2.ME13032 |
| [44] |
Yuan YX, Jiang M, Liu XT et al. Environmental variables influencing phytoplankton communities in hydrologically connected aquatic habitats in the Lake Xingkai Basin. Ecological Indicators, 2018, 91: 1-12. DOI:10.1016/j.ecolind.2018.03.085 |
| [45] |
Zhao DY, Shen F, Zeng J et al. Network analysis reveals seasonal variation of co-occurrence correlations between cyanobacteria and other bacterioplankton. Science of the Total Environment, 2016, 573: 817-825. DOI:10.1016/j.scitotenv.2016.08.150 |
| [46] |
Gong Y, Tang XM, Shao KQ et al. Dynamics of bacterial abundance and the related environmental factors in large shallow eutrophic Lake Taihu. Journal of Freshwater Ecology, 2017, 32(1): 133-145. DOI:10.1080/02705060.2016.1248506 |
| [47] |
Qin BQ, Yang GJ, Ma JR et al. Dynamics of variability and mechanism of harmful cyanobacteria bloom in Lake Taihu, China. Chinese Science Bulletin, 2016, 61(7): 759-770. DOI:10.1360/N972015-00400 |
 2023, Vol. 35
2023, Vol. 35 


