(2: 中国科学院大学, 北京 100049)
(3: 南京信息工程大学, 南京 210044)
(4: 中国科学院大学中丹学院, 北京 100049)
(5: 中国科学院抚仙湖高原深水湖泊研究站, 玉溪 652500)
(2: University of Chinese Academy of Sciences, Beijing 100049, P.R. China)
(3: Nanjing University of Information Science and Technology, Nanjing 210044, P.R. China)
(4: Sino-Danish College, University of Chinese Academy of Sciences, Beijing 100049, P.R. China)
(5: The Fuxianhu Station of Plateau Deep Lake research, Chinese Academy of Sciences, Yuxi 652500, P.R. China)
微生物群落是生态系统中重要组成部分,在元素循环等方面发挥着重要作用,微生物群落结构和功能变化,将影响生态系统提供正常功能与服务[1-3]。在全球变化背景下,越来越多的研究人员呼吁关注微生物群落对环境变化的响应和稳定性,从而更好地预测生态系统功能和服务的变化趋势[4]。其中,微生物群落稳定性是微生物群落在外界干扰或环境波动下保持不变或相对稳定的能力,它是衡量群落对环境变化响应的重要性质[3]。微生物群落稳定性及相关机制的研究可以帮助理解和预测变化环境下微生物群落结构和功能动态,服务于生态系统保护。
一般认为,环境条件会影响微生物群落的稳定性[2, 5-6]。一方面,高胁迫水平可能限制了微生物群落对干扰的响应[7],而高的营养水平则促进微生物群落对于干扰的响应[8],因为群落需要更多的资源做出调整以更好响应干扰[2, 9]。另一方面,环境条件可能通过对特定微生物的选择,进而影响群落对于干扰的响应[10]。例如,高的营养水平可能会选择富营养型菌(copiotrophs)[11-12],导致群落在面对干扰时更加敏感,但是因为较快的生长速率能够更快恢复到干扰前的生物量[13]。总体来说,环境变化(如营养水平变化),对于群落稳定性的影响可能是复杂的。
盐度是塑造全球微生物群落多样性的重要因子[14],对湖泊浮游微生物群落多样性和功能有着强烈作用[8, 15-17]。同时,全球湖泊可能因为人类活动(如过度水资源开发)或者全球气候变化(水分蒸发速率加快)面临着盐度增加的挑战[18]。在此背景下,浮游细菌群落在盐度干扰下如何维持稳定性具有重要意义。基于此,许多稳定性相关研究将盐度干扰作为淡水微生物群落的模式化干扰进行研究,为后续研究提供了重要借鉴[19-22]。因而,利用盐度干扰研究淡水浮游细菌群落对干扰的响应及稳定性机制是一个比较合适的范式。
群落稳定性虽然有着简单且直观的含义,但是至今仍没有一个统一的度量指标,并且在不同研究之间表现为一个多方面的复杂概念[23-24]。文献计量分析显示,绝大多数研究通过测量1~2个稳定性指标以反映稳定性,忽略了稳定性其多面性的特点[24-25]。为了解群落稳定性的多面性,一些稳定性研究开始尝试采用多个稳定性指标对群落稳定性进行分析[26-28]。这些研究发现不同稳定性指标之间相关性并不高,因此有必要了解多个稳定性指标的高低以综合判断群落的稳定性[25, 27, 29-31]。在以往微生物群落稳定性的研究中,对稳定性的描述通常集中于2个稳定性指标——抵抗力和恢复力[2, 3, 32-33]。抵抗力定义为群落抵抗干扰并使自身在干扰下保持不变的能力;恢复力定义为群落在干扰后恢复到原状的能力[32]。在微生物群落研究当中较少研究抵抗力、恢复力与其他稳定性指标的关系。在反映群落稳定性时,不同稳定性指标的结果是否一致也值得探讨。
本研究设置了2个营养水平(高营养水平和低营养水平)和3个盐度干扰水平(对照组、3‰和9‰),通过较高频次的样品采集,研究浮游细菌群落在盐度干扰后不同阶段群落结构和细胞密度,计算了基于细胞密度的多个稳定性指标(反应力、抵抗力、恢复力和时间稳定性),分析不同稳定性指标之间的相关性,并尝试揭示群落在盐度干扰下潜在的稳定性机制。
1 材料与方法 1.1 装置建立 1.1.1 水样采集本研究中使用的初始浮游细菌群落来自中国江苏省南京市内受人类活动影响相对较小的紫霞湖。紫霞湖(32°3′32.4″~32°3′36.0″N,118°50′34.8″~118°50′38.4″E)的总面积约为5 hm2,位于紫金山中部(海拔448 m)。从紫霞湖表层以下0.5 m处采集水样,作为初始浮游微生物群落。采样时水温接近22.8℃。
1.1.2 初始浮游细菌群落的构建通过真空泵抽取并依次进行1次5 μm孔径的聚碳酸酯滤膜(Millipore, USA)和2次0.8 μm孔径的聚碳酸酯滤膜(Millipore, USA)过滤,移除湖水中的大型浮游生物,得到的水样作为初始浮游细菌群落。大型浮游生物的移除主要为了剔除浮游细菌的捕食者——鞭毛虫和纤毛虫[34],排除捕食者对于浮游细菌群落稳定性的影响。在正式进行后续实验之前,初始浮游细菌群落保存在4℃冰箱冷藏。
1.1.3 不同营养水平设置本实验借助稀释的手段,来操纵群落的营养水平,避免额外添加营养(单一碳源或者氮磷等营养盐)变化带来的α多样性变化。将未过滤的湖水进行高温高压灭菌(121℃,30 min)以杀死湖水中所有生物,作为无菌培养基,用于培养浮游细菌群落。取2 L的0.8 μm滤后水装入3 L锥形瓶中,作为低营养水平组。将200 mL 0.8 μm滤后水加到装有1.8 L灭菌湖水的3 L锥形瓶中,作为高营养水平组。每个营养水平包含9个平行装置。值得注意的是,高营养水平组包含原位水体全部的有机碳(包含颗粒物),而低营养水平组仅包含水体中能通过0.8 μm孔径的有机碳。
1.1.4 培养条件所有瓶子在完成上述步骤以后,在超净台中用可以透气但不能通过微生物的半透膜覆盖,并置于23℃(与采样时水温相近)的黑暗环境下。每天上午和下午各摇晃一次,以促进氧气在水中的溶解。在正式添加干扰之前,设置了8天的适应期,让浮游细菌群落适应生长环境。
1.2 添加干扰通过文献调研,在研究浮游细菌群落的盐度强度-稳定性关系时,一些研究均使用了3‰盐度干扰[20, 35]并且Hu等的研究结果指出3‰盐度干扰是盐度-多样性关系的临界点[19],因此本文选用3‰盐度干扰作为其中一个干扰水平。而预实验表明,6‰比3‰盐度干扰对细胞密度差异不显著,因此最终选用了9‰和3‰盐度干扰研究不同干扰强度对于群落稳定性的影响。
用超纯水配置30、90 g/L的氯化钠溶液,并进行高温高压灭菌(121℃,20 min),用于后续模拟盐度干扰。在预培养8天以后,随机将同一稀释水平的9个锥形瓶分成3组:对照组、3‰盐度干扰组和9‰盐度干扰组,随后,分别加入200 mL灭菌后的超纯水、30 g/L氯化钠溶液(终浓度3‰)、90 g/L氯化钠液(9‰)。充分混合以后,放回暗处培养。盐度干扰的添加以及后续水样采集工作均在无菌工作台中完成,并在完成操作以后立刻用半透膜覆盖,以避免外界微生物扩散进入体系。阻断外界微生物扩散进入体系中,可以避免扩散引发的随机定殖导致群落的强烈波动[36]。
1.3 指标收集与测量 1.3.1 细胞计数在添加干扰前,干扰后6、24、48 h以及4、6、8、10、12、14和16 d,将每个装置混匀后,使用移液枪吸取3 mL的水样装入5 mL无菌离心管中。随后,添加40% 甲醛进行固定,使甲醛终浓度为2%,随后置于4℃冰箱中保存,直至实验结束后统一测量(最长保存时间为20 d)。细胞密度的测量采用Gong等的方法[37](cells/mL)。
1.3.2 DNA收集、提取和测序在实验干扰前(加入无菌水之前)以及干扰后的第1、2、4、8、16天对浮游细菌群落结构进行测定。除了16 d以外的其他时间点,从3个平行中各取200 mL的水样,混合后(总体积600 mL)配合0.2 μm聚碳酸酯滤膜(Millipore, USA)进行抽滤。在第16天的时候,分别从每一个平行取600 mL的水样,配合0.2 μm聚碳酸酯滤膜(Millipore, USA)进行抽滤。所有滤膜都保存在-20℃下至DNA提取。将滤膜剪碎以后,根据土壤DNA提取试剂盒(Mo Bio Laboratories, Carlsbad, CA, USA)说明书提取DNA。使用靶向16S rRNA基因V4可变区(515F-806R)的扩增子[38]对原核生物多样性进行调查,对DNA进行PCR以后,根据标准协议在Illumina MiSeq平台(上海凌恩生物科技有限公司)上对扩增子库进行双端测序(2×250 bp)。测序下机数据存放在美国国家生物信息中心(National Center for Biotechnology Information)中,登录号为PRJNA987196。
1.3.3 基础水化指标紫霞湖原位湖水测得的水化指标为:总磷浓度为0.052 mg/L,可溶性总磷为0.018 mg/L;总氮浓度为3.75 mg/L,可溶性总氮为3.40 mg/L;总有机碳为35.67 mg/L,可溶性有机碳为24.00 mg/L。在模拟实验中,取0.2 μm滤膜的滤后液装在棕色瓶中,并冷冻在-20℃环境直至测量。测量时,使用总有机碳分析仪(TOC-V CPN, Shimadzu, Japan)测定水样中的溶解性有机碳(mg/L)。
1.4 稳定性定义与计算参考White等[28]和Hillebrand等[27]对于稳定性的定义,本研究使用4个稳定性指标(图 1):反应力(reactivity)、抵抗力(resistance)、恢复力(resilience)和时间稳定性(temporal stability)。在稳定性的计算过程中,使用细胞密度的自然对数效应值(log-response ratio, LRR)对群落对干扰处理的响应进行定量化[27-28]。
| LRR=ln(XTreatment ˉXControl ) | (1) |
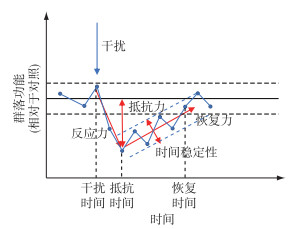
|
图 1 基于干扰过程的稳定性不同维度的定义 (其中虚线表示对照处理下功能的置信区间。此处假设当群落不受到干扰时,群落功能将维持在对照组的置信区间内) Fig.1 The definition of different dimensions of stability based on a disturbance process (The dash line represents the confidence interval of function under control group. Here we hypothesize that if community was not disturbed, the community function would have remained within the confidence interval of the control group) |
式中,XTreatment表示处理组的细胞密度,而XControl表示与对照组同一时间点对照组的细胞密度的平均值,ln表示以自然常数为底的对数。此处假设,处理组的群落如果没有受到干扰时,会与同一时间的处理组有着一致的功能表现,而不是维持在初始状态[27, 28]。因此,LRR反映了干扰处理导致了细胞密度变化。
4个稳定性指标总体来说描述了群落响应干扰不同过程,并且所有稳定性的数值越大表示群落稳定性越强。反应力表示群落放大干扰的能力,数学上为干扰后到达最大值过程中的斜率[28]。抵抗力表示群落抵抗干扰后变化的能力,数学上表示在干扰以后的最大变幅。抵抗力对应时间点为了方便后续表述,本文称之为抵抗时间。观察到群落恢复到正常水平(对照组置信区间范围内)的时间点为恢复时间[28];如果群落在观测结束时,没有恢复到对照水平,则取最后一次观测时间作为恢复时间。恢复力表示群落恢复到原来状态的快慢,数学上表示为抵抗时间和恢复时间之间的斜率;时间稳定性表示群落在恢复过程中的变异程度,数学上表示为抵抗时间和恢复时间之间残差的方差的倒数[27]。我们计算了每一个装置的反应力、抵抗力、恢复力和时间稳定性。值得注意的是,理论上,不同稳定性指标是独立的,因为它们描述了稳定性不同过程[30]。详细的计算代码可见附件1。
1.5 数据分析 1.5.1 扩增子分析使用R统计软件中“DADA2”包构建16S rRNA基因扩增子序列的单核苷酸突变(amplicon sequence variants,ASV)丰度表[39]。使用Cutadapter分别对左右两端序列切除引物[40]。随后进行质量控制时,并设置允许最大的预期错误(maxEE参数)为2。将左右两端序列合并,生成单核苷酸突变丰度表,并除去嵌合体。随后使用“DADA2”内置的朴素贝叶斯分类算法[41]比对核糖体数据库计划(Ribosomal Database Project)的训练集v18(trainset 18)[42],对获得的ASV序列进行注释,其中置信阈值为80%。将第16天中同一处理下的序列进行混合用于后续分析。将所有样品抽平至22609条序列进行后续分析。随后,我们使用ASV数目反映群落的物种丰富度,并计算了处理组相对于对照组的物种丰富度自然对数效应值(见公式(1));使用群落之间Bray-Curtis不相似度反映群落结构的变化。借助R统计软件中的“vegan”包,使用非度量多维排列(non-metric multidimensional scaling,NMDS)对群落结构变化进行降维,使用相似性分析(analysis of similarity, ANOSIM)对于不同营养水平以及不同干扰处理之间群落结构差异进行检验。借助R统计软件中的“VennDiagram”包计算两个营养水平下群落的特有物种和共有物种,并绘制韦恩图。
1.5.2 统计分析借助R统计软件(http://www.r-project.org)中的“lmerTest”包分别对不同营养水平下干扰强度对于细胞密度的作用进行混合线性模型的构建,其中设置干扰处理为确定效应(fixed effect),采样时间点为随机效应(random effect),并对参数进行估计。在分析不同处理(营养水平和干扰强度)对于稳定性的影响时,恢复力和时间稳定性在不同处理之间的方差并不一致,因此统一选用Scheirer-Ray-Hare检验进行非参数的双因素方差检验[43],借助R统计软件中的“rcompanion”包实现该检验。
除了特殊说明以外,本文所有图片均由R统计软件中的“ggplot2”和“ggpubr”包进行绘制。
2 结果 2.1 不同营养水平群落差异经过8 d的预培养后,高营养水平组中的溶解性有机碳((7.7±1.04) mg/L)显著高于低营养水平组((4.89±0.53) mg/L)(P=0.014)。营养水平影响了浮游细菌群落细胞密度:在对照处理下高营养水平下的浮游细菌群落的细胞密度要高于低营养水平群落(图 2a;P < 0.001)。因此,高营养水平通过更高的可利用物质,维持了更高的浮游细菌细胞密度。
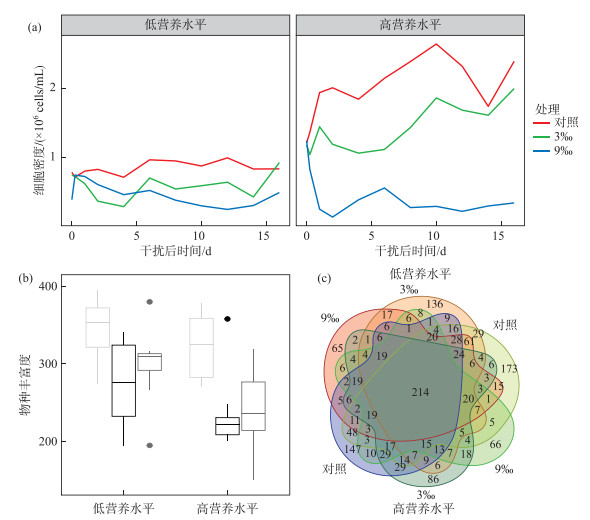
|
图 2 不同处理下浮游细菌群落的(a)细胞密度、(b)物种丰富度和(c)特有物种和共有物种 Fig.2 The (a) cell density, (b) species richness, and (c) unique and sharing species of bacterioplankton community under different treatments |
在多样性方面,对照处理下的高营养水平群落和低营养水平群落有着相近的物种丰富度(图 2b;P=0.21)。高营养水平群落和低营养水平群落在所有处理下的γ多样性也相近(图 2c)。总体来说,使用稀释的方法避免营养水平增加导致的物种丰富度降低,因此排除了营养水平通过物种丰富度变化影响群落的稳定性。同时,营养水平显著地改变群落结构(ANOSIM:P < 0.001)。高营养水平群落中γ变形菌纲(Gammaproteobacteria)的相对丰度更高,而低营养水平群落中疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)和浮霉菌门(Planctomycetes)的相对丰度更高(图 3a)。
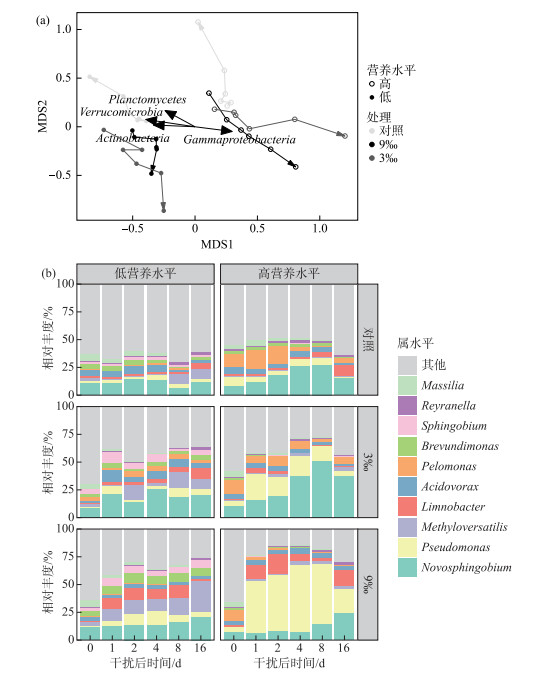
|
图 3 不同盐度干扰和营养水平下浮游细菌群落结构和优势属随时间变化 (其中(a)包含箭头的折线表示群落结构随时间变化的路径) Fig.3 Changes of community structure composition and dominant genus of bacterioplankton with time under different salinity disturbance intensities and nutrient levels ((a)The broken line containing an arrow represents the path of community structure dynamic over time) |
干扰添加后,处理组的浮游细菌群落细胞密度相对于对照组显著降低(图 4a),由此可见浮游细菌群落对盐度干扰敏感。在干扰后,随时间增加部分群落的细胞密度恢复到了对照组水平(与对照组无显著差异),而部分群落在实验结束时仍没有恢复(图 4a)。借助混合线性模型发现,无论是高营养水平群落还是低营养水平群落,9‰盐度干扰比3‰造成了更大的细胞密度损失(高营养水平:P < 0.001;低营养水平:P=0.046)。低营养水平群落在9‰盐度干扰下的细胞密度损失在干扰后0~4 d要小于3‰,反映了群落动态的复杂性。与干扰后细胞密度降低相对应,可以发现在处理组中溶解性有机碳相对于对照处理组在干扰后迅速增加,随着干扰后时间的增加而降低。
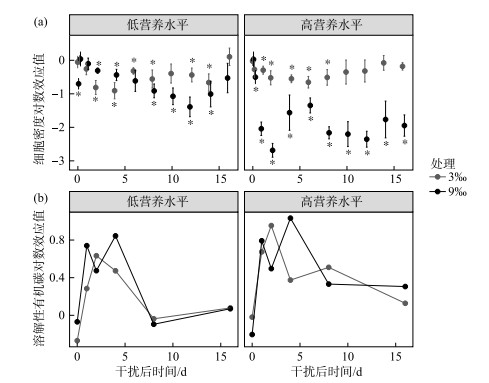
|
图 4 干扰前后不同时间(a)浮游细菌群落细胞密度(平均值±标准差)和(b)水体中溶解性有机碳相对于同一时间对照组的变化(基于对数效应值) Fig.4 The change of (a) cell density of bacterioplankton community (mean±standard deviation) and (b) dissolved organic carbon in water samples relative to the control group on the same census before and after disturbance (based on log-response ratio) |
无论是高营养水平和低营养水平群落中,不同强度的干扰均造成了浮游细菌群落物种丰富度的降低(图 2b;图 5a)。由此可以推测部分浮游细菌并不能够适应升高的盐度环境。浮游细菌群落结构在不同干扰下相对于对照组均发生了显著的变化(ANOSIM: P < 0.001;图 3)。沿着干扰后时间,干扰组和对照组之间的浮游细菌群落结构Bray-Curtis不相似度增加,即群落结构没有表现出恢复(图 5b)。
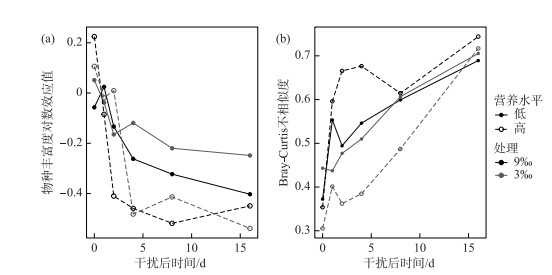
|
图 5 浮游细菌群落物种丰富度(基于对数效应值)和群落结构(基于Bray-Curtis不相似度) 相对于同一时间对照组的变化 Fig.5 The species richness change (based on log-response ratio) and community structure shift (Bray-Curtis dissimilarity) of bacterioplankton communities relative to the control group on the same census |
浮游细菌群落中,不同主要门或纲对于干扰的响应并不一致(图 6):γ变形菌纲的相对丰度在干扰后普遍增加;拟杆菌门(Bacteroidetes)、浮霉菌门和疣微菌门的相对丰度在干扰后普遍下降。部分细菌门类对于在不同营养水平和干扰强度下,响应不一致:β变形菌纲(Betaproteobacteria)的相对丰度在低营养水平群落中增加,但是在高营养水平群落中下降;α变形菌纲(Alphaproteobacteria)的相对丰度仅在9‰盐度干扰下的高营养水平群落中增加;而放线菌门(Actinobacteria)的相对丰度仅在9‰盐度干扰下的低营养水平群落中维持一个较高的水平。相似的,在属水平上,不同类群也表现出不同的响应(图 3b)。其中,Pseudomonas、Methyloversatilis、Limnobacter和Sphingobium等属的丰度在盐度干扰后增加;Pelomonas属的丰度在盐度干扰后减少(图 3b)。
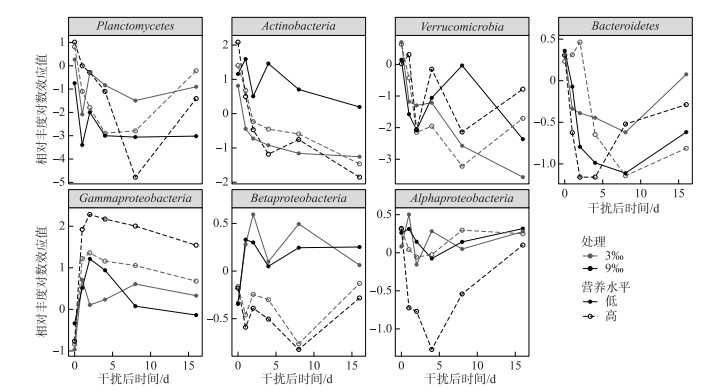
|
图 6 浮游细菌群落中优势门和纲相对丰度相对于同一时间对照组的变化(基于自然对数效应值) (变形菌门拆分成α-、β-和γ-变形菌纲) Fig.6 The change (based on log-response ratio) of relative abundance for abundant phyla/classes relative in bacterioplankton communities to control group on the same census (The phylum Proteobacteria is separated into α-, β-and γ-Proteobacteria) |
基于对照组和处理组之间细胞密度差异,计算了反应力、抵抗力、恢复力和时间稳定性描述浮游细菌群落在盐度干扰下不同过程的稳定性,其中4个稳定性数值越大,表示稳定性越高。高营养水平群落在9‰盐度干扰下稳定性最低(图 7)。其中,除了抵抗力和恢复力之间斯皮尔曼相关性不显著以外,稳定性的不同指标均表现出了较高的相关性(表 1)。而抵抗力和恢复力相关性不显著意味着两者代表了稳定性相对独立的维度,高的抵抗力不代表着高的恢复力;因此两者在反映稳定性具有一定的代表性。
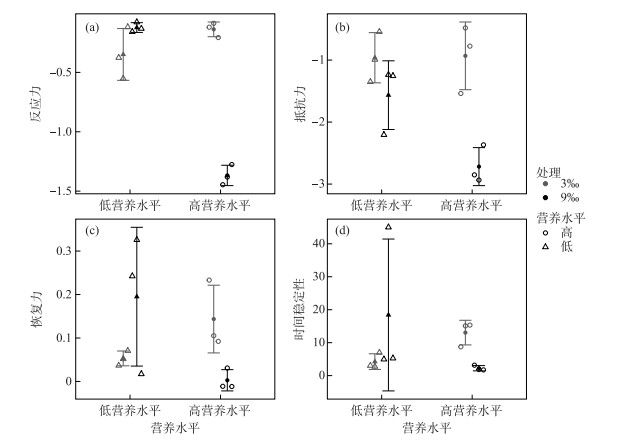
|
图 7 不同营养水平和干扰强度下浮游细菌群落基于细胞密度计算的反应力、抵抗力、恢复力和时间稳定性 (其中空心点表示实测值,实心点表示平均值。误差棒表示标准差) Fig.7 Reactivity, resistance, resilience and temporal stability based on cell density under different nutrient level and disturbance strength (The open circles represent measured value and the closed circles represent mean value; The error-bars represent the standard deviation) |
| 表 1 不同功能稳定性指标之间的斯皮尔曼相关性 Tab. 1 The spearman's correlation among different functional stability index |
使用Scheirer-Ray-Hare对稳定性进行双因素方差非参数检验,结果表明干扰强度显著改变了抵抗力(表 2),更高的干扰强度显著降低了抵抗力(图 7b)。干扰强度和营养水平对反应力、恢复力和时间稳定性存在显著的交互作用;在9‰盐度干扰下,高营养水平群落的反应力、恢复力和时间稳定性取到所有水平的最低值(图 7b~d)。
| 表 2 干扰强度和营养水平对不同稳定性指标的双因素方差分析的Scheirer-Ray-Hare检验 Tab. 2 The Scheirer-Ray-Hare test for two-way ANOVA of disturbance intensity and nutrient level for different stability metrics |
在所有干扰处理下,浮游细菌群落的动态变化存在一定的共性,这可能与群落的稳定性机制相关。具体来说,盐度干扰降低了群落细胞密度(图 4a),在群落结构方面也发现物种丰富度的损失(图 5a)。可以推测部分物种对盐度敏感,在干扰后死亡,进而导致整体细胞密度下降。而这些死亡的生物体裂解后将细胞内含物释放出来,可能为后续细胞生长提供底物[44]。这与我们观察到干扰组中溶解性有机碳相对于对照组快速增加一致(图 4b)。值得注意的是,在高营养水平组,溶解性有机质的可能来源除了死亡微生物释放的细胞内含物以外,还可能通过颗粒物分解产生。在盐度干扰后,微生物群落细胞密度的降低,可能导致对颗粒物分解产生的溶解性有机质的利用能力降低[17],进而使得溶解性有机质的积累。总体而言,干扰无论是导致敏感物种的死亡,还是抑制现有生物的活性,都增加了可利用的有机质,都为后续恢复过程中微生物的生长提供底物。
在群落结构方面,浮游细菌中拟杆菌门、浮霉菌门和疣微菌门等类群的相对丰度在不同处理下都相对于对照组降低,反映这些类群不适应盐度升高后的环境。这主要是因为盐度对物种有较强的筛选作用[17, 45-46]。仅有γ变形菌纲的相对丰度在不同营养水平下和干扰强度下均表现为增加。一些将盐度作为干扰的研究也报道,盐度干扰导致γ变形菌纲在干扰后增加[19-20, 22]。一般来说,相对于其他淡水优势细菌门类,γ变形菌纲能更加适应高盐度环境,甚至更加偏好高盐度环境[47];在高盐环境中,比如在盐湖和海洋中,γ变形菌纲比淡水湖泊中有着更高的相对丰度[47-48]。同时,γ变形菌纲也往往认为是富营养型菌,偏好富营养条件[47]。本研究也发现高营养水平群落比低营养水平群落有着更高比例的γ变形菌纲(图 6)。因此,推测γ变形菌纲的增加不仅与盐度的增加有关,而且可能与干扰后增加的溶解性有机碳密切相关。
总体来说,浮游细菌群落结构和功能的结果共同反映了盐度干扰后补偿过程:适应干扰后环境的盐度耐受物种,通过占据盐度敏感物种干扰腾出的生态位,甚至利用死亡微生物释放的有机物,从而促进群落生物量的恢复。而这一结果与前人的结果一致[49-50]。Flöder等发现,在改变盐度以后,生长快速的浮游植物种类占优势从而促进浮游植物群落恢复[49]。Steiner等发现,通过稀释降低水体多营养级群落生物量后,浮游植物的快速生长,促进群落水平上生物量的恢复[50]。
3.2 干扰强度和营养水平影响群落稳定性本研究发现,干扰强度越高,群落细胞密度的损失越大(图 4a)。这一结果与前人的研究结果相一致,例如Berga等研究盐度干扰强度和群落响应关系时,发现随着盐度干扰强度增加,浮游细菌群落的细胞密度、同化速率的损失也在增大[20, 35]。此外,本研究还发现干扰强度越高群落的抵抗力越低(图 7;表 2)。抵抗力越高(越接近于0),表示群落抵抗干扰并使自身在干扰下保持不变的能力越强[2];而抵抗力越低(越远离0),表示群落对干扰的敏感性越低。其中,高盐度干扰下低的抵抗力表示群落对强干扰更加敏感。
本研究发现,与抵抗力不同,反应力没有随着干扰强度的增加而降低,虽然两者都反映了群落对于干扰的敏感性[28]且表现出显著的正相关(表 1)。反应力表示群落放大干扰的能力,这与群落中物种适应环境变化的速度有关[30]。反应力越高(越接近于0),表示群落放大干扰的能力越弱,即群落越能够快速地适应干扰后的环境。在9‰盐度干扰下,低营养水平浮游细菌群落表现出了较高的反应力,这与其抵抗力的表现差异最大。这可能与部分微生物形成休眠体等手段避开干扰有关[51];在9‰盐度干扰下,低营养水平群落维持着相对较高比例的放线菌门(图 6),而放线菌可以形成孢子以避开不良环境[47]。这样一种干扰响应机制,可以延后群落的恢复过程,导致较低的反应力[30]。遗憾的是,以往的研究常常忽略了群落放大干扰的能力,比如Hillebrand等抵抗力定义为第一个时间点时处理组相对于对照组的变化[27],而与本研究的定义并不完全相同。在本研究中,干扰后最大变化往往不是干扰后的第一个时间点(干扰后6 h),一般在干扰后4~10 d才达到最大变化。反应力的研究可以为我们理解生物群落抵抗外界干扰提供新视角。
在3‰盐度干扰下,所有群落的细胞密度都在观测结束之前恢复到了对照组水平(落入到对照组置信区间内),并且在高营养水平群落表现出较高的恢复力。恢复力表示群落在干扰后恢复到原状的能力,在本研究主要指处理组恢复到对照组水平的快慢。恢复力越大,表示群落恢复到原先状态的能力越强,从而维持其正常的功能[32]。类似地,在植物群落的研究中也有发现营养水平的增加促进了群落生物量的恢复,并且往往通过快速生长的物种占优势实现的[52-53]。而高营养水平群落有着更高比例的富营养型菌,如本研究中的γ变形菌纲,可以促进群落的恢复[32]。相反,9‰盐度干扰下,部分低营养水平群落和所有高营养水平群落没有恢复到干扰前的水平。这可能是因为升高的盐度环境进一步增加细胞代谢成本,降低了生长效率[17]。而富营养型菌在干扰后占优势,一方面促进了细胞密度的恢复;另一方面,在资源有限的情况下也可能会限制群落的恢复,因为它们的生长往往是低效的,通过消耗大量底物用于自身生长[54]。比如,高营养水平群落在9‰盐度干扰后2~8天中观察到了细菌密度的快速但不完全的恢复(图 4a),这可能就是富营养型菌低效但快速增长的结果[55]。相对来说,低营养水平群落在9‰盐度干扰后,细胞快速增长的时间相对于高营养水平晚,对于溶解性有机质的消耗较少,最终部分低营养水平群落恢复到了干扰前的水平。总体来说,营养水平在3‰和9‰盐度干扰下对于恢复力表现出了不同的作用。
时间稳定性反应了群落在一定时间跨度上的变异程度[27]。时间稳定性越高,表示群落越能够缓冲环境的变化。在本研究中,时间稳定性与恢复力表现出显著正相关性,即更加快速的恢复越接近于线性的(残差较小)。而时间稳定性和恢复力,往往较少有显著相关的结果[27-28]。我们观察到显著的正相关可能是因为补偿效应造成了快速的恢复:完全且快速的恢复过程实现高恢复力的同时表现出较好的线性,即高的时间稳定性;而不完全但快速的恢复并不会展现出高的恢复力,并且时间稳定性较低。
值得注意的是,我们发现营养水平和干扰强度对反应力、恢复力和时间稳定性上存在显著的相互作用——高营养水平群落在9‰盐度干扰强度下表现出最低的稳定性。同时,在群落结构变化方面,营养水平和干扰强度也存在相互作用——仅高营养水平群落中,9‰盐度干扰下的群落结构变化高于3‰盐度干扰。我们推测,这可能是营养水平的增加重塑了浮游细菌群落结构,尤其是优势类群,导致群落对9‰盐度干扰呈较低的抵抗力[10, 52]。比如,高营养水平群落面对盐度干扰的α多样性损失要高于低营养水平(图 5a);低营养水平群落中β变形菌纲优势类群是Methyloversatilis属,而Methyloversatilis属在盐度干扰后增加(图 3b);而高营养水平群落中β变形菌优势类群是Pelomonas属,而Pelomonas属在盐度干扰后减少(图 3b)。此外,放线菌门可能通过形成孢子从而躲避剧烈的盐度干扰[51],以维持细胞密度的稳定,但是营养水平的增加导致了放线菌门相对丰度的减少(图 3a)。在9‰盐度干扰下高营养水平群落非常低的抵抗力(将近90% 的细胞死亡)和有限的营养来源最终限制浮游细菌群落恢复到干扰前的水平,进而表现为较低的恢复力和时间稳定性。
另一方面,盐度对于群落结构的影响明显小于营养水平(图 3a),这也是导致干扰强度对群落结构的影响并不显著。在生物地理格局上,盐度往往被认为是影响湖泊浮游细菌群落的最主要因素[8, 15-16],因此盐度干扰对于群落结构的作用应该强于营养水平。造成这一现象的原因有以下两种可能:(1)本研究所用的浮游细菌群落来源没有盐度干扰历史。有相似干扰历史的群落能够为新干扰提供更多潜在适应类群,促进群落结构对干扰的响应[2, 56]。(2)实验体系是一个封闭体系,与外界没有微生物的交流。从其他扩散源招募耐盐物种也可以帮助盐度改变的群落适应高盐环境,并且促进群落结构变化[22]。这两个原因均导致环境当中更加适应高盐环境的物种无法进入到体系当中,仅能从体系中已有物种中选择耐盐物种,大大减少了群落结构的变化,导致盐度干扰带来的变化小于营养水平。
在全球变化背景下,尤其是增温增强了水分蒸发和人类活动用水减少了淡水补给,内陆湖泊面临着盐度增加的风险[18]。野外调查的研究报道干旱/半干旱区域的内陆湖泊中高营养水平和高盐度水平往往同时发生,即高盐湖泊往往也有更高的营养水平[8, 57-59]。这可能是因为湖泊中水分蒸发的同时,未将水体中的营养元素带走,溶质浓度增加,导致盐度增加的同时也使得营养富集。本研究表明,高盐度和高营养水平下,浮游细菌群落结构变化最大,并且稳定性最低。类似地,Tang等也在野外调查中发现营养水平的增加进一步加剧了盐度对于浮游细菌群落多样性的作用[8]。总体来说,蒸发导致的盐度增加伴随着营养富集,会进一步加剧浮游细菌群落结构的变化和稳定性的降低,进而威胁浮游细菌群落执行正常功能;在此基础上,湖泊生态系统可能会变得更加脆弱。
4 结论1) 在细胞密度方面,不同处理下浮游细菌群落面对盐度干扰表现为敏感,其中部分群落在干扰后恢复到对照水平;在结构方面,盐度导致浮游细菌群落物种丰富度降低,部分敏感门类相对丰度降低,而部分适应干扰后环境的物种在干扰后占优势,导致群落结构发生显著变化。
2) 补偿效应在盐度干扰下是维持浮游细菌群落稳定的重要机制:盐度干扰降低了细胞密度,增加水体中可利用的有机物,促进了适应干扰后环境的物种生长,从而实现群落细胞密度上的恢复。
3) 更高的干扰强度显著降低群落细胞密度,降低抵抗力并且影响群落的恢复。而营养水平和干扰强度交互作用共同影响了群落结构的响应和稳定性,这主要是因素营养水平重塑了优势类群进而影响了群落对强盐度干扰的稳定性。我们观察到高营养水平和强盐度干扰下群落稳定性最低,这意味着内陆湖泊在盐度增加过程中伴随着的营养富集会进一步导致群落变得更加脆弱。
4) 多数稳定性指标之间表现出显著的正相关,可以协同指示浮游细菌群落的稳定性;抵抗力和恢复力之间并不显著相关,反映这两个指标可以从不同角度反映群落稳定性;多个稳定性指标的应用和高频次观测有助于了解浮游细菌群落对干扰的响应与稳定性机制。
5 附录附件1见电子版(DOI: 10.18307/2024.0234)。
附件1####R_code####
library(dplyr) #载入包
all_density < -read.csv('density_nutrient.csv') #读入文件,本文件包含treatment、time、nutrient、bottle和cells等五列指标;treatment包含3个处理,Control、Weak和Strong;time为干扰后时间;nutrient表示营养水平,HN和LN;bottle表示瓶子编号;cells表示测得的细胞密度。
####计算处理组每个实验单元相对于同一营养水平的对照组的LRR####
density_LRR <- all_density %>%
subset(treatment == 'Control') %>%
group_by(time, nutrient) %>%
summarise(mean = mean(cells), sd = sd(cells)) %>%
merge(all_density, by = c('time', 'nutrient')) %>%
#计算LRR,其中LRR为后续分析的主要指标,即处理组相对于对照组平均值的变化
mutate(LRR = log(cells / mean)) %>%
subset(treatment!= 'Control')
####计算处理组每个实验单元的抵抗力、抵抗时间和反应力####
resistance <-
density_LRR %>% group_by(bottle, treatment, nutrient) %>%
summarise(
#计算抵抗力resistance
resistance = min(LRR),
#计算抵抗时间resistance_time
resistance_time = time[(LRR = = min(LRR))],
#取抵抗时间以前的子集,以计算反应力Reactivity
reactivity = coef(summary(lm(
LRR ~ time2,
data = data.frame(LRR = LRR[time2 < = resistance_time],
time2 = time2[time2 < = resistance_time])
)))[2, 1]
)
density_after_resistance_time <- density_LRR %>%
merge(resistance, by = c('bottle', 'treatment', 'nutrient')) %>% #删除抵抗时间以前的数据,以免影响后续分析
subset(time > = resistance_time)
####计算处理组每个实验单元的恢复力与回复时间####
resilience_time <- density_after_resistance_time %>%
mutate(flag = ifelse(cells > (mean – sd * 2), 1, 0)) %>% #判断是否恢复到对照状态
group_by(bottle, treatment, nutrient) %>%
summarise(resilience_time = ifelse(sum(flag) = = 0, 16, min(time[flag = = 1]))) #计算恢复时间,如果群落未观察到恢复,则恢复时间记为最后一天
resilience <- density_after_resistance_time %>%
merge(resilience_time, by = c('bottle', 'treatment', 'nutrient')) %>%
subset(time < = resilience_time) %>%
group_by(bottle, treatment, nutrient) %>%
summarise(
#计算恢复力resilience
resilience = coef(summary(lm(LRR ~ time)))[2, 1],
#计算时间稳定性temporal_stability
temporal_stability = 1 / sd(residuals(lm(LRR ~ time)))
)
| [1] |
Falkowski PG, Fenchel T, Delong EF. The microbial engines that drive Earth's biogeochemical cycles. Science, 2008, 320(5879): 1034-1039. DOI:10.1126/science.1153213 |
| [2] |
Philippot L, Griffiths BS, Langenheder S. Microbial community resilience across ecosystems and multiple disturbances. Microbiology and Molecular Biology Reviews, 2021, 85(2). DOI:10.1128/mmbr.00026-20 |
| [3] |
Allison SD, Martiny JBH. Resistance, resilience, and redundancy in microbial communities. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 11512-11519. DOI:10.1073/pnas.0801925105 |
| [4] |
Cavicchioli R, Ripple WJ, Timmis KN et al. Scientists' warning to humanity: Microorganisms and climate change. Nature Reviews Microbiology, 2019, 17(9): 569-586. DOI:10.1038/s41579-019-0222-5 |
| [5] |
Hu JL, Amor DR, Barbier M et al. Emergent phases of ecological diversity and dynamics mapped in microcosms. Science, 2022, 378(6615): 85-89. DOI:10.1126/science.abm7841 |
| [6] |
Ratzke C, Barrere J, Gore J. Strength of species interactions determines biodiversity and stability in microbial communities. Nature Ecology & Evolution, 2020, 4(3): 376-383. DOI:10.1038/s41559-020-1099-4 |
| [7] |
Tobor-Kaplon MA, Bloem J, Romkens P et al. Functional stability of microbial communities in contaminated soils. Oikos, 2005, 111(1): 119-129. DOI:10.1111/j.0030-1299.2005.13512.x |
| [8] |
Tang XM, Xie GJ, Shao KQ et al. Aquatic bacterial diversity, community composition and assembly in the semi-arid inner Mongolia Plateau: Combined effects of salinity and nutrient levels. Microorganisms, 2021, 9(2): 208. DOI:10.3390/microorganisms9020208 |
| [9] |
Tobor-Kaplon MA, Bloem J, Romkens P et al. Functional stability of microbial communities in contaminated soils near a zinc smelter (Budel, The Netherlands). Ecotoxicology, 2006, 15(2): 187-197. DOI:10.1007/s10646-005-0050-4 |
| [10] |
Zhou LB, Wang SP. The bright side of ecological stressors. Trends in Ecology & Evolution, 2023, 38(6): 568-578. DOI:10.1016/j.tree.2023.01.010 |
| [11] |
Wu LW, Yang YF, Chen S et al. Microbial functional trait of rRNA operon copy numbers increases with organic levels in anaerobic digesters. The ISME Journal, 2017, 11(12): 2874-2878. DOI:10.1038/ismej.2017.135 |
| [12] |
Fierer N, Bradford MA, Jackson RB. Toward an ecological classification of soil bacteria. Ecology, 2007, 88(6): 1354-1364. DOI:10.1890/05-1839 |
| [13] |
de Vries FT, Shade A. Controls on soil microbial community stability under climate change. Frontiers in Microbiology, 2013, 4: 265. DOI:10.3389/fmicb.2013.00265 |
| [14] |
Lozupone CA, Knight R. Global patterns in bacterial diversity. Proceedings of the National Academy of Sciences, 2007, 104(27): 11436. DOI:10.1073/pnas.0611525104 |
| [15] |
Ji MK, Kong WD, Yue LY et al. Salinity reduces bacterial diversity, but increases network complexity in Tibetan Plateau Lakes. FEMS Microbiology Ecology, 2019, 95(12): fiz190. DOI:10.1093/femsec/fiz190 |
| [16] |
Wu QL, Zwart G, Schauer M et al. Bacterioplankton community composition along a salinity gradient of sixteen high-mountain lakes located on the Tibetan Plateau, China. Applied and Environmental Microbiology, 2006, 72(8): 5478-5485. DOI:10.1128/AEM.00767-06 |
| [17] |
Langenheder S, Kisand V, Wikner J et al. Salinity as a structuring factor for the composition and performance of bacterioplankton degrading riverine DOC. FEMS Microbiology Ecology, 2003, 45(2): 189-202. DOI:10.1016/S0168-6496(03)00149-1 |
| [18] |
Reid AJ, Carlson AK, Creed IF et al. Emerging threats and persistent conservation challenges for freshwater biodiversity. Biological Reviews, 2019, 94(3): 849-873. DOI:10.1111/brv.12480 |
| [19] |
Hu Y, Bai CR, Cai J et al. Low recovery of bacterial community after an extreme salinization-desalinization cycle. BMC Microbiology, 2018, 18(1): 1-12. DOI:10.1186/s12866-018-1333-2 |
| [20] |
Berga M, Zha YH, Székely AJ et al. Functional and compositional stability of bacterial metacommunities in response to salinity changes. Frontiers in Microbiology, 2017, 8: 948. DOI:10.3389/fmicb.2017.00948 |
| [21] |
Baho DL, Peter H, Tranvik LJ. Resistance and resilience of microbial communities-temporal and spatial insurance against perturbations. Environmental Microbiology, 2012, 14(9): 2283-2292. DOI:10.1111/j.1462-2920.2012.02754.x |
| [22] |
Comte J, Langenheder S, Berga M et al. Contribution of different dispersal sources to the metabolic response of lake bacterioplankton following a salinity change. Environmental Microbiology, 2017, 19(1): 251-260. DOI:10.1111/1462-2920.13593 |
| [23] |
Grimm V, Wissel C. Babel, or the ecological stability discussions: An inventory and analysis of terminology and a guide for avoiding confusion. Oecologia, 1997, 109(3): 323-334. DOI:10.1007/s004420050090 |
| [24] |
Kéfi S, Domínguez-García V, Donohue I et al. Advancing our understanding of ecological stability. Ecology Letters, 2019, 22(9): 1349-1356. DOI:10.1111/ele.13340 |
| [25] |
Donohue I, Hillebrand H, Montoya JM et al. Navigating the complexity of ecological stability. Ecology Letters, 2016, 19(9): 1172-1185. DOI:10.1111/ele.12648 |
| [26] |
Donohue I, Petchey OL, Montoya JM et al. On the dimensionality of ecological stability. Ecology Letters, 2013, 16(4): 421-429. DOI:10.1111/ele.12086 |
| [27] |
Hillebrand H, Langenheder S, Lebret K et al. Decomposing multiple dimensions of stability in global change experiments. Ecology Letters, 2018, 21(1): 21-30. DOI:10.1111/ele.12867 |
| [28] |
White L, O'Connor NE, Yang Q et al. Individual species provide multifaceted contributions to the stability of ecosystems. Nature Ecology & Evolution, 2020, 4(12): 1594-1601. DOI:10.1038/s41559-020-01315-w |
| [29] |
Chen JJ, Chi YG, Zhou W et al. Quantifying the dimensionalities and drivers of ecosystem stability at global scale. Journal of Geophysical Research: Biogeosciences, 2021, 126(4): e2020JG006041. DOI:10.1029/2020jg006041 |
| [30] |
Domínguez-García V, Dakos V, Kéfi S. Unveiling dimensions of stability in complex ecological networks. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(51): 25714-25720. DOI:10.1073/pnas.1904470116 |
| [31] |
Li ZY, Ye XZ, Wang SP. Ecosystem stability and its relationship with biodiversity. Chinese Journal of Plant Ecology, 2021, 45(10): 1127-1139. [李周园, 叶小洲, 王少鹏. 生态系统稳定性及其与生物多样性的关系. 植物生态学报, 2021, 45(10): 1127-1139.] |
| [32] |
Shade A, Peter H, Allison SD et al. Fundamentals of microbial community resistance and resilience. Frontiers in Microbiology, 2012, 3: 417. DOI:10.3389/fmicb.2012.00417 |
| [33] |
Orwin KH, Wardle DA. New indices for quantifying the resistance and resilience of soil biota to exogenous disturbances. Soil Biology and Biochemistry, 2004, 36(11): 1907-1912. DOI:10.1016/j.soilbio.2004.04.036 |
| [34] |
Roger F, Bertilsson S, Langenheder S et al. Effects of multiple dimensions of bacterial diversity on functioning, stability and multifunctionality. Ecology, 2016, 97(10): 2716-2728. DOI:10.1002/ecy.1518 |
| [35] |
Berga M, Székely AJ, Langenheder S. Effects of disturbance intensity and frequency on bacterial community composition and function. PLoS One, 2012, 7(5): e36959. DOI:10.1371/journal.pone.0036959 |
| [36] |
Vellend M. The theory of ecological communities. Princeton: Princeton University Press, 2016.
|
| [37] |
Gong Y, Tang XM, Shao KQ et al. Dynamics of bacterial abundance and the related environmental factors in large shallow eutrophic Lake Taihu. Journal of Freshwater Ecology, 2017, 32(1): 133-145. DOI:10.1080/02705060.2016.1248506 |
| [38] |
Caporaso JG, Lauber CL, Walters WA et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(supplement_1): 4516-4522. DOI:10.1073/pnas.1000080107 |
| [39] |
Callahan BJ, McMurdie PJ, Rosen MJ et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [40] |
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal, 2011, 17(1): 10. DOI:10.14806/ej.17.1.200 |
| [41] |
Wang Q, Garrity GM, Tiedje JM et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [42] |
Cole JR, Wang Q, Fish JA et al. Ribosomal Database Project: Data and tools for high throughput rRNA analysis. Nucleic Acids Research, 2014, 42(D1): 633-642. DOI:10.1093/nar/gkt1244 |
| [43] |
Sokal RR, Rohlf FJ. Biometry: The principles and practice of statistics in biological research. New York: Freeman, 1995.
|
| [44] |
Pherribo GJ, Taga ME. Bacteriophage-mediated lysis supports robust growth of amino acid auxotrophs. The ISME Journal, 2023, 17(10): 1785-1788. DOI:10.1038/s41396-023-01452-7 |
| [45] |
Li CC, Jin L, Zhang C et al. Destabilized microbial networks with distinct performances of abundant and rare biospheres in maintaining networks under increasing salinity stress. iMeta, 2023, 2(1): e79. DOI:10.1002/imt2.79 |
| [46] |
Mo YY, Peng F, Gao XF et al. Low shifts in salinity determined assembly processes and network stability of microeukaryotic plankton communities in a subtropical urban reservoir. Microbiome, 2021, 9(1): 128. DOI:10.1186/s40168-021-01079-w |
| [47] |
Newton RJ, Jones SE, Eiler A et al. A guide to the natural history of freshwater lake bacteria. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14-49. DOI:10.1128/MMBR.00028-10 |
| [48] |
Teittinen A, Virta L, Li MJ et al. Factors influencing the biodiversity of three microbial groups within and among islands of the Baltic Sea. FEMS Microbiology Ecology, 2021, 97(5): fiab049. DOI:10.1093/femsec/fiab049 |
| [49] |
Flöder S, Jaschinski S, Wells G et al. Dominance and compensatory growth in phytoplankton communities under salinity stress. Journal of Experimental Marine Biology and Ecology, 2010, 395(1/2): 223-231. DOI:10.1016/j.jembe.2010.09.006 |
| [50] |
Steiner CF, Long ZT, Krumins JA et al. Population and community resilience in multitrophic communities. Ecology, 2006, 87(4): 996-1007. DOI:10.1890/0012-9658(2006)87[996:pacrim]2.0.co;2 |
| [51] |
Dworkin J, Shah IM. Exit from dormancy in microbial organisms. Nature Reviews Microbiology, 2010, 8(12): 890-896. DOI:10.1038/nrmicro2453 |
| [52] |
Xu Q, Yang X, Song J et al. Nitrogen enrichment alters multiple dimensions of grassland functional stability via changing compositional stability. Ecology Letters, 2022, 25(12): 2713-2725. DOI:10.1111/ele.14119 |
| [53] |
Xu ZW, Liu HY, Meng YN et al. Nitrogen addition and mowing alter drought resistance and recovery of grassland communities. Science China Life Sciences, 2023, 66(7): 1682-1692. DOI:10.1007/s11427-022-2217-9 |
| [54] |
Roller BRK, Stoddard SF, Schmidt TM. Exploiting rRNA operon copy number to investigate bacterial reproductive strategies. Nature Microbiology, 2016, 1: 16160. DOI:10.1038/nmicrobiol.2016.160 |
| [55] |
Nemergut DR, Knelman JE, Ferrenberg S et al. Decreases in average bacterial community rRNA operon copy number during succession. The ISME Journal, 2016, 10(5): 1147-1156. DOI:10.1038/ismej.2015.191 |
| [56] |
Santillan E, Seshan HR, Constancias F et al. Trait-based life-history strategies explain succession scenario for complex bacterial communities under varying disturbance. Environmental Microbiology, 2019, 21(10): 3751-3764. DOI:10.1111/1462-2920.14725 |
| [57] |
Yue LY, Kong WD, Ji MK et al. Community response of microbial primary producers to salinity is primarily driven by nutrients in lakes. Science of the Total Environment, 2019, 696: 134001. DOI:10.1016/j.scitotenv.2019.134001 |
| [58] |
Hu Y, Jiang XY, Shao KQ et al. Convergency and stability responses of bacterial communities to salinization in arid and semiarid areas: Implications for global climate change in lake ecosystems. Frontiers in Microbiology, 2022, 12: 741645. DOI:10.3389/fmicb.2021.741645 |
| [59] |
Li ZX, Gao Y, Wang SY et al. Phytoplankton community response to nutrients along lake salinity and altitude gradients on the Qinghai-Tibet Plateau. Ecological Indicators, 2021, 128: 107848. DOI:10.1016/j.ecolind.2021.107848 |
 2024, Vol. 36
2024, Vol. 36 


