(2: 河海大学, 水灾害防御全国重点实验室, 南京 210098)
(3: 中国科学院大学中丹学院, 北京 100049)
(4: 中国科学院南京地理与湖泊研究所, 鄱阳湖湖泊湿地综合研究站, 九江 332899)
(2: The National Key Laboratory of Water Disaster Prevention, Hohai University, Nanjing 210098, P. R. China)
(3: Sino-Danish College, University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
(4: Poyang Lake Wetland Research Station, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Jiujiang 332899, P. R. China)
植物附着细菌(epiphytic bacteria)是指生长在植物表面、依靠或者不依靠植物的代谢产物作为营养、和植物形成紧密或者松散的共生关系的一类细菌[1]。关于植物附着细菌的研究可追溯至1946年,James等对小麦幼苗的可培养附着细菌进行显微镜观察和计数[2]。而水生植物附着细菌的研究远远滞后于陆生植物,直到1971年Allen等首次描述了湖滨带几种大型水生植物的附着细菌,并探究了其与藻类生产力、物质代谢能力以及植物间的相互作用[3]。而后至今,大量研究表明水生植物及其附着细菌会相互作用并作为一个整体共同行使着包括水质净化和污染物去除等重要生态功能,对于维持水生生态系统健康具有重要意义[4-6]。一方面,水生植物为细菌定殖提供微生态位,并通过分泌氧气和低分子有机碳为细菌的生长提供相应支持[5]。另一方面,由于环境中的大部分营养物质通常不能被植物直接吸收利用,附着细菌可以将这些物质转化为更易被利用的小分子形态供植物吸收,同时转化过程中产生的次生代谢产物可以用来抵御无脊椎动物及有害微生物对植物的不利影响[1, 7]。此外,在水生植物衰亡阶段,附着细菌连同其他分解者共同参与植物残体的分解矿化过程,在水生生态系统的养分循环和能量流动过程中发挥重要作用[8-9]。
尽管水生植物附着细菌在水生生态系统中占据重要生态角色和地位,目前对其群落结构、对环境变化的响应以及潜在的生态学构建机制还缺乏系统认知。同时,水生植物的种类和组织结构复杂多样,关于不同种类水生植物组织结构上附着细菌群落的研究尚处于探索阶段。叶片和根系作为水生植物的重要组织,在净化水质和降解污染物等过程中扮演着重要角色,也是目前被研究最多的植物生态位[4-5]。因此,探究水生植物叶片和根系附着细菌群落多样性和组成特征、构建机制及其在水生生态系统中的生态功能,可以丰富对这些特殊界面生境中细菌群落的认知,加深对水生生态系统中细菌群落结构与功能形成机制、多样性保护、生物资源挖掘及利用等方面的理解。
使用Web of Science核心合集数据库,以主题词:((“aquatic plants” or “submerged plants” or “emerged plants” or “floating plants” or “floating-leaved plants” or “macrophytes” or “emerged macrophytes” or “submerged macrophytes” or “floating macrophytes” or “floating-leaved macrophytes”) and (“rhizosphere” or “phyllosphere” or “root” or “leaf”) and (“bacteria”)) 对数据库中1900—2023年的文献进行检索,文献类型界定为“Article(论文)”和“Review(综述)”。结果表明,全球水生植物叶片和根系附着细菌群落相关研究发文量总计1097篇,其中近10年发表文章741篇,我国相关主题论文数量为307篇,接近全球发文总量的1/3。由此可见,水生植物附着细菌群落研究在国内外处于广受关注且快速发展阶段。此外,根据检索文献的关键词,水生植物附着细菌群落的研究热点也逐渐由多样性和组成向构建机制和功能特征转变。因此,本文在当前已发表文献的基础上,对水生植物附着细菌群落(主要针对叶片和根系)的组成和多样性、构建机制及其在湖泊碳氮循环过程中的作用进行综述,并对未来水生植物附着细菌群落的研究方向进行了展望。
2 水生植物附着细菌群落的多样性及组成 2.1 水生植物附着细菌群落多样性特征在过去几十年间,分子生物学以及生物信息学等研究技术在环境微生物学领域快速发展,为深入探究水生植物附着细菌奠定了研究基础。上世纪末和本世纪初,部分学者基于细胞染色和显微镜观测技术对水生植物叶片及根系附着细菌群落进行了量化研究,结果表明:水生植物叶片附着细菌数量通常为105~107个/cm2,其中活性菌占比为2.2%~42.9% [10-11]。基于分离培养技术,可通过观察到的菌落单元(colony-forming unit, CFU)数目来反映可培养细菌丰度。有研究表明,沉水植物穗状狐尾藻(Myriophyllum spicatum)叶片附着细菌可培养数目为1.08×106~7.82×106 CFU/cm2 [12],而挺水植物芦苇(Phragmites australis)和光千屈菜(Lythrum anceps)根系附着细菌可培养数目最高可分别达4.0×108和1.1×108 CFU/g[13]。值得注意的是,分离培养只适用于具有活性的可培养细菌类群,对于休眠细菌以及普遍存在的不可培养细菌检测能力有限。
近年来,针对细菌16S核糖体RNA(16S rRNA)的高通量测序技术为研究水生植物附着细菌群落的多样性和组成提供了基于DNA水平的视野。一些研究通过评估细菌序列的可操作分类单元(operational taxonomic unit,OTU)或扩增子序列变体(amplicon sequence variant,ASV)的不同指数(包括Shannon指数和Simpson指数等)探究了不同水生植物附着细菌群落的多样性。例如:He等通过高通量测序确定了苦草(Vallisneria natans)和黑藻(Hydrilla verticillata)叶片附着细菌群落的OTU数目为500~2000个[14];而Huang等发现芦苇和菰(Zizania latifolia)的根际附着细菌的OTU数目为7000~8000个[15]。然而,这些结果对于测序深度具有较高的依赖性,并且不能代表相应生境细菌群落的绝对丰度,未来随着技术手段的升级,可能提供更全面的水生植物附着细菌多样性信息。
除了描述水生植物附着细菌在不同种类或不同组织结构间的多样性差异,一些研究也试图通过将其与周围环境如水体和沉积物中的细菌群落多样性进行比较,以期进一步探究水生植物附着细菌的形成机制及多样性分布规律。许多研究在陆生植物中普遍观察到从土壤到植物根际,细菌群落多样性逐渐下降的趋势,并将其归因于植物根系施加的环境梯度(例如:碳源、pH值或养分等)对细菌群落的选择作用[16-17]。然而,不同于陆生植物,一些学者发现芦苇[18-19]、菖蒲(Acorus calamus)[20]、互花米草(Spartina alterniflora)[21]、菰[15]、苦草[22]等多种水生植物根际细菌多样性显著高于非根际。这一方面可能是由于水生植物根系泌氧改变了根际沉积物的绝对厌氧条件,从而增加了好氧细菌类群的生长机会;另一方面,根系释放的分泌物减少了细菌成员间对养分资源的竞争并且促进了一些稀有类群的发育,从而提高了细菌群落多样性[19]。Wang等通过比较陆生植物蒺藜苜蓿(Medicago truncatula)和淹水植物水稻(Oryza sativa)根际细菌群落发现,淹水植物根际细菌群落比陆生植物更加丰富多样,并且通过比较根际细菌的相对丰度和绝对丰度,间接证明了相比于定量方法,16S扩增子测序可能无法完全捕捉到根际细菌多样性的增加[23]。
相比之下,针对水生植物叶际附着细菌多样性的研究较少。He等比较了中国12个淡水湖泊中菹草(Potamogeton crispus)叶片附着细菌和水体浮游细菌的多样性差异,发现叶片附着细菌的物种丰富度和系统发育多样性均显著高于水体浮游细菌[24]。此外,研究表明,水生植物的种类及叶片结构对其附着细菌的多样性也具有显著影响[25]。例如,Levi等发现,与叶片形态较为复杂的线叶水马齿(Callitriche hermaphroditica)相比,叶片形态相对简单的小黑三棱(Sparganium emersum)叶片附着细菌的丰富度和均匀度较低[26]。近期,He等通过室内微宇宙实验提出了苦草叶片和根系附着细菌的多样性分布模型,认为无论在叶片还是根系,相比于对应的植物内生组织或者外界环境,附着细菌群落具有最高的多样性[27]。
2.2 水生植物附着细菌群落的组成及潜在功能研究表明,水生植物附着细菌大都隶属于变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、酸杆菌门(Acidobacteria)和放线菌门(Actinobacteria)等(表 1、表 2)。此外,沉水植物叶片附着细菌还含有较高丰度的蓝细菌门(Cyanobacteria),而部分挺水植物根系附着大量绿弯菌门(Chloroflexi)(表 1、表 2)。
| 表 1 水生植物叶际细菌群落分类学组成 Tab. 1 Taxonomic composition of bacterial communities in the phyllosphere of aquatic macrophytes |
| 表 2 水生植物根际细菌群落分类学组成 Tab. 2 Taxonomic composition of bacterial communities in the rhizosphere of aquatic macrophytes |
变形菌门作为在多数自然生境中占绝对优势的细菌类群,在水生植物根叶表面具有相对于周围环境更高的相对丰度,这可能与其是r-策略细菌密切相关,因为它们可以迅速利用水生植物表面分泌的低分子有机物并且迅速繁殖[15, 19]。其中,β-变形菌门(Betaproteobacteria)经常在溪流、河流和湖泊中不同介质表面的生物膜细菌群落中占优[28-29]。而α-变形菌门(Alphaproteobacteria)的成员大多具有降解腐殖质和抗牧食能力[30]。在水生植物附着细菌类群中丰度较高的拟杆菌门具有利用复杂大分子的能力[31-32],在降解悬浮颗粒中发挥重要作用[30]。此外,许多沉水植物叶片特异性富集大量的蓝细菌门,这可能与其光合能力有关,但是过量的蓝细菌可能与植物竞争光照资源,导致水生植物的衰亡[33-34]。表 1和表 2总结了不同类型的常见水生植物叶片和根系附着细菌的组成,其中关于叶片附着细菌的研究多集中于沉水植物,而与根系附着细菌相关的研究多集中于挺水植物。
在更低的分类学水平,一些研究识别了不同水生植物附着细菌中的优势类群。例如,沉水植物轮叶狐尾藻(Myriophyllum verticillatum)叶片附着细菌中的优势物种为好氧或兼性厌氧的Comamonadaceae(β-变形菌门)、Methylophilus(β-变形菌门)、Pirellula(浮霉菌门)和Methylophilaceae(β-变形菌门)[35]。而同属沉水植物的菹草叶片表面附着大量的Rhodobacteraceae(α-变形菌门)、Porphyrobacter(α-变形菌门)和Duganella(β-变形菌门),它们可以形成初始生物膜并且具有降解多环芳烃的能力[24]。与氮、磷转化密切相关的几个隶属于蓝细菌门的属,包括Nostoc_PCC_8976、Calothrix_KVSF5和Leptolyngbya_PCC_6406以及Acidibacter(γ-变形菌门)等类群在微齿眼子菜(Potamogeton maackianus)叶片附着细菌中占绝对优势[36]。相比之下,水生植物根系表面大多聚集具有促进植物生长、降解有机物和抑制有害藻类生长等功能以及参与硝化、反硝化过程的细菌类群,并且在不同水生植物物种间具有宿主特异性。例如,芦苇根系附着细菌以Geobacter(δ-变形菌门)、Acinetobacter(γ-变形菌门)、Pseudomonas(γ-变形菌门)、Candidatus_Koribacter(酸杆菌门)、Candidatus_Solibacter(酸杆菌门)、Dactylosporangium(放线菌门)和Bacillus(厚壁菌门)为优势类群,而南荻(Triarrhena lutarioriparia)根系附着大量Rhodoplanes(α-变形菌门)和Bacillus(厚壁菌门),这些类群均具有促进植物生长的作用,并且部分类群也参与硝化和反硝化过程[37]。此外,由于水生植物根系泌氧造成根际微域氧化还原电位的强烈变化,水生植物根际富集了一系列参与不同元素氧化还原过程的细菌类群[38]。例如,杉叶蕨藻(Caulerpa taxifolia)根系大量富集了具有包括氮、硫和铁在内的好氧和厌氧营养循环功能的Desulfobulbaceae和Desulfococcus(均隶属于δ-变形菌门)[39]。摩羯大叶藻(Zostera capricorni)根际显著富集的Sulfurimonas(ε-变形菌门)可以还原硫化合物(如H2S)作为电子供体,并且利用硝酸盐、亚硝酸盐或氧气作为电子受体来产生硫酸盐[39]。
3 水生植物附着细菌群落形成的主要驱动因素水生植物附着细菌群落具有动态变化特征,其多样性和组成与宿主和环境因素密切相关,同时也受到细菌种间相互作用的影响(图 1)。
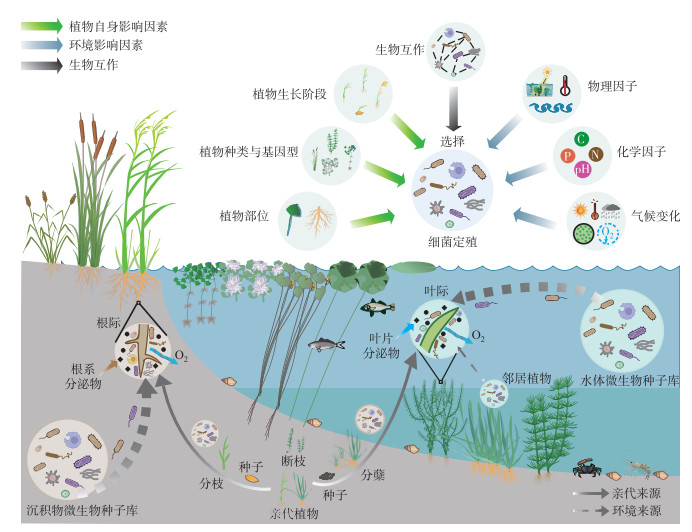
|
图 1 水生植物附着细菌群落的来源及影响因素 Fig.1 The sources and influencing factors of aquatic macrophytes epiphytic bacterial community |
来自水生植物不同生态位、种类或基因型以及生长阶段的宿主选择效应在其附着细菌群落的塑造过程中起着决定性作用。
3.1.1 植物附着生态位水生植物叶和根生长的环境背景差异造成叶际和根际附着细菌的来源及迁移途径不同[63]。水生植物的根大多深植于沉积物中,后者为根际提供了丰富的微生物资源库。沉积物中的细菌类群首先通过被动扩散到达植物根际,进一步受“根际效应”影响,经宿主选择定殖于植物根际。相比之下,水生植物叶际细菌群落的主要来源包括水体(沉水植物的叶片浸没于水体中)和空气(挺水植物、浮叶植物和漂浮植物的部分叶片暴露于空气中)。Lu等通过溯源分析发现沉水植物水盾草叶际细菌主要来源于水体浮游细菌(约65.79%)[57]。此外,考虑到浮游细菌受到水体流动性的影响,具有较高的群落周转率[64],并且对水体环境变化尤为敏感,因此水生植物叶际细菌群落可能具有较高的时空变异性。
水生植物附着细菌除了来源于周围环境,叶片和根系内生细菌也会向外释放。He等对苦草全生态位细菌群落进行了溯源分析,发现苦草根内生细菌贡献了根际细菌来源的25.4%,而叶内生细菌贡献了叶际细菌来源的51.4%,表明内生细菌可以向附着生态位贡献相对比例的细菌群落,并且叶片具有相比于根系更高的细菌水平传播能力,这可能是由于沉水植物叶片比根系具有更高的再生和代谢能力[27]。此外,水生植物不同组织部位具有功能性状差异,导致叶片和根系提供的微环境显著不同[65]。显微分析发现,水生植物叶片表皮的蜡质层相对陆生植物较薄,在促进养分吸收的同时也导致叶片表面的营养相对贫瘠,使得细菌的分布并不均匀[66]。而挺水植物、漂浮植物和浮叶植物的叶片由于长期暴露在空气中,面临着包括温/湿度波动大、紫外线辐射强等诸多不利条件。由于叶片营养资源有限,在面对非生物胁迫时,水生植物叶际细菌群落也会呈现出与根际细菌群落截然不同的重构策略[67]。相比之下,水生植物的根际完全浸没于沉积物中,通过释放大量根系分泌物为细菌提供了营养丰富的活动热区。因此,较多的研究结果表明水生植物根际细菌群落多样性显著高于叶际细菌群落[40, 67]。
3.1.2 植物种类与基因型不同种类的水生植物在功能性状、免疫系统和营养元素调节机制等方面存在遗传差异,这些均会导致其附着细菌群落具有宿主特异性[68]。此外,水生植物在不同基因型间也会对附着细菌表现出强烈的宿主选择效应,如:大陆尺度的野外调查研究发现芦苇不同谱系对其根际细菌群落结构的影响要强于环境因素,揭示了芦苇基因型在空间尺度上塑造根际细菌群落的关键作用[69]。
水生植物种类或基因型主要通过调控植物代谢产物的组成、叶片和根系的物理结构特征等影响其附着细菌群落结构。如:陆松柳等利用水培法收集了3种挺水植物根系分泌物,发现与菱白(Zizania caduciflora)和美人蕉(Canna indica)相比,千屈菜(Lythrum salicaria)根系分泌了更多的小分子有机酸、酚类和芳香族蛋白质,从而导致微生物可利用碳源增多以及根际细菌群落多样性增加[70]。而同属沉水植物的黑藻和微齿眼子菜由于根系分泌的有机酸组成不同,根际微环境中无机氮形态和浓度存在显著差异,使得二者根际分别倾向于富集更多的硝化菌和厌氧氨氧化菌[22]。水生植物根叶结构(如大小和形状等)也是影响细菌定殖的重要因素。与其他类型的水生植物相比,挺水植物根系较为发达,可为细菌提供更广泛的附着界面,从而具有相对较高的根际细菌群落多样性[71]。此外,不同种类的沉水植物叶片形态各异,例如,金鱼藻的叶片呈针状,苦草叶片为线型,而微齿眼子菜、黑藻和穗状狐尾藻等叶片为披针形,不同的叶形可为细菌定殖提供不同的物理生境和附着面积。研究发现,由于穗状狐尾藻的叶片表面体积比高于穿叶眼子菜(Potamogeton perfoliatus),因此具有更高的叶际细菌细胞数量[72]。
3.1.3 植物的生长阶段水生植物根叶的生理状态及其代谢产物会随着植物的生长发育发生变化。因此,水生植物不同生长阶段叶片和根系的物理结构、化学组成及代谢产物的变化是影响附着细菌群落多样性及组成变化的主要因素。
由于水生植物广泛采用无性繁殖体进行种群扩散[73],植物可能通过断枝、分蘖、分枝等引起叶际和根际细菌群落的传播扩散。此外,也有研究表明,当水生植物利用种子繁殖时,垂直传播可能在植物生长初期起到决定先锋物种的关键作用,随着种子发芽,植物开始广泛富集环境微生物[74]。当植物生长发育趋于成熟时,宿主选择作用增强,附着细菌群落逐渐形成稳定的动态变化结构[75]。例如,黑藻、苦草和篦齿眼子菜叶际细菌群落的物种丰富度从幼苗期到成熟期均持续增加,在进入成熟期后趋于稳定且同种植物样本间细菌群落差异变小[14, 76]。范婤调查了太湖典型沉水植物竹叶眼子菜(Potamogeton malaianus)、金鱼藻和菹草叶际功能微生物在植物生长阶段的动态变化,发现随着叶片年龄的增加,沉水植物叶际富集了更多氮循环相关的功能细菌类群[77]。基于代谢组学的研究发现,水芹(Oenanthe javanica)在繁殖期、幼苗期和成熟期根系活性代谢物,包括甜菜碱、硬脂酸和芥酸酰胺等参与植物化感作用和反硝化细菌富集过程的重要代谢产物存在显著差异[78]。以上研究表明:水生植物可根据其生长发育需求选择性塑造附着细菌群落。
此外,水生植物从幼苗到成熟植株的发育过程中,植物根系不断变化的形态学特征也是影响附着细菌定殖的重要因素。由于植物根的类型(初生、次生)和区域(根尖、根管、侧生)不同,细菌群落在植物根际中的分布并不均匀,并且在植物生长过程中动态运动,从而在根际微环境中表现出高度的时空动态变化[79]。水生植物生长发育过程中也伴随着季节更替,因此水生植物生长阶段对附着细菌的选择效应除了与植物代谢和形态结构等因素密切相关,同时还受季节性复杂环境因素的影响。
3.2 环境因素对水生植物附着细菌群落的调控作用水生植物附着细菌群落与植物宿主及其周围环境相互依存,除宿主自身选择效应外,外界环境因素包括区域尺度的气候和海拔变化、湖泊的理化特征以及植物叶/根微界面的理化参数也是影响水生植物附着细菌群落的重要因素。此外,作为水生植物附着细菌的重要种子库,生活在水域中的水体浮游细菌和定殖于沉积物中的底栖细菌群落均对环境变化极为敏感,因此,环境因素也可能通过影响浮游细菌和底栖细菌群落进而改变水生植物附着细菌群落的多样性与组成[57]。
3.2.1 区域尺度上的环境因子在较大的空间尺度上,环境因素对水生植物附着细菌群落的影响主要来源于不同地理位置或采样点的气候、海拔、纬度以及环境理化因子的差异。如:He等调查了长江中下游14个湖滨带芦苇根际和非根际沉积物中的细菌群落,发现空间距离和沉积物的pH、总氮、总磷以及有机质含量等驱动了芦苇根际细菌群落的多样性和组成变化[19]。另一项关于长江中下游12个湖泊沉水植物菹草叶际和水体细菌群落的对比研究发现,环境和空间因子对菹草叶际细菌群落多样性的影响要高于浮游细菌群落,其中水体总氮和氨氮是叶际细菌群落多样性变化最主要的解释因子[24]。
3.2.2 湖泊的理化特征在湖泊内部,水深、流速、光照、温度等物理条件在影响水生植物附着细菌群落组成上发挥着重要作用。已有研究发现水体流速通过促进叶际溶解氧扩散以及引起沉积物再悬浮进一步影响苦草和黑藻叶际细菌群落多样性[80]。水深和光照直接或间接影响水生植物的光合作用及生长发育,进而影响其附着细菌群落组成。尤其是生活在低氧和弱光环境中的沉水植物,对水深和光照的变化尤为敏感。Li等发现苦草叶片附着细菌群落的多样性随水深(0.3、0.6、1.2和1.5 m)呈现先增加后降低的单峰模式,表明水深过浅(< 0.6 m)或过深(>1.2 m)都会抑制苦草的光合作用,对植物造成氧化损伤,影响其叶片代谢,进而改变叶际细菌群落多样性[81]。自然条件下的季节变换和昼夜交替引起的温度差异可通过影响水生植物的生长繁殖和代谢速率进而引起其附着细菌群落的动态变化[18, 82]。
此外,水生植物附着细菌群落对氮、磷等营养盐浓度和有机物浓度的变化也会产生明显响应。一方面,水生生态系统中的碳、氮、磷等生源要素可作为细菌生长的营养物质对水生植物附着细菌群落的多样性和组成产生直接影响,且这一影响受到水体和沉积物营养水平的本底值[83]、营养盐输入浓度和频率[84]以及植物物种[85]等多种因素的调控;另一方面高浓度营养盐输入也会对水生植物造成氧化压力和营养胁迫,宿主植物可通过选择性富集功能细菌类群来缓解非生物胁迫。例如:在高浓度的氮素输入下,沉水植物苦草叶际会选择性富集氮循环相关的功能细菌类群来促进氮素转化[67, 86]。最近的研究也越来越关注全球变化背景下水生植物附着细菌群落与植物生长和健康的关系,探讨多个环境因子对水生植物附着细菌群落的复合影响,包括流速和温度[87]、流速和水深[88]、营养盐和升温[89]等。
3.2.3 水生植物叶/根微界面的环境因子受植物代谢和附着细菌的生命活动影响,水生植物叶/根微界面的环境因子(主要包括溶解氧(DO)、pH和氧化还原电位(ORP)等)及溶解性物质含量显著不同于周围水体和沉积物[90],这些微环境中理化因子的时空变化也会影响水生植物附着细菌群落组成[91]。例如,沉水植物叶际的氧浓度和pH均显著高于周围水体,且具有明显的空间差异,由此在叶际形成富氧—厌氧交替出现的氧化—还原微环境,促进参与氮素循环的细菌类群在叶际富集[66]。此外,Han等利用平面光电极技术发现苦草根际微域中存在好氧层,并可显著促进活性磷向非活性磷的转化,这可能与根际泌氧促进特定细菌类群的富集有关[92]。
3.3 种间相互作用对水生植物附着细菌群落的影响除宿主和环境因素外, 细菌类群之间的相互作用对于水生植物附着细菌群落稳定性和植物健康也起到重要的调节作用。对于定殖在水生植物叶际和根际的细菌群落而言,栖息在宿主同一生态位上的不同细菌类群既可以通过营养依赖、群体感应等机制相互合作,也会通过资源抢夺、接触杀伤、释放挥发性有机化合物等竞争机制相互拮抗,从而形成稳定的共存群落[93]。例如:许多植物附着细菌的基因组可编码种间和种内杀菌机制,从而调节植物宿主内其他细菌类群的分布、丰度和多样性[94]。此外,不同细菌类群间代谢产物的相互交换也有利于细菌生长[95]。如:从芦苇和香蒲(Typha orientalis)等水生植物根际分离得到的枯草芽孢杆菌(Bacillus subtilis)不仅具有水质净化功能,对病原菌也有明显的抑制作用[96]。Ishizawa等构建了由6种细菌组成的人工合成菌群,并将其接种到无菌浮萍(Lemna minor)的叶片表面,发现6个细菌菌株之间存在强烈的种间相互作用,单个菌株的移除均会影响整个细菌群落结构[97]。因此,细菌类群间的协作与竞争关系不仅能够影响细菌群落自身的组成和稳定性,而且可能进一步削弱或促进宿主植物抗逆性。
由于受微生物可培养性的限制,目前开发出的共现网络分析方法能够基于高通量测序数据推断微生物群落内物种的共现模式,识别微生物群落中的关键物种[98-99]。多项研究发现与水体和沉积物细菌群落相比,水生植物叶际和根际细菌群落共现网络的复杂度显著增加[15, 37, 57]。尽管这些分子生态网络研究中观察到的物种间的相关性不一定能够完全准确地预测水生植物附着细菌类群间的互作关系,但网络中的关键节点可以为筛选附着细菌群落内的核心物种提供借鉴。
4 影响水生植物附着细菌群落构建的生态学过程尽管已知宿主选择效应和外部环境因子在水生植物附着细菌群落的构建中发挥重要作用,明确不同生态学过程如何塑造水生植物附着细菌群落对理解物种多样性的形成和维持机制至关重要。生态位理论和中性理论共同解释了植物微生物组多样性的维持机制,但是其相对重要性受到多种因素影响[69, 100]。生态位理论强调物种在所有环境中均存在,但是不同的环境选择了不同的物种,因此其代表的选择过程属于确定性过程;而中性理论则认为物种在不同环境中的差异来源于扩散或者生态漂变;此外,成种/多样化/突变也对微生物的群落构建起到至关重要的作用[101-102]。前人归纳了以这4种过程为代表的确定性和随机性过程对于植物微生物群落构建的影响,但主要聚焦于陆生植物[100],水生植物附着细菌群落构建的相关研究尚处于起步阶段。本文尝试更全面地阐述不同生态学过程在水生植物叶片和根系附着细菌群落构建中的贡献。
4.1 选择过程不同的生物及非生物因素驱动了水生植物附着细菌群落的组成及动态变化。正如3.1节所述,不同的水生植物种类、生态位以及生长阶段通常富集特定细菌类群,这是由于这些生物因素导致水生植物产生了物理、化学以及生物特性方面的差异,这些差异一方面来自水生植物结构上的异同,另一方面来源于其暴露环境的差异。例如,叶片和根系生理结构不同,且分别暴露在水体/空气和沉积物中,因此对其附着细菌类群施加了不同的选择强度和方向[40, 63]。除了水生植物本身,由非生物因素导致的环境变化也可对水生植物附着细菌群落产生较大的直接或间接影响。环境变化,包括水深、流速、光照、温度、营养条件,可以直接影响或者通过影响植物生长状态间接塑造水生植物附着细菌类群[80-82, 103]。例如,随着流速或者温度的增加,穗状狐尾藻叶片附着细菌的群落构建过程逐渐由随机性过程主导转变为确定性过程主导[87]。有研究表明,气候和地理因素在一定程度上驱动了芦苇根际细菌群落的生物地理学差异,并且随着尺度的增加,以选择或环境过滤作用为代表的确定性过程对于附着细菌群落构建的影响逐渐增强,表明水生植物根系附着细菌的群落构建过程具有显著的尺度依赖效应[19]。最后,细菌类群间的相互作用也会对水生植物附着细菌群落产生较大影响,其中特定的细菌组合甚至单一类群的存在都可通过拮抗或有益的相互作用来影响水生植物附着细菌群落[83, 104]。
4.2 扩散过程扩散是指生物个体在空间的迁移[102],包括扩散限制和均质扩散。扩散限制通常会导致群落相异性升高, 而均质扩散则造成群落相异性降低[105]。水生植物附着细菌有多种来源途径,包括沉积物、水体、空气、水生动物以及邻近植物等[22, 100],使得扩散过程对附着细菌群落的构建产生重要影响。一些研究强调了扩散限制在塑造不同水生植物叶际细菌群落中的主导作用。例如,Li等发现自然湿地中金鱼藻、眼子菜(Potamogetonaceae)、路南海菜花和粉绿狐尾藻等不同水生植物的叶片附着生物膜反硝化细菌群落主要受到包括扩散限制(64.52%)和其他不确定过程(23.20%)等随机过程的驱动,说明在无序水动力交换的水体环境中细菌群落多样性模式更容易受扩散影响[106]。而在区域尺度,扩散限制对水生植物的根际附着细菌群落也具有显著的塑造作用,例如:关于长江中下游14个湖滨带芦苇根际细菌群落多样性模式的研究发现,扩散限制在根际细菌群落构建中的重要性随着尺度的增加线性增强[19]。相比于扩散限制,均质扩散对水生植物附着细菌群落的影响还缺乏研究[107]。此外,扩散也可以通过引入先锋效应,即凭借细菌类群的到达顺序来促进或者抑制未来定殖者的成功定殖,从而塑造水生植物附着细菌群落[100]。
4.3 生态漂变过程生态漂变是指物种的随机出生和死亡,在某些情况下是群落组成变化的有力驱动因素,特别在群落物种少、总体丰度低、环境选择压力弱的条件下,群落构建过程受随机性影响增强[100]。研究发现:相比于寒冷的冬季,在温暖的夏季,芦苇根际细菌群落更多受随机性过程驱动,从而呈现出较高的群落相异性,这一方面可能是由于环境温度胁迫的解除,另一方面夏季植物生长旺盛可能促进了根系分泌活动,从而削弱了根际微域的营养限制,这些均可能导致根际细菌群落生态漂变的增强[18]。而另一项针对苦草和黑藻叶际附着细菌群落的研究表明:除了扩散限制(46.43%),生态漂变(32.14%)对于细菌群落的驱动效应大于异质性选择(21.43%)[108],这一方面可能是由于相对均匀的水体环境削弱了环境选择或者过滤的发生;另一方面叶际细菌具有较低的细菌群落丰度和多样性,可能更容易受到群落中物种随机出生和死亡事件的影响[108-109]。值得注意的是,目前大多数研究并没有直接对生态漂变进行定量分析的准确方法,而是将除了选择和扩散之外的不确定过程等同于生态漂变。
4.4 成种/多样化/突变过程成种/多样化/突变是产生新基因变异的生物进化过程, 也是物种形成和灭绝之间的平衡状态,尽管它们是影响群落构建的重要生态过程, 但却非常难以研究, 因为其通常涉及到长期的进化过程[101-102]。在水生植物附着生境,细菌群落与植物宿主共同进化,形成复杂的共生体系。例如:隶属于热脱硫杆菌(Desulfobacterota)的电缆细菌可以进行硫化物氧化,同时还原氧气或硝酸盐/亚硝酸盐,然而电缆细菌不是在单个细胞的尺度上耦合硫化氢氧化和氧还原,而是与海草[110]和包括水稻[111]在内的几种植物的生根结构有关并可形成跨越厘米级距离的电子传导通道[112]。因此,尽管关于成种/多样化/突变这一生态学过程在植物附着微生物群落中的贡献仍然缺乏认识,但其对于水生植物附着细菌群落构建也具有不容忽视的影响。
综上所述,尽管多种形式的选择过程塑造了水生植物附着细菌群落,但近年来的研究结果表明,生态漂移、扩散和成种/多样化/突变等过程也影响了水生植物附着细菌群落多样性和组成。然而,由于野外采样通常具有时间和空间随机性,并且在空间上无法精确定义群落边界,可能忽略了水生植物附着细菌群落的时空变化。检测手段的进步以及未来在精细的空间尺度和时间跨度对宿主细菌类群进行原位调查,有助于加强对水生植物附着细菌群落构建过程和机制的进一步理解。
5 水生植物附着细菌群落对湖泊碳氮循环过程的调控作用水生植物叶/根微界面是控制和调节生物体与水体/沉积物之间物质输送和交换的重要途径,也是影响湖泊生态系统碳、氮等元素转化的关键界面,对湖泊生态系统物质循环以及水环境质量的改善具有重要影响(图 2)。此外,水生植物叶/根表面存在的泥沙、碎屑和新型污染物微塑料等也为细菌群落提供附着载体和微环境,这些非生物基质通过自身的物理和化学特性促进细菌定殖,改变附着细菌群落结构和功能,最终影响生态系统中碳氮等生源要素的地球化学循环过程[66, 113]。
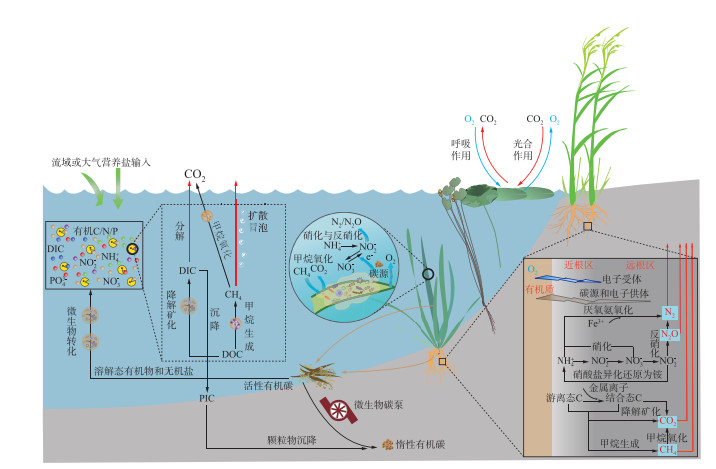
|
图 2 水生植物附着细菌群落参与的湖泊生态系统中的碳、氮循环过程 Fig.2 The aquatic macrophytes epiphytic bacterial communities involved carbon and nitrogen cycling processes in the lake ecosystems |
作为湖泊生态系统中最重要的生源要素之一,碳元素的循环和代谢驱动着湖泊生态系统的基本过程,是湖泊生态系统物质循环和能量流动的主要载体。湖泊生态系统中的各层级动植物、微生物都积极参与到碳转化过程,其中大型水生植物作为湖泊生态系统净生产力的重要组成部分,一方面通过光合作用吸收部分碳后以植物碳和微生物碳形式储存在生态系统中[114],另一方面通过其附着细菌参与甲烷代谢和呼吸作用等多个重要的碳循环过程,从而对湖泊中碳的迁移转化和源汇过程产生重要影响[5]。
一项关于39种植物对CH4原位消耗的比较研究发现:水生植物相较于木本植物、海藻和海草等具有显著更高的甲烷消耗;定量PCR结果表明:水生植物叶际附着细菌群落的pmoA基因丰度最高,主要为I型甲烷氧化菌(γ-变形菌)[115]。Sorrell等对新西兰10个湖泊中13种水生植物表面的甲烷氧化菌活性研究发现:甲烷氧化菌的活性受植物种类和样点营养程度影响,其中富营养水体中沉水植物叶际的甲烷氧化菌活性最高[116]。尽管如此,水生植物在生长过程中封存的碳会伴随着植物的自然衰亡逐渐释放,重新进入生态系统,因此水生植物的叶片凋落物是湖泊生态系统碳和养分循环的重要驱动力[117]。在植物衰亡过程中,水生植物附着细菌群落作为植物与外界物质和能量流动的中间体,既可直接从植物残体中吸收部分营养物质,又能够分解凋落物中富含碳的有机物,产生CO2和CH4,改变终产物向水体和沉积物的动态输入[118]。由于细菌自身的C/N或C/P通常低于植物凋落物,因此在参与植物残体分解过程中对氮、磷等养分的需求较高。研究表明,与挺水植物相比,沉水植物叶片的木质素、纤维素、半纤维素含量较低,但氮、磷含量较高,因此沉水植物在分解过程中其附着细菌群落也更为活跃[119]。
近年来,越来越多的研究强调了微生物碳泵对生态系统中有机碳库周转和长期固持的重要作用[120]。在湖泊生态系统中,水生植物通过光合作用将固定的碳储存在植物根和叶中,然后再以分泌物、脱落物以及凋落物等形式进入湖泊有机碳库。在微生物碳泵的作用下,易分解的水生植物源碳经附着微生物作用逐渐转变为稳定的微生物残体碳,进一步通过续埋效应进入沉积物中,形成稳定碳库,从而促进湖泊生态系统中有机碳的封存[121]。此外,一些金属离子可以通过吸附、络合以及共沉淀等方式结合游离态有机碳,提高湖泊生态系统中有机碳的稳定性[122]。
综上,水生植物在整个生命周期中对湖泊中的碳具有吸收、积累和归还作用,其附着细菌群落通过对碳的分解、转化和同化作用调控碳循环过程,二者在植物生长和衰亡过程中共同推动湖泊生态系统中碳的封存及温室气体的减排。
5.2 水生植物附着细菌群落对湖泊氮循环的影响在湖泊生态系统中,氮素是所有生物体不可缺少的组成元素,同时也是水体富营养化的主要诱因之一,在湖泊营养循环中占据重要地位。自1989年Reddy提出了湿地生态系统中水生植物根—沉积物界面的硝化—反硝化理论以来[123],众多研究聚焦于水生植物根际微域发生的氮转化过程,尤其是脱氮过程。水生植物根际的脱氮主要通过微生物的氨化—硝化—反硝化作用或厌氧氨氧化作用实现,氨化细菌首先将沉积物中的有机态氮降解为铵态氮(NH4+-N),在有氧条件下,氨氧化细菌或古菌进一步将NH4+-N转化为亚硝态氮(NO2--N),随后亚硝酸盐氧化菌将NO2--N转化为硝态氮(NO3--N),最后反硝化菌在缺氧条件下将NO3--N逐步还原为气态氮(N2O或N2),或厌氧氨氧化菌在厌氧条件下将NH4+-N和NO2--N直接转化为N2[124]。与非根际沉积物相比,水生植物根际的氨氧化、反硝化和厌氧氨氧化细菌数量增加了几倍至几十倍[125-126],附着细菌群落的脱氮速率也显著提高,具有更强的脱氮能力[127-128]。这是因为一方面水生植物通过通气组织将氧气运输到根系,引起根际微域的氧化还原波动,促进根际细菌群落进行有机氮矿化,由此产生的无机氮进一步加速了硝化—反硝化作用的耦合[129]。另一方面,根系分泌物含有丰富的营养物质,为脱氮微生物提供可利用碳源,增强了其繁殖能力和活性,使得植物根际具有较高的与氮循环相关的细菌类群丰度[130]。此外,根系分泌的大量小分子有机酸会改变根际微域的pH,促进矿物溶解、活化难溶养分,从而加速根际的脱氮过程[131]。
尽管水生植物根际微生物主导的硝化—反硝化和厌氧氨氧化过程可以显著减少湖泊氮素污染,但是由于氨氧化作用和不完全的反硝化作用会产生温室气体N2O[132-133],加速全球变暖,而厌氧氨氧化微生物可直接生成N2,因此水生植物根际微生物介导的反硝化和厌氧氨氧化对脱氮的相对贡献及其对N2O排放的影响受到广泛关注。Deng等利用15N同位素配对技术调查了长江中下游39个河岸湿地中13种水生植物根际和非根际土壤的脱氮过程,结果表明:根际土壤反硝化和厌氧氨氧化速率均显著高于非根际,土壤的pH和氮素可利用性是影响这两个过程脱氮速率最主要的环境因子[134]。其他研究也发现了反硝化和厌氧氨氧化对于湖泊中水生植物根际微生物介导的脱氮过程的相对贡献受到植物物种[22]、湖泊的富营养化程度[135]、生境中的环境理化[134]等因素影响。此外,硝酸盐异化还原为铵(DNRA)的过程以有机质为电子受体,将NO3--N还原为NH4+-N重新进入水生环境中,成为湖泊氮循环重要的中间过程。在高浓度的有机质和硝态氮环境中,硝酸盐异化还原速率可超过反硝化和厌氧氨氧化[136]。有研究发现芦苇根际土壤中硝酸盐异化还原过程对硝酸盐还原的贡献率高达67.5%,对岸边带土壤硝酸盐还原起到了重要作用[137]。此外,许多水生植物根系表面普遍形成铁膜,由根际细菌驱动的厌氧氨氧化耦合铁还原过程也会强烈影响根际脱氮过程[138]。以上研究表明,在湖泊生态系统,尤其是富营养化水体中,水生植物附着细菌群落是硝化、反硝化、厌氧氨氧化和硝酸盐异化还原为铵等氮素迁移转化过程的重要基础,在减轻湖泊富营养化程度和N2O减排中具有重要作用。
6 研究展望综上所述,得益于多学科的融合和多组学技术的发展, 近十多年关于水生植物附着细菌群落多样性、组成特征、驱动因素、构建机制、生态功能及其与植物宿主的相互作用机制等研究迅速发展。但相比于陆生植物—微生物互作研究,水生植物附着微生物研究起步较晚,研究的广度和深度远远落后。为了更好地理解水生植物及其附着细菌群落形成的“共生功能体”对环境变化的响应机制,我们需要系统探究水生植物附着细菌群落与宿主适应性及生态系统功能之间复杂的相互作用,以及这些细菌类群在植物营养吸收、生长促进和生存维持等方面的生态功能。未来关于水生植物及其附着细菌群落的互作研究,还有以下方面需要深入探讨。
6.1 水生植物附着细菌群落的功能特征与多样性和分类学组成相比,驱动水生植物附着细菌群落功能的因素受到的关注较少,利用多组学技术能够进一步解析水生植物附着细菌群落的多样性模式和分类学特征如何与生态系统功能相关联,具体包括:(ⅰ)利用宏基因组学、转录组学、蛋白质组学和全基因组等组学技术,将细菌类群基因与植物定殖、生理和适应性进行关联分析;(ⅱ)使用高通量分离培养方法,通过特定细菌类群的移除或添加实验验证菌株功能和菌株间的相互作用。此外,除细菌外,水生植物叶/根表面附着多种微生物类群还有真菌、原生动物和微型后生动物等,这些跨界的微生物类群以多营养级微食物网形式共存,对于植物生长健康和功能的维持以及生态系统中的营养物质和能量传递具有重要作用。因此,未来仍需进一步探究水生植物表面不同营养级微生物间的相互作用关系,构建水生植物生态位立体的微食物网结构以及物质和能量传递通路,解析多营养级微生物类群多样性与生态系统功能之间的联系。
6.2 核心菌群挖掘和合成群落应用尽管在多种因素的相互作用下,水生植物叶际和根际形成了多样化的细菌类群,越来越多的研究发现植物存在“核心细菌类群”。由于这些核心细菌类群携带的基因往往具有宿主适应性所必须的功能,因此在植物细菌群落中普遍存在,且在生长激素产生、营养转移和保护植物免受生物和非生物胁迫中发挥关键作用。基于此,水生植物叶际和根际的核心细菌类群可为功能菌株筛选提供理论依据,进一步通过合成群落构建促进植物生长及其生态功能发挥。然而,目前对水生植物核心细菌类群的识别多基于类群的相对丰度和样品中的存在频率,或基于共现网络分析和机器学习等计算方法,植物核心细菌功能与植物生长健康之间的因果关系和互作机制在很大程度上仍未得到探索。未来的研究需要结合高通量培养技术揭示核心细菌的功能特征, 进一步利用合成群落结合无菌体系验证其功能及作用机制,阐明水生植物核心菌株富集的分子机制,以及菌群调控对植物生长发育的影响。
6.3 全球变化背景下水生植物附着细菌群落的响应研究受人类活动和全球气候变化的叠加影响,湖泊表层水体变暖、水体溶解氧降低、富营养化程度加重以及其他污染物输入严重威胁了水生生态系统的生物多样性和生态过程[139-140]。但到目前为止,不同环境驱动因素的交互作用在很大程度上被忽视了,且大多环境扰动下的水生植物附着细菌群落的动态变化主要是基于短期实验,缺乏基于长期监测的研究。全球环境变化因子间通常不是独立的,而是彼此之间相互作用的,两个或多个全球变化因子之间的相互作用可能导致叠加效应或拮抗效应,因此,针对全球变化因素影响下的水生植物及其附着细菌群落互作开展长期实验对阐明水生植物细菌群落应对气候变化的响应及提高植物适应性机制至关重要。
6.4 水生植物—微生物协同降污增汇技术研究水生植物和微生物协同作用可以促进水生生态系统中的水质净化和污染物去除。然而,在污染物浓度较高的生境中,生物或非生物因素胁迫增强,进而影响植物生长并限制附着微生物群落功能发挥,削弱水生植物—微生物协同修复效率。如何通过植物根际界面微生境特征调控微生物群落多样性及功能,从而更好地发挥微生物碳泵作用,将湖泊生态系统中的不稳定态碳进一步转变为稳定态碳,最终通过续埋效应变成稳定碳库封存于沉积物中,形成长期持续增碳作用,达到最优的长效降污增汇效果,是未来研究要重点解决的关键问题之一。
| [1] |
He D, Ren LJ, Xing P et al. Research progress of diversity and community structure of epiphytic bacteria on submerged macrophytes. Chinese Bulletin of Life Sciences, 2014, 26(2): 161-168. [何聃, 任丽娟, 邢鹏等. 沉水植物附着细菌群落结构及其多样性研究进展. 生命科学, 2014, 26(2): 161-168. DOI:10.13376/j.cbls/2014025] |
| [2] |
Hossell JC, Baker JH. A note on the enumeration of epiphytic bacteria by microscopic methods with particular reference to two freshwater plants. Journal of Applied Bacteriology, 1979, 46(1): 87-92. DOI:10.1111/j.1365-2672.1979.tb02584.x |
| [3] |
Allen HL. Primary productivity, chemo-organotrophy, and nutritional interactions of epiphytic algae and bacteria on macrophytes in the littoral of a lake. Ecological Monographs, 1971, 41(2): 97-127. DOI:10.2307/1942387 |
| [4] |
Srivastava JK, Chandra H, Kalra SJS et al. Plant-microbe interaction in aquatic system and their role in the management of water quality: A review. Applied Water Science, 2017, 7(3): 1079-1090. DOI:10.1007/s13201-016-0415-2 |
| [5] |
Wijewardene L, Wu NC, Fohrer N et al. Epiphytic biofilms in freshwater and interactions with macrophytes: Current understanding and future directions. Aquatic Botany, 2022, 176: 103467. DOI:10.1016/j.aquabot.2021.103467 |
| [6] |
张松贺, 韩冰, 庞思等. 沉水植物附着生物膜体系特征与生态功能. 北京: 科学出版社, 2022.
|
| [7] |
Hashidoko Y. Ecochemical studies of interrelationships between epiphytic bacteria and host plants via secondary metabolites. Bioscience Biotechnology and Biochemistry, 2005, 69(8): 1427-1441. DOI:10.1271/bbb.69.1427 |
| [8] |
Huang FM, Sun N, Xie P et al. Research progress on decomposition process of aquatic plants and the influencing factors. Chinese Journal of Ecology, 2023, 42(2): 471-480. [黄法铭, 孙宁, 谢培等. 水生植物的腐解过程及其影响因素研究进展. 生态学杂志, 2023, 42(2): 471-480. DOI:10.13292/j.1000-4890.202302.018] |
| [9] |
Zhang L, Huang SZ, Peng X et al. Potential ecological implication of Cladophora oligoclora decomposition: Characteristics of nutrient migration, transformation, and response of bacterial community structure. Water Research, 2021, 190: 116741. DOI:10.1016/j.watres.2020.116741 |
| [10] |
Kidd-Haack S, Bitton G, Laabes D. Epiphytic bacteria: Development of a method for determining respiring bacteria on leaves. Journal of Applied Bacteriology, 1985, 59(6): 545-548. DOI:10.1111/j.1365-2672.1985.tb03358.x |
| [11] |
Hempel M, Blume M, Blindow I et al. Epiphytic bacterial community composition on two common submerged macrophytes in brackish water and freshwater. BMC Microbiology, 2008, 8: 58. DOI:10.1186/1471-2180-8-58 |
| [12] |
Chand T, Harris RF, Andrews JH. Enumeration and characterization of bacterial colonists of a submersed aquatic plant, Eurasian watermilfoil (Myriophyllum spicatum L.). Applied and Environmental Microbiology, 1992, 58(10): 3374-3379. DOI:10.1128/aem.58.10.3374-3379.1992 |
| [13] |
Tanaka Y, Tamaki H, Matsuzawa H et al. Microbial community analysis in the roots of aquatic plants and isolation of novel microbes including an organism of the candidate phylum OP10. Microbes and Environments, 2012, 27(2): 149-157. DOI:10.1264/jsme2.me11288 |
| [14] |
He D, Ren LJ, Wu QL. Growing season drives the compositional changes and assembly processes of epiphytic bacterial communities of two submerged macrophytes in Taihu Lake. FEMS Microbiology Ecology, 2020, 96(4): fiaa025. DOI:10.1093/femsec/fiaa025 |
| [15] |
Huang R, Zeng J, Zhao DY et al. Sediment microbiomes associated with the rhizosphere of emergent macrophytes in a shallow, subtropical lake. Limnology and Oceanography, 2020, 65(1): S38-S48. DOI:10.1002/lno.11325 |
| [16] |
Bulgarelli D, Schlaeppi K, Spaepen S et al. Structure and functions of the bacterial microbiota of plants. Annual Review of Plant Biology, 2013, 64: 807-838. DOI:10.1146/annurev-arplant-050312-120106 |
| [17] |
Reinhold-Hurek B, Bünger W, Burbano CS et al. Roots shaping their microbiome: Global hotspots for microbial activity. Annual Review of Phytopathology, 2015, 53: 403-424. DOI:10.1146/annurev-phyto-082712-102342 |
| [18] |
He RJ, Zeng J, Zhao DY et al. Contrasting patterns in diversity and community assembly of Phragmites australis root-associated bacterial communities from different seasons. Applied and Environmental Microbiology, 2020, 86(14): e00379-e00320. DOI:10.1128/AEM.00379-20 |
| [19] |
He RJ, Zeng J, Zhao DY et al. Decreased spatial variation and deterministic processes of bacterial community assembly in the rhizosphere of Phragmites australis across the middle-lower Yangtze Plain. Molecular Ecology, 2022, 31(4): 1180-1195. DOI:10.1111/mec.16298 |
| [20] |
Zhao LY, Tao JX, Liu M. Effects of aquatic macrophyte planting on functional diversity of microbial community in sediment. Journal of Yangtze River Scientific Research Institute, 2015, 32(6): 81-86. [赵良元, 陶晶祥, 刘敏. 水生植物对沉积物微生物群落功能多样性的影响. 长江科学院院报, 2015, 32(6): 81-86. DOI:10.3969/j.issn.1001-5485.2015.06.015] |
| [21] |
Hong YW, Liao D, Hu AY et al. Diversity of endophytic and rhizoplane bacterial communities associated with exotic Spartina alterniflora and native mangrove using Illumina amplicon sequencing. Canadian Journal of Microbiology, 2015, 61(10): 723-733. DOI:10.1139/cjm-2015-0079 |
| [22] |
Yin XJ, Lu J, Wang YC et al. The abundance of nirS-type denitrifiers and anammox bacteria in rhizospheres was affected by the organic acids secreted from roots of submerged macrophytes. Chemosphere, 2020, 240: 124903. DOI:10.1016/j.chemosphere.2019.124903 |
| [23] |
Wang XL, Wang MX, Xie XG et al. An amplification-selection model for quantified rhizosphere microbiota assembly. Science Bulletin, 2020, 65(12): 983-986. DOI:10.1016/j.scib.2020.03.005 |
| [24] |
He D, Ren LJ, Wu QL. Contrasting diversity of epibiotic bacteria and surrounding bacterioplankton of a common submerged macrophyte, Potamogeton crispus, in freshwater lakes. FEMS Microbiology Ecology, 2014, 90(3): 551-562. DOI:10.1111/1574-6941.12414 |
| [25] |
Ang ZQ, Sun XJ, Cao XY et al. Diversity and network structure of epiphytic bacterial communities on different submerged macrophytes. J Lake Sci, 2020, 34(4): 1234-1249. [昂正强, 孙晓健, 曹新益等. 不同沉水植物叶片附着细菌群落多样性及网络结构差异. 湖泊科学, 2020, 34(4): 1234-1249. DOI:10.18307/2022.0416] |
| [26] |
Levi PS, Starnawski P, Poulsen B et al. Microbial community diversity and composition varies with habitat characteristics and biofilm function in macrophyte-rich streams. Oikos, 2017, 126(3): 398-409. DOI:10.1111/oik.03400 |
| [27] |
He RJ, Hu SW, Li QS et al. Greater transmission capacities and small-world characteristics of bacterial communities in the above- than those in the below-ground niches of a typical submerged macrophyte, Vallisneria natans. Science of the Total Environment, 2023, 903: 166229. DOI:10.1016/j.scitotenv.2023.166229 |
| [28] |
Battin TJ, Wille A, Sattler B et al. Phylogenetic and functional heterogeneity of sediment biofilms along environmental gradients in a glacial stream. Applied and Environmental Microbiology, 2001, 67(2): 799-807. DOI:10.1128/AEM.67.2.799-807.2001 |
| [29] |
Besemer K. Biodiversity, community structure and function of biofilms in stream ecosystems. Research in Microbiology, 2015, 166(10): 774-781. DOI:10.1016/j.resmic.2015.05.006 |
| [30] |
Newton RJ, Jones SE, Eiler A et al. A guide to the natural history of freshwater lake bacteria. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14-49. DOI:10.1128/MMBR.00028-10 |
| [31] |
Anderson-Glenna MJ, Bakkestuen V, Clipson NJW. Spatial and temporal variability in epilithic biofilm bacterial communities along an upland river gradient. FEMS Microbiology Ecology, 2008, 64(3): 407-418. DOI:10.1111/j.1574-6941.2008.00480.x |
| [32] |
Bartrons M, Catalan J, Casamayor EO. High bacterial diversity in epilithic biofilms of oligotrophic mountain lakes. Microbial Ecology, 2012, 64(4): 860-869. DOI:10.1007/s00248-012-0072-4 |
| [33] |
Chen ZH, Dolfing J, Zhuang SY et al. Periphytic biofilms-mediated microbial interactions and their impact on the nitrogen cycle in rice paddies. Eco-Environment & Health, 2022, 1(3): 172-180. DOI:10.1016/j.eehl.2022.09.004 |
| [34] |
Sand-Jensen K, Borum J. Interactions among phytoplankton, periphyton, and macrophytes in temperate freshwaters and estuaries. Aquatic Botany, 1991, 41(1/2/3): 137-175. DOI:10.1016/0304-3770(91)90042-4 |
| [35] |
Liu XD, Zhu HZ, Jiang MZ et al. Characteristics of the rhizosphere and phyllosphere microbial community of three submerged plants in the dragon-shaped water system of Beijing. Chinese Journal of Biotechnology, 2021, 37(10): 3663-3674. [柳晓东, 朱海珍, 姜民志等. 北京龙形水系三种沉水植物根际及叶际微生物群落特征. 生物工程学报, 2021, 37(10): 3663-3674. DOI:10.13345/j.cjb.210415] |
| [36] |
Yu WC, Li JH, Ma XW et al. Community structure and function of epiphytic bacteria attached to three submerged macrophytes. Science of the Total Environment, 2022, 835: 155546. DOI:10.1016/j.scitotenv.2022.155546 |
| [37] |
Zhang XM, He RJ, Su R et al. Composition and co-occurrence network of the rhizosphere bacterial community of two emergent macrophytes and implications for phytoremediation. Marine and Freshwater Research, 2021, 72(7): 1053-1064. DOI:10.1071/MF20082 |
| [38] |
Ling N, Wang TT, Kuzyakov Y. Rhizosphere bacteriome structure and functions. Nature Communications, 2022, 13(1): 836. DOI:10.1038/s41467-022-28448-9 |
| [39] |
Gribben PE, Nielsen S, Seymour JR et al. Microbial communities in marine sediments modify success of an invasive macrophyte. Scientific Reports, 2017, 7: 9845. DOI:10.1038/s41598-017-10231-2 |
| [40] |
Zhou Q, Zhang XM, He RJ et al. The composition and assembly of bacterial communities across the rhizosphere and phyllosphere compartments of Phragmites australis. Diversity, 2019, 11(6): 98. DOI:10.3390/d11060098 |
| [41] |
Lukwambe B, Zhao L, Nicholaus R et al. Bacterioplankton community in response to biological filters (clam, biofilm, and macrophytes) in an integrated aquaculture wastewater bioremediation system. Environmental Pollution, 2019, 254: 113035. DOI:10.1016/j.envpol.2019.113035 |
| [42] |
Song ZL, Huang YY, Liu Q et al. Discovering the characteristics of community structures and functional properties of epiphytic bacteria on Spartina alterniflora in the coastal salt marsh area. Journal of Marine Science and Engineering, 2022, 10(12): 1981. DOI:10.3390/jmse10121981 |
| [43] |
Han B, Zhang SH, Zhang LS et al. Characterization of microbes and denitrifiers attached to two species of floating plants in the wetlands of Lake Taihu. PLoS One, 2018, 13(11): e0207443. DOI:10.1371/journal.pone.0207443 |
| [44] |
Liu Q, Zhang HC, Chang FQ et al. eDNA revealed in situ microbial community changes in response to Trapa japonica in Lake Qionghai and Lake Erhai, southwestern China. Chemosphere, 2022, 288(Pt3): 132605. DOI:10.1016/j.chemosphere.2021.132605 |
| [45] |
Pang S, Zhang SH, Lv XY et al. Characterization of bacterial community in biofilm and sediments of wetlands dominated by aquatic macrophytes. Ecological Engineering, 2016, 97: 242-250. DOI:10.1016/j.ecoleng.2016.10.011 |
| [46] |
Manirakiza B, Zhang SH, Addo FG et al. Exploring microbial diversity and ecological function of epiphytic and surface sediment biofilm communities in a shallow tropical lake. Science of the Total Environment, 2022, 808: 151821. DOI:10.1016/j.scitotenv.2021.151821 |
| [47] |
Rotini A, Mejia AY, Costa R et al. Ecophysiological plasticity and bacteriome shift in the seagrass Halophila stipulacea along a depth gradient in the northern red sea. Frontiers in Plant Science, 2017, 7: 2015. DOI:10.3389/fpls.2016.02015 |
| [48] |
Ahmed A, Khurshid A, Tang XH et al. Structural and functional impacts of microbiota on Pyropia yezoensis and surrounding seawater in cultivation farms along coastal areas of the Yellow Sea. Microorganisms, 2021, 9(6): 1291. DOI:10.3390/microorganisms9061291 |
| [49] |
Gordon-Bradley N, Li N, Williams HN. Bacterial community structure in freshwater springs infested with the invasive plant species Hydrilla verticillata. Hydrobiologia, 2015, 742(1): 221-232. DOI:10.1007/s10750-014-1988-0 |
| [50] |
Dobretsov S, Véliz K, Romero MS et al. Impact of UV radiation on the red seaweed Gelidium lingulatum and its associated bacteria. European Journal of Phycology, 2021, 56(2): 129-141. DOI:10.1080/09670262.2020.1775309 |
| [51] |
Lozada M, Diéguez MC, García PE et al. Undaria pinnatifida exudates trigger shifts in seawater chemistry and microbial communities from Atlantic Patagonian coasts. Biological Invasions, 2021, 23(6): 1781-1801. DOI:10.1007/s10530-021-02471-1 |
| [52] |
Xia PH, Yan DB, Sun RG et al. Community composition and correlations between bacteria and algae within epiphytic biofilms on submerged macrophytes in a plateau lake, southwest China. Science of the Total Environment, 2020, 727: 138398. DOI:10.1016/j.scitotenv.2020.138398 |
| [53] |
Sun L, Wang JS, Wu YY et al. Community structure and function of epiphytic bacteria associated with Myriophyllum spicatum in Baiyangdian Lake, China. Frontiers in Microbiology, 2021, 12: 705509. DOI:10.3389/fmicb.2021.705509 |
| [54] |
Geng N, Xia YF, Lu DB et al. The bacterial community structure in epiphytic biofilm on submerged macrophyte Potamogetom crispus L. and its contribution to heavy metal accumulation in an urban industrial area in Hangzhou. Journal of Hazardous Materials, 2022, 430: 128455. DOI:10.1016/j.jhazmat.2022.128455 |
| [55] |
Shi L, Xia PH, Lin T et al. Temporal succession of bacterial community structure, co-occurrence patterns, and community assembly process in epiphytic biofilms of submerged plants in a plateau lake. Microbial Ecology, 2023, 85(1): 87-99. DOI:10.1007/s00248-021-01956-9 |
| [56] |
Ma J, Shi RJ, Han RM et al. Community structure of epiphytic bacteria on Potamogeton pectinatus and the surrounding bacterioplankton in Hongze Lake. Marine and Freshwater Research, 2021, 72(7): 997-1003. DOI:10.1071/MF20212 |
| [57] |
Lu SJ, He RJ, Zhao DY et al. Effects of shading levels on the composition and co-occurrence patterns of bacterioplankton and epibiotic bacterial communities of Cabomba caroliniana. Science of the Total Environment, 2021, 785: 147286. DOI:10.1016/j.scitotenv.2021.147286 |
| [58] |
Hernández-Del Amo E, Dolinová I, La Ramis-Jorba G et al. Limited effect of radial oxygen loss on ammonia oxidizers in Typha angustifolia root hairs. Scientific Reports, 2020, 10(1): 15694. DOI:10.1038/s41598-020-72653-9 |
| [59] |
Ge YY, Wang QL, Wang L et al. Response of soil enzymes and microbial communities to root extracts of the alien Alternanthera philoxeroides. Archives of Agronomy and Soil Science, 2018, 64(5): 708-717. DOI:10.1080/03650340.2017.1373186 |
| [60] |
Wang WG, Zhao YH, Jiang GM et al. The nutrient removal ability and microbial communities in a pilot-scale horizontal subsurface flow constructed wetland fed by slightly polluted lake water. Wetlands, 2020, 40(6): 2085-2096. DOI:10.1007/s13157-020-01327-z |
| [61] |
Fu WG, Wang YX, Wei W et al. Species diversity and functional prediction of soil bacterial communities in constructed wetlands with different plant conditions. Current Microbiology, 2019, 76(3): 338-345. DOI:10.1007/s00284-019-01634-7 |
| [62] |
Wang C, Liu SY, Zhang Y et al. Effects of planted versus naturally growing Vallisneria natans on the sediment microbial community in West Lake, China. Microbial Ecology, 2017, 74(2): 278-288. DOI:10.1007/s00248-017-0951-9 |
| [63] |
Crump BC, Wojahn JM, Tomas F et al. Metatranscriptomics and amplicon sequencing reveal mutualisms in seagrass microbiomes. Frontiers in Microbiology, 2018, 9: 388. DOI:10.3389/fmicb.2018.00388 |
| [64] |
Jiao CC, Zhao DY, Zeng J et al. Eutrophication in subtropical lakes reinforces the dominance of balanced-variation component in temporal bacterioplankton community heterogeneity by lessening stochastic processes. FEMS Microbiology Ecology, 2022, 98(6): fiac051. DOI:10.1093/femsec/fiac051 |
| [65] |
Muller DB, Vogel C, Bai Y et al. The plant microbiota: systems-level insights and perspectives. Annual Review of Genetics, 2016, 50: 211-234. DOI:10.1146/annurev-genet-120215-034952 |
| [66] |
Wang WL, Liu B, Han RM et al. Research advancements and perspectives on leaf and stem micro-interfaces in submerged macrophytes and its effect on water nitrogen cycling. Acta Ecologica Sinica, 2014, 34(22): 6409-6416. [王文林, 刘波, 韩睿明等. 沉水植物茎叶微界面及其对水体氮循环影响研究进展. 生态学报, 2014, 34(22): 6409-6416. DOI:10.5846/stxb201308152085] |
| [67] |
Hu SW, He RJ, He XW et al. Niche-specific restructuring of bacterial communities associated with submerged macrophyte under ammonium stress. Applied and Environmental Microbiology, 2023, 89(7): e0071723. DOI:10.1128/aem.00717-23 |
| [68] |
Cordovez V, Dini-Andreote F, Carrión VJ et al. Ecology and evolution of plant microbiomes. Annual Review of Microbiology, 2019, 73: 69-88. DOI:10.1146/annurev-micro-090817-062524 |
| [69] |
Bowen JL, Kearns PJ, Byrnes JEK et al. Lineage overwhelms environmental conditions in determining rhizosphere bacterial community structure in a cosmopolitan invasive plant. Nature Communications, 2017, 8: 433. DOI:10.1038/s41467-017-00626-0 |
| [70] |
Lu SL, Zhang C, Xu JW. Root exudates of wetland plants and the influence on the microbial community in constructed wetlands. Ecology and Environmental Science, 2011, 20(4): 676-680. [陆松柳, 张辰, 徐俊伟. 植物根系分泌物分析及对湿地微生物群落的影响研究. 生态环境学报, 2011, 20(4): 676-680.] |
| [71] |
杨瑞. 挺水植物根际微生物生态研究[学位论文]. 天津: 天津大学, 2012.
|
| [72] |
Hempel M, Grossart HP, Gross EM. Community composition of bacterial biofilms on two submerged macrophytes and an artificial substrate in a pre-alpine lake. Aquatic Microbial Ecology, 2009, 58: 79-94. DOI:10.3354/ame01353 |
| [73] |
Chen KN, Qiang S, Li WC et al. Studies on reproduction diversity in Potamogeton petinatus. Acta Phytoecologica Sinica, 2003, 27(5): 672-676. [陈开宁, 强胜, 李文朝等. 蓖齿眼子菜繁殖多样性研究. 植物生态学报, 2003, 27(5): 672-676. DOI:10.17521/cjpe.2003.0098] |
| [74] |
Xu NH, Zhao QQ, Zhang ZY et al. Phyllosphere microorganisms: Sources, drivers, and their interactions with plant hosts. Journal of Agricultural and Food Chemistry, 2022, 70(16): 4860-4870. DOI:10.1021/acs.jafc.2c01113 |
| [75] |
Chaparro JM, Badri DV, Vivanco JM. Rhizosphere microbiome assemblage is affected by plant development. The ISME Journal, 2014, 8(4): 790-803. DOI:10.1038/ismej.2013.196 |
| [76] |
马杰. 浅水湖泊典型沉水植物附着细菌群落结构特征[学位论文]. 南京: 南京师范大学, 2016.
|
| [77] |
范婤. 太湖典型沉水植物附着细菌及功能微生物多样性研究[学位论文]. 南京: 南京师范大学, 2016.
|
| [78] |
Sun LH, Chang YJ, Han SQ et al. Metabolomics analysis of bioactive compounds in culture water planting floating bed water dropwort in different development stages. Research of Environmental Sciences, 2021, 34(8): 1860-1875. [孙林鹤, 常雅军, 韩士群等. 浮床水芹不同生长阶段水体活性物质代谢组学分析. 环境科学研究, 2021, 34(8): 1860-1875. DOI:10.13198/j.issn.1001-6929.2021.03.04] |
| [79] |
Philippot L, Raaijmakers JM, Lemanceau P et al. Going back to the roots: The microbial ecology of the rhizosphere. Nature Reviews Microbiology, 2013, 11: 789-799. DOI:10.1038/nrmicro3109 |
| [80] |
Han B, Zhang SH, Wang PF et al. Effects of water flow on submerged macrophyte-biofilm systems in constructed wetlands. Scientific Reports, 2018, 8: 2650. DOI:10.1038/s41598-018-21080-y |
| [81] |
Li HM, Li Q, Luo X et al. Responses of the submerged macrophyte Vallisneria natans to a water depth gradient. Science of the Total Environment, 2020, 701: 134944. DOI:10.1016/j.scitotenv.2019.134944 |
| [82] |
Zhao YH, Xiong X, Wu CX et al. Influence of light and temperature on the development and denitrification potential of periphytic biofilms. Science of the Total Environment, 2018, 613/614: 1430-1437. DOI:10.1016/j.scitotenv.2017.06.117 |
| [83] |
Hu SW, He RJ, Zeng J et al. Lower compositional variation and higher network complexity of rhizosphere bacterial community in constructed wetland compared to natural wetland. Microbial Ecology, 2023, 85(3): 965-979. DOI:10.1007/s00248-022-02040-6 |
| [84] |
Yan LL, Mu XY, Han B et al. Ammonium loading disturbed the microbial food webs in biofilms attached to submersed macrophyte Vallisneria natans. Science of the Total Environment, 2019, 659: 691-698. DOI:10.1016/j.scitotenv.2018.12.423 |
| [85] |
Gong LX, Zhang SH, Chen DQ et al. Response of biofilms-leaves of two submerged macrophytes to high ammonium. Chemosphere, 2018, 192: 152-160. DOI:10.1016/j.chemosphere.2017.09.147 |
| [86] |
Zhang ZQ, Chen HZ, Mu XY et al. Nitrate application decreased microbial biodiversity but stimulated denitrifiers in epiphytic biofilms on Ceratophyllum demersum. Journal of Environmental Management, 2020, 269: 110814. DOI:10.1016/j.jenvman.2020.110814 |
| [87] |
Mu XY, Zhang SH, Lv X et al. Water flow and temperature drove epiphytic microbial community shift: Insight into nutrient removal in constructed wetlands from microbial assemblage and co-occurrence patterns. Bioresource Technology, 2021, 332: 125134. DOI:10.1016/j.biortech.2021.125134 |
| [88] |
Xu D, Xia Y, Li ZX et al. The influence of flow rates and water depth gradients on the growth process of submerged macrophytes and the biomass composition of the phytoplankton assemblage in eutrophic water: An analysis based on submerged macrophytes photosynthesis parameters. Environmental Science and Pollution Research, 2020, 27(25): 31477-31488. DOI:10.1007/s11356-020-09404-w |
| [89] |
Esposito C, Nijman TPA, Veraart AJ et al. Activity and abundance of methane-oxidizing bacteria on plants in experimental lakes subjected to different nutrient and warming treatments. Aquatic Botany, 2023, 185: 103610. DOI:10.1016/j.aquabot.2022.103610 |
| [90] |
Dong B, Han RM, Wang GX. Research advances in and perspectives on characteristics of the micro-boundary layer around leaves and stems of submerged macrophytes. Acta Ecologica Sinica, 2017, 37(6): 1769-1776. [董彬, 韩睿明, 王国祥. 沉水植物茎叶微界面特性研究进展. 生态学报, 2017, 37(6): 1769-1776. DOI:10.5846/stxb201511112288] |
| [91] |
董彬. 淡水附植生物的组成结构特征及其生态功能. 北京: 化学工业出版社, 2018.
|
| [92] |
Han C, Ren JH, Wang ZD et al. A novel hybrid sensor for combined imaging of dissolved oxygen and labile phosphorus flux in sediment and water. Water Research, 2017, 108: 179-188. DOI:10.1016/j.watres.2016.10.075 |
| [93] |
Hassani MA, Durán P, Hacquard S. Microbial interactions within the plant holobiont. Microbiome, 2018, 6(1): 58. DOI:10.1186/s40168-018-0445-0 |
| [94] |
Trivedi P, Leach JE, Tringe SG et al. Plant-microbiome interactions: From community assembly to plant health. Nature Reviews Microbiology, 2020, 18: 607-621. DOI:10.1038/s41579-020-0412-1 |
| [95] |
Rodriguez PA, Rothballer M, Chowdhury SP et al. Systems biology of plant-microbiome interactions. Molecular Plant, 2019, 12(6): 804-821. DOI:10.1016/j.molp.2019.05.006 |
| [96] |
赵晓芬. 几种湿地植物根际微生物的分离鉴定及其污水净化效果的研究[学位论文]. 青岛: 中国海洋大学, 2012.
|
| [97] |
Ishizawa H, Tada M, Kuroda M et al. Synthetic bacterial community of duckweed: A simple and stable system to study plant-microbe interactions. Microbes and Environments, 2020, 35(4): ME20112. DOI:10.1264/jsme2.ME20112 |
| [98] |
Barberán A, Bates ST, Casamayor EO et al. Using network analysis to explore co-occurrence patterns in soil microbial communities. The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [99] |
Banerjee S, Schlaeppi K, van der Heijden MGA. Keystone taxa as drivers of microbiome structure and functioning. Nature Reviews Microbiology, 2018, 16(9): 567-576. DOI:10.1038/s41579-018-0024-1 |
| [100] |
Fitzpatrick CR, Salas-González I, Conway JM et al. The plant microbiome: From ecology to reductionism and beyond. Annual Review of Microbiology, 2020, 74: 81-100. DOI:10.1146/annurev-micro-022620-014327 |
| [101] |
Hanson CA, Fuhrman JA, Horner-Devine MC et al. Beyond biogeographic patterns: Processes shaping the microbial landscape. Nature Reviews Microbiology, 2012, 10: 497-506. DOI:10.1038/nrmicro2795 |
| [102] |
Vellend M. Conceptual synthesis in community ecology. Quarterly Review of Biology, 2010, 85(2): 183-206. DOI:10.1086/652373 |
| [103] |
Yan LY, Zhang SH, Lin D et al. Nitrogen loading affects microbes, nitrifiers and denitrifiers attached to submerged macrophyte in constructed wetlands. Science of the Total Environment, 2018, 622/623: 121-126. DOI:10.1016/j.scitotenv.2017.11.234 |
| [104] |
Zhou Q, He RJ, Zhao DY et al. Contrasting patterns of the resident and active rhizosphere bacterial communities of Phragmites australis. Microbial Ecology, 2022, 83(2): 314-327. DOI:10.1007/s00248-021-01767-y |
| [105] |
Stegen JC, Lin XJ, Fredrickson JK et al. Estimating and mapping ecological processes influencing microbial community assembly. Frontiers in Microbiology, 2015, 6: 370. DOI:10.3389/fmicb.2015.00370 |
| [106] |
Li GQ, Yan DB, Xia PH et al. Community structure and assembly of denitrifying bacteria in epiphytic biofilms in a freshwater lake ecosystem. Journal of Oceanology and Limnology, 2022, 40(3): 1039-1050. DOI:10.1007/s00343-021-1084-z |
| [107] |
Gao C, Guo LD. Progress on microbial species diversity, community assembly and functional traits. Biodiversity Science, 2022, 30: 22429. [高程, 郭良栋. 微生物物种多样性、群落构建与功能性状研究进展. 生物多样性, 2022, 30: 22429. DOI:10.17520/biods.2022429] |
| [108] |
Qin ZR, Zhao ZH, Xia LL et al. Unraveling the ecological mechanisms of bacterial succession in epiphytic biofilms on Vallisneria natans and Hydrilla verticillata during bioremediation of phenanthrene and pyrene polluted wetland. Journal of Environmental Management, 2022, 321: 115986. DOI:10.1016/j.jenvman.2022.115986 |
| [109] |
Bai CR, Gao G, Tang XM et al. Contrasting diversity patterns and community assembly mechanisms of bacterioplankton among different aquatic habitats in Lake Taihu, a large eutrophic shallow lake in China. Environmental Pollution, 2022, 315: 120342. DOI:10.1016/j.envpol.2022.120342 |
| [110] |
Martin BC, Bougoure J, Ryan MH et al. Oxygen loss from seagrass roots coincides with colonisation of sulphide-oxidising cable bacteria and reduces sulphide stress. The ISME Journal, 2019, 13(3): 707-719. DOI:10.1038/s41396-018-0308-5 |
| [111] |
Scholz VV, Müller H, Koren K et al. The rhizosphere of aquatic plants is a habitat for cable bacteria. FEMS Microbiology Ecology, 2019, 95(6): fiz062. DOI:10.1093/femsec/fiz062 |
| [112] |
Malkin S, Cardini U. Facilitative interactions on the rise: Cable bacteria associate with diverse aquatic plants. New Phytologist, 2021, 232(5): 1897-1900. DOI:10.1111/nph.17664 |
| [113] |
邢书语. 水生植物体表及非生物基质表面附着生物膜氮循环功能微生物分布特征研究[学位论文]. 镇江: 江苏大学, 2021.
|
| [114] |
吴振斌. 水生植物与水体生态修复. 北京: 科学出版社, 2011.
|
| [115] |
Yoshida N, Iguchi H, Yurimoto H et al. Aquatic plant surface as a niche for methanotrophs. Frontiers in Microbiology, 2014, 5: 30. DOI:10.3389/fmicb.2014.00030 |
| [116] |
Sorrell BK, Downes MT, Stanger CL. Methanotrophic bacteria and their activity on submerged aquatic macrophytes. Aquatic Botany, 2002, 72(2): 107-119. DOI:10.1016/s0304-3770(01)00215-7 |
| [117] |
Baldy V, Gobert V, Guerold F et al. Leaf litter breakdown budgets in streams of various trophic status: Effects of dissolved inorganic nutrients on microorganisms and invertebrates. Freshwater Biology, 2007, 52(7): 1322-1335. DOI:10.1111/j.1365-2427.2007.01768.x |
| [118] |
Hayer M, Wymore AS, Hungate BA et al. Microbes on decomposing litter in streams: Entering on the leaf or colonizing in the water?. The ISME Journal, 2022, 16(3): 717-725. DOI:10.1038/s41396-021-01114-6 |
| [119] |
Cao PP, Liu MS, Tang JY et al. A comparative study on the decomposition processes among some aquatic plants. Acta Ecologica Sinica, 2014, 34(14): 3848-3858. [曹培培, 刘茂松, 唐金艳等. 几种水生植物腐解过程的比较研究. 生态学报, 2014, 34(14): 3848-3858. DOI:10.5846/stxb201212011724] |
| [120] |
Liang C, Schimel JP, Jastrow JD. The importance of anabolism in microbial control over soil carbon storage. Nature Microbiology, 2017, 2(8): 17105. DOI:10.1038/nmicrobiol.2017.105 |
| [121] |
Whalen ED, Grandy AS, Sokol NW et al. Clarifying the evidence for microbial- and plant-derived soil organic matter, and the path toward a more quantitative understanding. Global Change Biology, 28(24): 7167-7185. DOI:10.1111/gcb.16413 |
| [122] |
Zhong ZL, Li C, Chen MS et al. The root tip of submerged plants: An efficient engine for carbon mineralization. Environmental Science & Technology Letters, 2023, 10(4): 385-390. DOI:10.1021/acs.estlett.3c00065 |
| [123] |
Reddy KR, Patrick Jr WH, Lindau CW. Nitrification-denitrification at the plant root-sediment interface in wetlands. Limnology and Oceanography, 1989, 34(6): 1004-1013. DOI:10.4319/lo.1989.34.6.1004 |
| [124] |
Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16: 263-276. DOI:10.1038/nrmicro.2018.9 |
| [125] |
Huang R, Zeng J, Zhao DY et al. Co-association of two nir denitrifiers under the influence of emergent macrophytes. Microbial Ecology, 2020, 80(4): 809-821. DOI:10.1007/s00248-020-01545-2 |
| [126] |
Zhao JW, Xu YF, Peng L et al. Diversity of anammox bacteria and abundance of functional genes for nitrogen cycling in the rhizosphere of submerged macrophytes in a freshwater lake in summer. Journal of Soils and Sediments, 2019, 19(10): 3648-3656. DOI:10.1007/s11368-019-02340-4 |
| [127] |
Su R, Huang R, Zeng J et al. Rhizosphere-associated nosZII microbial community of Phragmites australis and its influence on nitrous oxide emissions in two different regions. Journal of Soils and Sediments, 2021, 21(10): 3326-3341. DOI:10.1007/s11368-021-02967-2 |
| [128] |
严定波. 沉水植物附着生物膜细菌及反硝化菌群落多样性研究[学位论文]. 贵州: 贵州师范大学, 2020.
|
| [129] |
Bezbaruah AN, Zhang TC. pH, redox, and oxygen microprofiles in rhizosphere of bulrush (Scirpus validus) in a constructed wetland treating municipal wastewater. Biotechnology and Bioengineering, 2004, 88(1): 60-70. DOI:10.1002/bit.20208 |
| [130] |
Li X, Li YY, Wu JS. Different in root exudates and rhizosphere microorganisms effect on nitrogen removal between three emergent aquatic plants in surface flow constructed wetlands. Chemosphere, 2023, 337: 139422. DOI:10.1016/j.chemosphere.2023.139422 |
| [131] |
杨诗卡. 水生植物根系有机酸驱动沉积物氮、磷转化机制[学位论文]. 西安: 西安工业大学, 2019.
|
| [132] |
Ravishankara AR, Daniel JS, Portmann RW. Nitrous oxide (N2O): The dominant ozone-depleting substance emitted in the 21st century. Science, 2009, 326(5949): 123-125. DOI:10.1126/science.1176985 |
| [133] |
Hallin S, Philippot L, Löffler FE et al. Genomics and ecology of novel N2O-reducing microorganisms. Trends in Microbiology, 2018, 26(1): 43-55. DOI:10.1016/j.tim.2017.07.003 |
| [134] |
Deng DL, Ding BJ, He G et al. The contribution of anammox to nitrogen removal is greater in bulk soils than in rhizosphere soils in riparian wetlands along the Yangtze River. Global Biogeochemical Cycles, 2023, 37(5): e2022GB007576. DOI:10.1029/2022GB007576 |
| [135] |
Xu YF, Lu J, Huang SS et al. Submerged plants alleviated the impacts of increased ammonium pollution on anammox bacteria and nirS denitrifiers in the rhizosphere. Environmental Science and Pollution Research, 2021, 28(41): 58755-58767. DOI:10.1007/s11356-021-14715-7 |
| [136] |
Wang SY, Pi YX, Song YP et al. Hotspot of dissimilatory nitrate reduction to ammonium (DNRA) process in freshwater sediments of riparian zones. Water Research, 2020, 173: 115539. DOI:10.1016/j.watres.2020.115539 |
| [137] |
Wang SY, Pi YX, Jiang YY et al. Nitrate reduction in the reed rhizosphere of a riparian zone: From functional genes to activity and contribution. Environmental Research, 2020, 180: 108867. DOI:10.1016/j.envres.2019.108867 |
| [138] |
Li XF, Hou LJ, Liu M et al. Evidence of nitrogen loss from anaerobic ammonium oxidation coupled with ferric iron reduction in an intertidal wetland. Environmental Science and Technology, 2015, 49(19): 11560-11568. DOI:10.1021/acs.est.5b03419 |
| [139] |
Woolway RI, Kraemer BM, Lenters JD et al. Global lake responses to climate change. Nature Reviews Earth & Environment, 2020, 1: 388-403. DOI:10.1038/s43017-020-0067-5 |
| [140] |
Pan YJ, García-Girón J, Iversen LL. Global change and plant-ecosystem functioning in freshwaters. Trends in Plant Science, 2023, 28(6): 646-660. DOI:10.1016/j.tplants.2022.12.013 |
 2024, Vol. 36
2024, Vol. 36 


