(2: 中国科学院南京地理与湖泊研究所, 湖泊与环境国家重点实验室, 南京 210008)
(3: 中国科学院抚仙湖高原深水湖泊研究站, 玉溪 652500)
(4: 河北大学生命科学学院, 保定 071002)
(5: 中国科学院大学, 北京 100049)
(6: 中国科学院大学中丹学院, 北京 100049)
(2: State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, P. R. China)
(3: The Fuxianhu Station of Plateau Deep Lake Research, Chinese Academy of Sciences, Yuxi 652500, P. R. China)
(4: School of Life Sciences, Hebei University, Baoding 071002, P. R. China)
(5: University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
(6: Sino-Danish College, University of Chinese Academy of Sciences, Beijing 100049, P. R. China)
浮游植物种类多、数量大、分布广,是湖泊生态系统的重要组成部分,监测其物种多样性和群落组成是了解湖泊生态功能的关键。浮游植物是湖泊生态系统中重要的初级生产者,物种多样性高且对水环境变化敏感,其多样性和群落组成的变化是反映湖泊环境梯度和生态系统特征的重要指标[1]。浮游植物的物种组成能够反映水体的温度、营养盐,甚至集团内捕食等营养级关系的变化。Da等利用黄颡鱼(Pelteobagrus fulvidraco)和秀丽白虾(Exopalaemon modestus)构成的集团内捕食(黄颡鱼和秀丽白虾之间既存在食物资源——浮游动物的竞争,又存在捕食关系)的中宇宙实验,发现集团内捕食作用削弱了捕食者对浮游植物的牧食压力,从而影响了浮游植物的物种组成[2]。有研究报道硅藻在相对低温的环境条件下比其他浮游植物更具竞争优势;在温度恒定而营养盐水平较低时,硅藻和绿藻会成为优势种;而当营养盐浓度进一步升高后,则会引起蓝藻水华的发生[3]。因此,检测浮游植物的群落组成和多样性变化有利于更好地了解湖泊生态系统的生态变化,并为水质监测以及对蓝藻水华预警做出贡献。
新近发展的环境DNA技术被越来越多地应用于浮游植物多样性的调查,但其与传统形态学鉴定结果的一致性有待进一步研究。环境DNA(environment DNA, eDNA)指从水、沉积物、土壤、粪便、空气等环境介质中提取的各种生物的全部DNA,可获得多种生物多样性信息[4]。eDNA宏条形码技术(本文简称环境DNA技术)采用通用引物对环境DNA中特定基因片段进行聚合酶链式反应(PCR)扩增,借助高通量测序获得DNA条形码序列,通过生信分析,就能获得物种多样性及组成数据[5-6]。随着高通量测序技术的发展,环境DNA技术被越来越多地应用于浮游植物等生物多样性的研究中[7]。例如:Malviya等通过环境DNA技术探究了全球海洋中硅藻的物种多样性和群落组成[8]。Rivera等使用该技术对法国境内河流网络中的真核藻类进行了大规模的检测[9]。Boopathi等基于该技术揭示了水库中真核浮游植物季节性演替的过程[10]。环境DNA技术能提供与形态学相似的结果,一直被视为一种有望替代形态学鉴定的方法[11-13]。Rimet等在对多个湖泊硅藻的研究中,验证了环境DNA技术在硅藻检测上具有与形态学鉴定一样的识别能力[14]。Obertegger等对不同年份、不同水层真核浮游植物群落的研究结果也支持这一结论[15]。与此相反,Groendahl等在对真核附着藻类的研究中发现,环境DNA技术与形态学鉴定所检测到的浮游植物物种不尽相同[16]。Brown等在对美国大盐湖真核浮游植物的研究中也指出,与形态学鉴定相比,环境DNA技术检测得到的硅藻物种多样性偏高,而绿藻物种多样性偏低[17]。可见,目前环境DNA技术主要关注于真核浮游植物,缺少同时检测真核和原核浮游植物的研究,并且其监测结果是否与形态学鉴定一致尚无定论。因此,基于两种检测方法的浮游植物物种多样性需要更进一步的研究[18-20]。
本研究以不同牧食强度下中宇宙实验体系中的浮游植物为研究对象,采用形态学鉴定和环境DNA技术同时检测真核和原核浮游植物物种多样性,探究了两种方法检测结果的一致性和精确性,以期为在浮游植物多样性检测中更好地应用环境DNA技术提供依据和参考。
1 材料与方法 1.1 实验设计本研究中宇宙实验体系由24个500 L大型聚乙烯实验桶构建,于2020年3月24日开始在位于江苏省无锡市太湖梅梁湾的中国科学院太湖生态系统研究站开展实验。通过设置4组处理:无捕食者(对照组,C)、单独添加黄颡鱼组(鱼添加组,F)、单独添加秀丽白虾组(虾添加组,S)和同时添加黄颡鱼、秀丽白虾组(鱼虾混合组,FS),以模拟集团内对浮游植物群落施加不同程度的牧食强度,实验设置详见文献[21]。为保证环境DNA技术与形态学鉴定的结果具有可比性,两种方法均取1 L水样对浮游植物生物多样性指标进行检测。
1.2 浮游植物形态学分析样品的采集和鉴定在中宇宙实验开始运行的第84天(即6月30日),由于秀丽白虾已繁殖一代且后代已生长至成体阶段,对4个处理组进行样品采集。先分别将每个实验体系内的水混匀以避免空间异质性,然后使用采水器分别采集水面50 cm以下的水体11 L,其中1 L水样加入10 mL鲁哥试剂保存。参照《淡水浮游生物研究方法》[22]和《中国淡水藻类:系统、分类及生态》[23]进行浮游植物鉴定、计数和生物量的计算。
剩余10 L水样使用灭菌聚乙烯瓶分装并置于4 ℃保温箱内,随后带回实验室用于浮游植物环境DNA分析及理化环境因子的测定[2]。
1.3 浮游植物环境DNA分析样品的收集和生物信息分析在实验室内分别取上述剩余10 L水体样品中的1 L,在负压(< 0.02 MPa)下通过孔径为0.2 μm的聚碳酸酯膜(DuraporeTM Millipore, USA)收集浮游植物环境DNA分析样品[24],抽滤后立即将滤膜取下,置于2 mL无菌冻存管中,保存于-80 ℃的低温冰箱内直至DNA提取[2]。
采用改进的酚—氯仿提取、乙醇沉淀法从富集浮游植物DNA的聚碳酸酯膜上提取DNA[25]。本研究使用环境DNA技术同时检测了真核和原核浮游植物多样性,其中真核浮游植物的环境DNA高通量测序数据下载自Da等利用18S rRNA基因测序数据,其引物为EK-NSF573 5′-CGCGGTAATTCCAGCTCCA-3′和Ek-NSR951 5′-TTGGYRAATGCTTTCGC-3′[26];使用16S rRNA基因测序前端引物515F(5Y-CTGCCAGCMGCCGCGG-3Y)和末端引物806R(5Y-GGACTACHVGGGTWTCTAAT-3Y)对原核浮游植物多样性进行分析[27]。真核和原核浮游植物的PCR扩增反应体系和流程分布参照Mangot和Caporaso等的研究[26-27]。真核和原核浮游植物的所有样品均进行3次PCR扩增,以减少生成产物的随机误差;然后将3次PCR产物混合,使用QIAquick凝胶提取试剂盒(Qiagen, Germantown, MD, USA)进行纯化,在每个样品纯化后的PCR扩增产物中添加Phix(Illumina, SanDiego, CA, USA)后,使用Illumina MiSeq平台进行测序[28]。
对于高通量测序所得的原始数据,使用TrimGalore v0.6.10软件去除末尾的引物及adapter序列[29],再使用Fastx v0.014 (http://hannonlab.cshl.edu/fastx_toolkit/commandline.html)去除末尾质量低于Q15的碱基后用FLASH2软件对数据进行双端合并,得到有效序列[30]。然后使用USEARCH v7.1软件丢弃引物错配两个碱基以上、长度短于100 bp、总碱基错误率高于2的序列,得到优化后的序列[31]。最后使用DADA2算法计算得到ASV(amplicon sequence variant)[32],获得ASV代表序列后,分别使用Silva138.1和PR2 v5.0.0数据库对原核和真核浮游植物进行物种注释,剔除所在样品中仅出现1次及分类为叶绿体或线粒体的ASV。最终18S rRNA基因高通量测序获得1774693条平均长度为450 bp的高质量序列,将所得序列按照最少样品序列数(20066条)进行抽平,获得360个真核浮游植物ASVs;16S rRNA基因高通量测序获得11297条平均长度为250 bp的高质量序列,将所得序列按照最少样品序列数(470条)进行抽平,获得41个原核浮游植物ASVs。最后使用真核和原核浮游植物抽平后的数据进行后续数据分析。
1.4 统计与分析 1.4.1 浮游植物优势度物种优势度(Y)[33]表示原核或真核浮游植物类群中某一物种所占的优势程度,计算公式为:
| Y=NiNfi | (1) |
式中,Ni表示第i种浮游植物的个体数(序列数),N表示样品中浮游植物总个体数(总序列数),fi表示第i种浮游植物在各采样点出现的频率。在优势度的计算中,形态学鉴定和环境DNA技术计算的是属水平的优势度,本文将Y>0.02的浮游植物确定为优势属,Y>0.1的浮游植物确定为绝对优势属[34]。
1.4.2 浮游植物alpha多样性指数利用物种丰富度指数和Shannon-Wiener多样性指数表征浮游植物alpha多样性[35-37],使用R 4.2.2软件(http://www.r-project.org)中Vegan 2.5-7包计算两种方法所得的浮游植物属水平alpha多样性[38]。
1.4.3 环境DNA技术和形态学鉴定精确性评价指标由于真实环境中的生物多样性未知,因此多用精确性来反映生物多样性检测方法的可靠性,生物检测精确性的检验需要同时考虑生物重复和技术重复[39]。本研究中两种方法精确性指标基于每组处理6个生物重复进行计算。对于单个环境DNA样品,宏条形码技术直接获得的数据包括遗传分类单元(ASVs)数及其相对丰度、可注释的物种分类单元数及其相对丰度。通过这些数据,可计算物种多样性指数,例如物种丰富度、Shannon-Wiener多样性指数等。因此对两种方法的精确性评价应该包括物种分类单元以及物种多样性指数的评估[11]。其中评价两种方法物种检出精确性指标分为物种检出精确性(Dp)和物种检出率(Dc),评价两种方法所得物种多样性的精确性指标为变异系数(CV)。计算公式为:
| Dp=3×共有物种数A+B+C | (2) |
| Dc=2×只在两个平行共有的物种数+3×共有物种数A+B+C | (3) |
| CV=SDˉx | (4) |
本实验设立了4组处理,每组处理有6个生物重复。对同组6个平行样品进行检测,随机选取3个平行的检测结果,A为平行A的检测物种数,B为平行B的检测物种数,C为平行C的检测物种数;SD为基于两种方法检测的物种丰富度和Shannon-Wiener多样性指数的标准差,x为基于两种方法检测的丰富度和Shannon-Wiener多样性指数的平均值。
1.4.4 统计和分析使用软件R中的microeco v0.16.0包对两种方法获得的数据进行物种组成计算[40];分别基于浮游植物细胞个数和浮游植物生物量对形态学鉴定所得数据进行物种组成分析;使用Vegan 2.5-7包进行浮游植物alpha多样性指数计算及群落组成主坐标分析(PCoA);使用置换多元方差分析(ADONIS)和相似性分析(ANOSIM)检验不同实验处理体系中浮游植物群落组成差异。应用软件R 4.2.2中ANOVA函数对不同处理组浮游植物物种丰富度、Shannon-Wiener多样性指数和CV的差异显著性进行统计学分析;应用t.test函数对两种方法所检测的物种检出精确性和物种检出率进行配对样本t检验[41]。在软件R 4.2.2中计算平行组ASVs的物种检出精确性指标和藻类优势度[11]。
2 结果 2.1 浮游植物检测结果概况基于两对引物的环境DNA技术对浮游植物检测的结果显示,在24个样品中共检测到6门17纲43目71科111属的浮游植物,其中基于18S rRNA基因测序检测到真核浮游植物5门16纲36目64科103属,基于16S rRNA基因测序检测到原核浮游植物1门1纲7目7科8属(表 1)。
| 表 1 不同分类水平下两种方法检测的浮游植物结果概况对比 Tab. 1 Comparison of the detection results of phytoplankton at different taxonomic levels using two methods |
基于形态学鉴定对浮游植物的检测结果显示,共检测到6门6纲10目16科20属的浮游植物,其中检测到真核浮游植物5门5纲7目13科17属,原核浮游植物检测到1门1纲3目3科3属(表 1)。通过光学显微镜对24个水样中的浮游植物进行鉴定,共计数得到浮游植物8.50×108 cells/L。其中真核浮游植物为7.04×108 cells/L,原核浮游植物为1.46×108 cells/L。浮游植物生物量方面,形态学鉴定获得浮游植物总生物量270.85 mg/L, 其中真核浮游植物生物总生物量为265.79 mg/L,原核浮游植物总生物量为5.06 mg/L(附表Ⅰ)。
两种方法共同检出真核浮游植物5门5纲5目7科10属,共同检出原核浮游植物1门1纲3目2科2属(图 1)。真核浮游植物的检测中,环境DNA技术检出的物种类别与形态学鉴定检出的物种类别在不同分类水平上的重复占比如下:门重复占比100%,纲重复占比100%,目重复占比71.43%,科重复占比53.85%,属重复占比58.82%。原核浮游植物的检测中,两种方法在不同分类水平上检出的物种类别重复占比如下:门重复占比100%,纲重复占比100%,目重复占比100%,科重复占比66.67%,属重复占比66.67% (附表Ⅱ)。
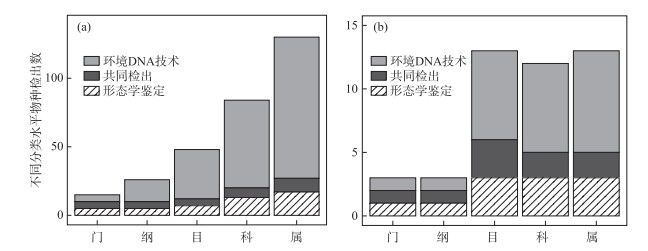
|
图 1 两种方法不同分类水平下浮游植物物种数统计:(a)真核浮游植物;(b)原核浮游植物 Fig.1 The number of phytoplankton species detected by the two methods at different taxonomy levels: (a) Eukaryotic phytoplankton; (b) Prokaryotic phytoplankton |
依据两对引物的环境DNA技术共检出浮游植物111个属,共检测到浮游植物6门17纲43目71科(表 1)。根据优势度计算结果(Y>0.02为优势属,Y>0.1为绝对优势属),对照组中优势属有3个,包括2个绿藻门(Chlorophyta)和1个隐藻门(Cryptophyta),其中绿藻门的葡萄藻属(Botryococcus)和链带藻属(Desmodesmus)为绝对优势属,优势度分别为0.72和0.65(表 2);绿藻门的葡萄藻属(Botryococcus)优势度最大(0.72),隐藻门的隐藻属(Cryptomonas)优势度最小(0.02)。鱼添加组的优势属有5个,包括绿藻门3个以及隐藻门和轮藻门(Streptophyta)各1个,其中绿藻门的栅藻属(Scenedesmus)和链带藻属(Desmodesmus)为绝对优势属,优势度分别为0.41和0.32;绿藻门的栅藻属优势度最大(0.41),绿藻门的葡萄藻属优势度最小(0.03)。鱼虾混合组中优势属有5个,均为绿藻门,其中绿藻门的链带藻属、栅藻属和多芒藻属(Golenkinia)为绝对优势属,其优势度分别为0.45、0.14和0.13,绿藻门的链带藻属优势度最大(0.45),绿藻门的Asterarcys和Hariotina的优势度最小(0.02)。虾添加组中优势属有6个,包括绿藻门5个和轮藻门1个,其中绿藻门的栅藻属、多芒藻属和链带藻属为绝对优势属,优势度分别为0.30、0.18和0.17;绿藻门的栅藻属优势度最大(0.30),绿藻门的Asterarcys优势度最小(0.04)。
| 表 2 两种方法检测得到的浮游植物优势属对比 Tab. 2 Comparison of dominant genera of phytoplankton detected by two methods |
形态学鉴定在所有样品中共检出浮游植物20个属,分属于6门6纲10目16科。对照组中的优势属有3个,包括2个绿藻门和1个隐藻门,绝对优势属是绿藻门的十字藻属(Crucigenia),其优势度最大(0.47),隐藻门的隐藻属优势度最小(0.04)。鱼添加组的优势种有5个,包括绿藻门4个和隐藻门1个,其中绿藻门的栅藻属为绝对优势属,其优势度最大(0.63),轮藻门的四棘鼓藻属(Arthrodesmus)优势度最小(0.03)。鱼虾混合组中优势属有4个,包括绿藻门3个和轮藻门1个,其中绿藻门的栅藻属和十字藻属为绝对优势属,其优势度分别为0.48和0.22,绿藻门的盘星藻属(Pediastrum)优势度最小(0.04)。虾添加组中优势属有4个,包括绿藻门2个和隐藻门、轮藻门各1个,其中绿藻门的栅藻属和轮藻门的四棘鼓藻属为绝对优势属,优势度分别为0.21和0.16,轮藻门的四角藻属(Tetraedron)优势度最小(0.02)。
在门和纲分类水平上,形态学鉴定出的浮游植物均能被依据两对引物的环境DNA技术检出,并且后者能够检出更多的物种分类(图 1、图 2)。对于原核浮游植物,在门和纲分类水平上两种方法检出一致,均检出1个蓝藻门,1个蓝藻纲。在目分类水平上,环境DNA技术和形态学分别检出7和3目,二者共同检出3目,分别为念珠藻目、聚球藻目和伪鱼腥藻目。在科水平上两种方法分别检出7和3科,二者共同检出2科,分别为微囊藻科和伪鱼腥藻科;其中环境DNA技术单独检出5科,形态学鉴定单独检出1科(附表Ⅲ)。在属分类水平上,两种方法分别检出8和3属,二者共同检出2属,分别为微囊藻属(Microcystis)和伪鱼腥藻属(Pseudanabaena)(附表Ⅲ)。
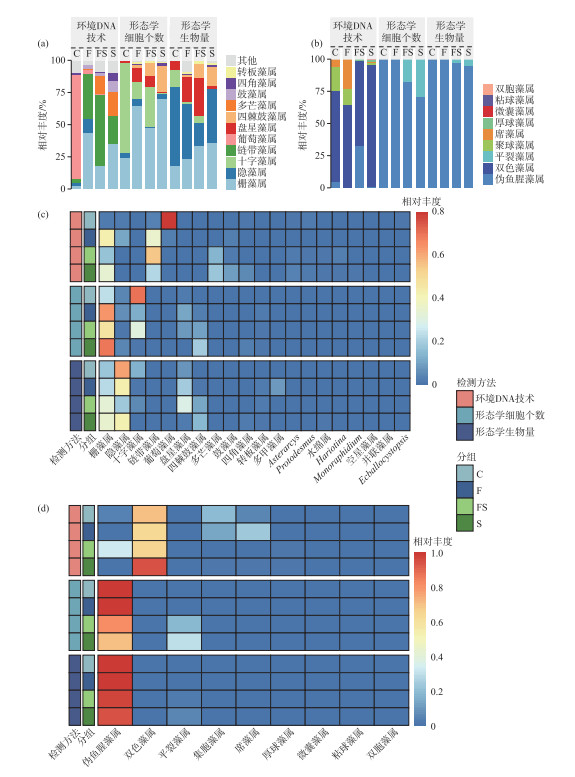
|
图 2 两种方法不同处理组中浮游植物群落组成比较:(a)真核浮游植物属水平相对丰度排名前10的群落组成;(b)原核浮游植物属水平群落组成;(c)真核浮游植物属水平相对丰度排名前20热图;(d)原核浮游植物属水平相对丰度热图(C为对照组,F为鱼添加组,FS为鱼虾混合组,S为虾添加组;下同) Fig.2 Comparison of phytoplankton community composition in different treatment groups using the two methods: (a)Top 10 community composition at genus level of eukaryotic phytoplankton; (b) Community composition at genus level of prokaryotic phytoplankton; (c) Top 20 relative abundance heatmap of eukaryotic phytoplankton; (d) Relative abundance heatmap of prokaryotic phytoplankton (C represents the control group, F represents the fish addition group, FS represents the fish and shrimp mixture group, S represents the shrimp addition group; The same as below) |
对于真核浮游植物,在门分类水平上,两种方法检出结果一致,均检出相同的5个门类,分别为绿藻门、轮藻门、褐藻门、甲藻门和隐藻门(图 2,附表Ⅳ);在纲分类水平上,环境DNA技术和形态学鉴定分别检出16纲和5纲,二者共同检出的5个纲分别为绿藻纲、隐藻纲、甲藻纲、双星藻纲和硅藻纲,其中环境DNA技术单独监测出11纲(附表Ⅳ);在目分类水平上,环境DNA技术和形态学鉴定分别检出36和7目,二者共同检出5目,分别为隐藻目、鞘藻目、多甲藻目、环藻目和双星藻目,其中环境DNA技术单独检出31目,形态学鉴定单独检出2目(附表Ⅳ);在科水平上,环境DNA技术和形态学鉴定分别检出64和13科,二者共同检出7科,分别为隐藻科、水网藻科、鞘藻科、栅藻科、月牙藻科、环藻科、双星藻科,其中环境DNA技术单独检出57科,形态学鉴定单独检出6科(附表Ⅳ);在属分类水平上,环境DNA技术和形态学鉴定分别检出103和17属,二者共同检出10属,分别为纤维藻属(Ankistrodesmus)、空星藻属(Coelastrum)、隐藻属、异极藻属(Gomphonema)、转板藻属(Mougeotia)、盘星藻属(Pediastrum)、鼓藻属(Staurastrum)、四角藻属、栅藻属和水绵属(Spirogyra),其中环境DNA技术单独检出93属,形态学鉴定单独检出7属(附表Ⅳ)。
2.3 浮游植物alpha多样性使用物种丰富度和Shannon-Wiener多样性指数评估两种方法在各实验组中获得的浮游植物alpha多样性的变化趋势。环境DNA高通量测序技术检测到的浮游植物alpha多样性指数均高于形态学鉴定,且基于18S rRNA基因的环境DNA技术获得的浮游植物alpha多样性在不同处理组间的变化趋势和形态学鉴定的结果一致(图 3),但基于16S rRNA基因测序的环境DNA技术与形态学鉴定的结果一致性较差。在真核浮游植物的检测结果中,环境DNA技术分析获得的物种丰富度均在30以上,大于形态学鉴定的结果(最大为9);原核浮游植物的检测结果中,环境DNA技术所得也普遍大于形态学鉴定。两种方法对真核浮游植物的检测结果表明,不同处理组间的浮游植物alpha多样性无显著差异(one-way ANOVA,P>0.05),但是真核浮游植物的物种丰富度在鱼虾混合组中最高,鱼添加组和虾添加组次之,对照组最低(图 3);Shannon-Wiener多样性指数的变化趋势为鱼虾混合组最高,虾添加组和鱼添加组次之,对照组最低,这与形态学鉴定检测到的真核浮游植物alpha多样性在不同处理组间的变化趋势一致。对于原核浮游植物的检测,两种方法得到了不同结果。基于16S rRNA基因测序的环境DNA技术分析结果表明,对照组、虾添加组与鱼组、鱼虾混合组之间物种丰富度具有显著差异(P < 0.05),这与基于形态学鉴定检测的物种丰富度结果不一致:对照组与虾添加组具有显著差异(P < 0.05)。基于16S rRNA基因测序的环境DNA技术表明对照组、虾组与鱼虾混合组之间的Shannon-Wiener多样性指数具有显著差异(P < 0.05),而形态学鉴定所得的Shannon-Wiener多样性指数各组之间均无显著差异(P>0.05)。
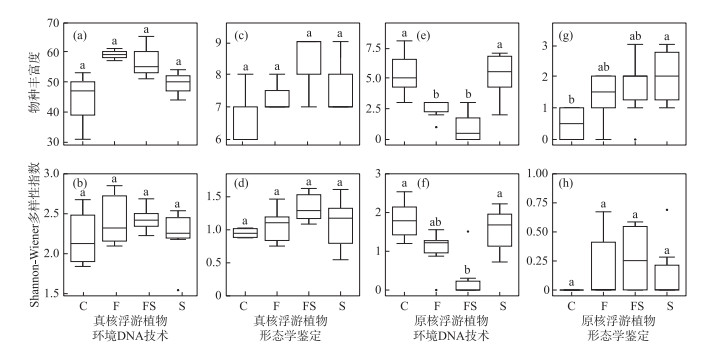
|
图 3 两种方法在不同处理组中检测得到的浮游植物alpha多样性:环境DNA技术检测得到的真核浮游植物物种丰富度(a)和Shannon-Wiener多样性指数(b);形态学检测得到的真核浮游植物物种丰富度(c)和Shannon-Wiener多样性指数(d);环境DNA技术检测得到的原核浮游植物物种丰富度(e)和Shannon-Wiener多样性指数(f);形态学检测得到的原核浮游植物物种丰富度(g)和Shannon-Wiener多样性指数(h) Fig.3 Phytoplankton alpha diversity in the different treatments detected by the two different ways: species richness (a) and Shannon-Wiener index (b) of eukaryotic phytoplankton detected by environmental DNA metabarcoding technology; species richness (c) and Shannon-Wiener index (d) of eukaryotic phytoplankton detected by morphology; species richness (e) and Shannon-Wiener index (f) of prokaryotic phytoplankton detected by environmental DNA metabarcoding technology; species richness (g) and Shannon-Wiener index (h) of prokaryotic phytoplankton detected by morphology |
PCoA用于研究不同处理组中浮游植物群落之间的群落组成。基于Bray-Curtis距离分别对环境DNA技术和形态学鉴定获得的真核和原核浮游植物群落组成进行主坐标分析。基于18S rRNA基因测序的环境DNA技术获得的真核浮游植物群落组成的PCoA中,前两个主坐标的方差贡献率分别为30.98% 和15.67%。形态学鉴定得真核浮游植物群落组成的PCoA中,前两个主坐标的方差贡献率分别为22.74% 和14.79%。基于16S rRNA基因测序的环境DNA技术获得的原核浮游植物群落组成的PCoA中,前两个主坐标的方差贡献率分别为37.19% 和19.44%。形态学鉴定获得的原核浮游植物群落组成的PCoA中,前两个主坐标的方差贡献率分别为30.49% 和25.9%。两种方法检测到的浮游植物物种分别沿PCoA1、2主坐标进行排序(图 4)。基于Bray-Curtis度量的置换多元方差分析(ADONIS)结果表明,环境DNA技术能够检测到不同处理组间浮游植物群落组成的差异(P < 0.05, 表 3),对于真核浮游植物,虾添加组和鱼虾混合组中的浮游植物群落组成相似(P>0.05, 表 3),其余各组之间群落组成均存在显著差异(P < 0.05, 表 3);对于原核浮游植物,对照组与鱼虾混合组以及鱼添加组和虾添加组群落组成相似(P>0.05, 表 3),其余各组之间群落组成均存在显著差异(P < 0.05, 表 3)。基于形态学鉴定的浮游植物群落组成PCoA分析表明,无论是真核浮游植物还是原核浮游植物,各处理组间的浮游植物群落组成均无显著性差异(P>0.05, 表 3),这与集团内捕食作用可以通过削弱食物网的营养级联强度进而影响低营养生物(浮游植物)群落组成的理论及实验结果不一致[42-44]。
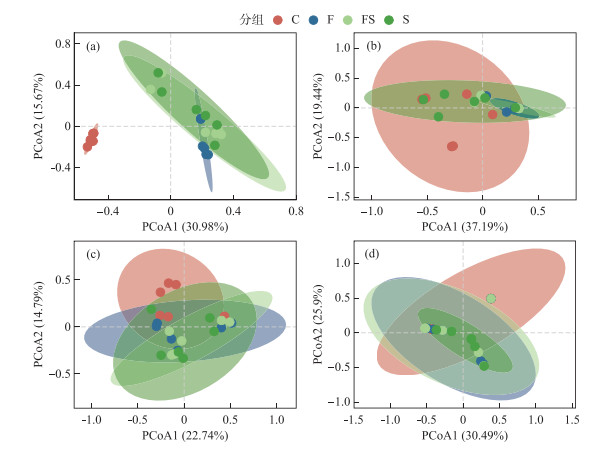
|
图 4 两种方法在不同处理组中检测得到的浮游植物群落组成PCoA排序:(a)环境DNA技术获得的真核浮游植物群落组成的PCoA分析;(b)环境DNA技术获得的原核浮游植物群落组成的PCoA分析;(c)形态学鉴定获得的真核浮游植物群落组成的PCoA分析;(d)形态学鉴定获得的原核浮游植物群落组成的PCoA分析 Fig.4 PCoA ordination and the comparison of difference in phytoplankton community composition based on Bray-Curtis distance obtained by the two methods: (a) PCoA analysis of eukaryotic phytoplankton community composition obtained by environmental DNA macrobarcoding technology; (b) PCoA analysis of prokaryotic phytoplankton community composition obtained by environmental DNA macrobarcoding technology; (c) PCoA analysis of eukaryotic phytoplankton community composition obtained by morphological identification; (d) PCoA analysis of prokaryotic phytoplankton community composition obtained by morphological identification |
| 表 3 浮游植物群落组成的ADONIS和ANOSIM检验* Tab. 3 ADONIS and ANOSIM test for phytoplankton community structure |
真核浮游植物的检测中,基于18S rRNA基因测序的环境DNA技术物种检出精确性指标均高于形态学鉴定;原核浮游植物检测中,虽然两种方法的精确性指标在各处理组间无显著差异,但是形态学鉴定的精确性指标要普遍高于基于16S rRNA基因测序的环境DNA技术。在真核浮游植物属水平上,对两种方法的物种检出精确性指标进行配对t检验的结果表明,基于18S rRNA基因测序的环境DNA技术和形态学鉴定的物种检出率在对照组中无显著差异(P>0.05,图 5),但在其余处理组中环境DNA技术的物种检出率均显著高于形态学鉴定(P < 0.01,图 5);在物种检出精确性上,环境DNA技术和形态学鉴定在对照组中无显著差异(P>0.05,图 5),其余处理组中环境DNA技术的物种检出率均显著高于形态学鉴定(P < 0.01,图 5)。在原核浮游植物属水平上,对两种方法的物种检出精确性指标进行配对t检验的结果表明,基于16S rRNA基因测序的环境DNA技术和形态学鉴定的物种检出率和物种检出精确性在各处理组之间均无显著差异(P>0.05,图 5)。
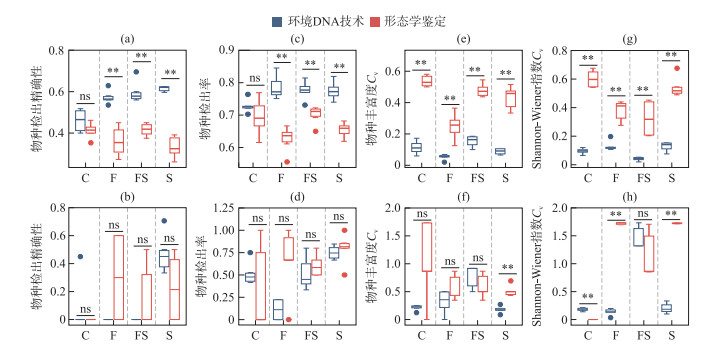
|
图 5 属水平两种检测方法的精确性指标比较:真核浮游植物(a、c、e、g)、原核浮游植物(b、d、f、h) (ns表示无显著差异,*表示显著性差异(P < 0.05),**表示极显著性差异(P < 0.01)) Fig.5 Comparison of precision indicators of the two detection methods at genus level: eukaryotic phytoplankton (a, c, e, g) and prokaryotic phytoplankton (b, d, f, h) |
变异系数用于衡量数据的波动情况和精确性。总体来看,在真核浮游植物的检测中,基于18S rRNA基因测序的环境DNA技术分析所得的alpha多样性变异系数更小,表现了更高的精确性;在原核浮游植物的检测中,基于16S rRNA基因测序的环境DNA技术与形态学鉴定所得的alpha多样性变异系数在各处理组中并不一致。在真核浮游植物中,通过配对样本t检验发现,两种方法所获得的alpha多样性指数(物种丰富度和Shannon-Wiener多样性指数)的变异系数具有显著差异(P < 0.05, 图 5);环境DNA技术所获得的alpha多样性指数变异系数均小于形态学鉴定(图 5),说明基于6次生物重复下的环境DNA技术所获得的alpha多样性指数数据波动更小,具有更高的精确性。而对于原核浮游植物,虾添加组中,基于16S rRNA基因测序的环境DNA技术所得的物种丰富度变异系数显著小于形态学鉴定(P < 0.05, 图 5),其余处理组中两种方法无显著差异;对照组中,环境DNA技术所得的Shannon-Wiener多样性指数变异系数显著大于形态学鉴定(P < 0.05, 图 5);在鱼添加组和虾添加组中,环境DNA技术所得的Shannon-Wiener多样性指数变异系数要显著小于形态学鉴定(P < 0.05, 图 5);在鱼虾混合组中,两种方法所得的Shannon-Wiener多样性指数变异系数无显著差异(P>0.05, 图 5)。
3 讨论 3.1 基于18S rRNA基因高通量测序的环境DNA技术和形态学鉴定检测出的浮游植物多样性变化趋势基本一致已有的研究表明,环境DNA技术与形态学鉴定能获得相同的真核浮游植物多样性变化趋势[45-47]。Rimet等和Kulaš等在对湖泊和河流中的硅藻多样性检测中也发现类似结论[14, 18]。Huo等的比较研究发现,环境DNA技术与形态学鉴定所得的真核浮游植物Shannon-Wiener多样性指数具有显著相关性[20]。本研究利用环境DNA技术同时对真核和原核浮游植物多样性进行检测,发现基于两对引物的环境DNA技术可以获得更多的藻类多样性信息,但只发现基于18S rRNA基因高通量测序的环境DNA技术与形态学鉴定检测到的真核浮游植物alpha多样性变化趋势一致,基于16S rRNA基因高通量测序的环境DNA技术与形态学鉴定检测到的原核浮游植物alpha多样性变化趋势具有差异(图 3)。这一方面验证了基于18S rRNA基因高通量测序环境DNA技术与形态学鉴定在浮游植物多样性检测方面具有相同的能力,另一方面也说明引物的选择对环境DNA检出样品中相关物种的重要性。Brown等使用两种方法对大盐湖浮游植物的比较研究发现,与形态学鉴定相比,环境DNA技术对硅藻物种多样性的检测结果偏高,对绿藻物种多样性的检测结果偏低,这可能是该研究中使用的高通量测序引物(p23SrV_f1和Diam23Sr1)存在PCR扩展偏差的缘故[17]。与之类似,本研究试图通过在被广泛使用的16S rRNA基因通用引物高通量获得的数据中挖掘样品中原核浮游植物的多样性信息,发现虽然与形态学鉴定相比,基于16S rRNA基因高通量测序的环境DNA技术能检测到更多的原核浮游植物种类(图 1),并且能区分不同处理组间群落组成的差异(图 4),但是较少的序列数和与形态学鉴定结果的差异说明16S rRNA基因通用引物在原核浮游植物的检测能力上有待提高。有研究表明可以借助16S rRNA基因测序中获得的原核浮游植物叶绿体基因来注释样品中存在的原核浮游植物[48],也有研究专门开发了关于16S rRNA基因序列的蓝藻注释库[49],但是这些基于16S rRNA基因注释原核浮游植物的研究仍处于发展阶段,想要提高16S rRNA基因测序对原核浮游植物的检测能力仍然需要进一步研究。
3.2 环境DNA技术比形态学鉴定能获得更高的浮游植物物种多样性及更为详细的群落组成信息越来越多的研究发现,相比较形态学鉴定,环境DNA技术拥有更高的物种鉴定分辨率[42, 50-52]。具体表现在环境DNA技术能获得更多的物种分类和更高的物种多样性指数[20, 53-54]。本研究也发现环境DNA技术能展现更高的浮游植物物种多样性和更为详细的群落组成信息。例如:在物种检出数量上,形态学鉴定在属水平上检测出20属,环境DNA技术检测出111属,远超形态学鉴定(表 1)。优势属的比较中发现,环境DNA技术能检测出9种具有优势地位的浮游植物,形态学鉴定只能检测出7种(表 2)。环境DNA技术检测所得物种丰富度均大于形态学鉴定(图 3);对两种方法获得的浮游植物物种组成进行PCoA分析发现,环境DNA技术能检测到各组之间物种组成存在差异,验证了同资源种团内捕食作用可以通过削弱食物网的营养级联强度进而影响低营养级生物(浮游植物)群落组成的结论[42-44],而本研究中的形态学鉴定并未检测到这些差异(图 3)。
环境DNA技术能够得到更高的浮游植物物种多样性和更详细的群落组成,可能主要来源于以下几点原因:首先,与形态学鉴定相比,基于高通量测序的环境DNA技术能检测到更多的物种分类,包括不易被形态学所捕获的浮游植物种类,如微型藻类、隐匿和稀有物种等[16, 19, 55-57]。本研究中,环境DNA技术检测到的浮游植物物种数是形态学检测到的5.55倍(表 1)。其中环境DNA技术和形态学鉴定分别检测到17和6纲浮游植物类群,有11纲的物种在后者中未被检测出来。其次,当相同形态的浮游植物在分子水平表现出遗传差异的时候,环境DNA技术则可能会揭示这类浮游植物隐藏的物种多样性[58-63]。例如:在纲水平上,本研究发现在形态学鉴定中无法找到环境DNA技术所鉴定的11类浮游植物,而环境DNA技术则能在两种方法共同检测到的6类浮游植物中发现更多的浮游植物分类数(形态学检测到7目,环境DNA技术检测到16目)。最后,发现环境DNA技术在更低分类水平上能检测到更多的浮游植物类群(表 1),并且各处理组间检出的物种类别差异较大,而形态学鉴定所检出物种类别在各处理组间较为接近。因此,基于环境DNA技术的PCoA分析结果能展现各组别群落组成的差异,而形态学鉴定的结果则无法展现这些差异(图 3)。
3.3 基于18S rRNA基因高通量测序的环境DNA技术检测到的浮游植物多样性的精确性高于形态学鉴定在比较环境DNA技术和形态学鉴定结果差异的研究中,缺少对两种方法所得结果精确性的研究[64-66]。Zhang等在对环境DNA技术精确性评价时使用重复样本间的OTU交叉率和物种多样性指数变异系数作为精确性评价的指标[11]。在以往的形态学鉴定中,有研究使用变异系数来衡量物种多样性指数的变异程度[67]。本研究结合了两种方法对精确性的评价指标,对环境DNA技术与形态学鉴定结果进行比较。研究结果表明,在精确性上,基于18S rRNA基因高通量测序的环境DNA技术的精确性均优于形态学鉴定(图 5);但是基于16S rRNA基因高通量测序的环境DNA技术与形态学鉴定的精确性并无显著差异。18S rRNA基因高通量测序的良好结果可能是因为环境DNA技术基于高通量测序,具有标准化流程和可重复性的技术优势[11-13, 63],从而避免了形态学因结果可重复性差以及受鉴定人员经验水平等因素的不良影响[20, 38]。反观基于16S rRNA基因高通量测序的环境DNA技术与形态学鉴定的原核浮游植物结果,较少的序列数和样品中本就稀少的原核浮游植物种类可能是导致环境DNA技术精确性不高的原因。因此,在使用环境DNA技术和形态学鉴定的结果时,应该注重结果的精确性和测序样品的序列数。有研究表明同时使用环境DNA技术和形态学鉴定对浮游植物进行检测,可以得到更加精确的物种多样性指标和群落组成[16, 18-20, 53]。但是在结合两种方法的结果之前,需要对两种方法获得的结果进行精确性比较;在确保两种方法所得结果可靠后,方可结合两种方法的结果。
3.4 目前环境DNA技术存在的不足与形态学鉴定方法相比,虽然环境DNA技术具备非侵入性、灵敏度高和不受环境条件限制等优点[7, 68-71],但也存在一系列亟待改进的问题,因此在实际应用中受到一定限制,并不能完全取代传统形态学调查方法。
首先,目前环境DNA的提取方案缺乏标准的操作流程。有研究表明,不同的DNA收集方法和提取方案均会对最终的检测结果产生影响[5, 72-73]。随着环境DNA技术在不同生物和环境中的应用,需要对来自不同生物(部位)以及不同环境的DNA进行提取,但是目前对这些不同来源的DNA提取方案并不统一。此外,环境DNA技术会受到采样环境、范围、时间以及目标物种等多重因素的限制,使得研究者在提取环境DNA的过程中操作方法不尽相同,因此想要对不同环境DNA研究结果进行比较还存在一定的挑战[74]。
其次,环境DNA技术存在假阳性和假阴性的现象。假阳性和假阴性错误是基于研究区目标物种的DNA是否被检出以及目标物种是否真实存在而产生的,主要受技术敏感性、引物偏倚性等因素的影响[75]。实际操作中,假阳性和假阴性的出现可能会导致相反的实验结果和结论。因此,应该提高引物的通用性,严格控制实验过程中的准确性,并且设置阳性和阴性对照以保证实验结果的可靠性,对实验中的外来以及交叉污染进行监控,从而减少和避免实验中的假阳性和假阴性现象[71]。
再次,环境DNA技术因为其非侵入性,无法直接观察生物本体,不能进行生物学的测量,进而无法获得诸如浮游植物形状、大小、重量等生物量指标[76]。虽然有研究表明环境DNA技术所获得的DNA含量能反映环境中部分物种的生物量,但是因为基因拷贝数的种间差异性以及PCR引入的技术偏差,环境DNA技术在检测环境中实现绝对定量仍具有挑战性[71]。除此之外,环境DNA技术对于生物体的个体状态以及生物群落所处生命阶段的测量等方面仍缺乏可行的衡量指标。
最后,环境DNA技术的本地物种参考数据库需要进一步完善和补充。基因序列和物种比对数据库是对环境DNA宏条形码进行物种鉴定的关键[15]。虽然现在存在NCBI、SILVA、PR2和Greengenes等常用数据库[77],但是在对测序数据进行物种注释的时候仍然会出现无法确定OTU分类地位的情况[60]。因此,本地物种参考数据库的建立和完善是保障利用环境DNA技术进行物种鉴定的广度和精确性的关键。除此之外,数据库的不断完善,还可以提高环境DNA技术的可用性和有效性,并增加基因序列注释到物种水平的概率[78]。
4 结论1) 在不同牧食强度下的中宇宙实验体系中,基于18S rRNA基因高通量测序的环境DNA技术与形态学鉴定检测到的浮游植物alpha多样性变化趋势一致。
2) 与形态学鉴定相比,基于16S rRNA基因和18S rRNA基因高通量测序的环境DNA技术均可以获得更多的浮游植物多样性信息,即能够检测到更多的浮游植物种类和更详细的群落组成信息。
3) 与形态学鉴定相比,基于18S rRNA基因高通量测序的环境DNA技术在浮游植物多样性检测上具有更高的精确性。
5 附录附表Ⅰ~Ⅳ见电子版(DOI: 10.18307/2024.0511)。
| 附表Ⅰ 两种方法检测结果统计 Appendix Ⅰ Two methods for result detection and statistics |
| 附表Ⅱ 两种方法共同检出物种数占比 Appendix Ⅱ Proportion of common species detected by two methods |
| 附表Ⅲ 两种方法检出原核浮游植物物种比较 Appendix Ⅲ Comparison of prokaryotic phytoplankton detected by two methods |
| 附表Ⅳ 两种方法检出真核浮游植物物种比较 Appendix Ⅳ Comparison of eukaryotic phytoplankton detected by two methods |
| [1] |
Zhang T, Chen L, Liu XX et al. Spatial pattern and influencing factors of phytoplankton in lakes of central and southern Yunnan in summer. Chinese Journal of Ecology, 2020, 39(10): 3350-3362. [张涛, 陈丽, 刘晓曦等. 云南中部和南部湖泊夏季浮游植物空间分布及其影响因子. 生态学杂志, 2020, 39(10): 3350-3362. DOI:10.13292/j.1000-4890.202010.010] |
| [2] |
Da J, Xi YL, Cheng YS et al. The effects of intraguild predation on phytoplankton assemblage composition and diversity: A mesocosm experiment. Biology, 2023, 12(4): 578. DOI:10.3390/biology12040578 |
| [3] |
Zhou Q, Chen GJ, Li R et al. A comparative study on spatio-temporal variations and environmental drivers of lake-water chlorophyll-a and diatom communities in Lake Yilong. J Lake Sci, 2023, 35(6): 1891-1904. DOI: 10.18307/2023.0614. [周起, 陈光杰, 李蕊等. 水体叶绿素a和硅藻群落时空分布及影响因子的对比分析——以云贵高原异龙湖为例. 湖泊科学, 35(6): 1891-1904. ]
|
| [4] |
Bálint M, Pfenninger M, Grossart HP et al. Environmental DNA time series in ecology. Trends in Ecology & Evolution, 2018, 33(12): 945-957. DOI:10.1016/j.tree.2018.09.003 |
| [5] |
Deiner K, Walser JC, Mächler E et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation, 2015, 183: 53-63. DOI:10.1016/j.biocon.2014.11.018 |
| [6] |
Ruppert KM, Kline RJ, Rahman MS. Past, present, and future perspectives of environmental DNA (eDNA) metabarcoding: A systematic review in methods, monitoring, and applications of global eDNA. Global Ecology and Conservation, 2019, 17: e00547. DOI:10.1016/j.gecco.2019.e00547 |
| [7] |
Yang HL, Zhang H, Du H et al. A framework for standardizing the processes of eDNA monitoring and an accessible vision of the future. J Lake Sci, 2023, 35(1): 12-31. [杨海乐, 张辉, 杜浩等. eDNA监测方法标准化框架及未来图景. 湖泊科学, 2023, 35(1): 12-31. DOI:10.18307/2023.0100] |
| [8] |
Malviya S, Scalco E, Audic S et al. Insights into global diatom distribution and diversity in the world's ocean. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(11): E1516-E1525. DOI:10.1073/pnas.1509523113 |
| [9] |
Rivera SF, Vasselon V, Bouchez A et al. Diatom metabarcoding applied to large scale monitoring networks: Optimization of bioinformatics strategies using Mothur software. Ecological Indicators, 2020, 109: 105775. DOI:10.1016/j.ecolind.2019.105775 |
| [10] |
Boopathi T, Ki JS. Unresolved diversity and monthly dynamics of eukaryotic phytoplankton in a temperate freshwater reservoir explored by pyrosequencing. Marine and Freshwater Research, 2016, 67(11): 1680. DOI:10.1071/mf15136 |
| [11] |
Zhang LJ, Xu S, Zhao Z et al. Precision of eDNA metabarcoding technology for biodiversity monitoring of eukaryotic phytoplankton in lakes. Environmental Science, 2021, 42(2): 796-807. [张丽娟, 徐杉, 赵峥等. 环境DNA宏条形码监测湖泊真核浮游植物的精准性. 环境科学, 2021, 42(2): 796-807. DOI:10.13227/j.hjkx.202007236] |
| [12] |
Zhang XW. Environmental DNA shaping a new era of ecotoxicological research. Environmental Science & Technology, 2019, 53(10): 5605-5612. DOI:10.1021/acs.est.8b06631 |
| [13] |
Debroas D, Domaizon I, Humbert JF et al. Overview of freshwater microbial eukaryotes diversity: A first analysis of publicly available metabarcoding data. FEMS Microbiology Ecology, 2017, 93(4): fix023. DOI:10.1093/femsec/fix023 |
| [14] |
Rimet F, Vasselon V, A-Keszte B et al. Do we similarly assess diversity with microscopy and high-throughput sequencing? Case of microalgae in lakes. Organisms Diversity & Evolution, 2018, 18(1): 51-62. DOI:10.1007/s13127-018-0359-5 |
| [15] |
Obertegger U, Pindo M, Flaim G. Do inferences about freshwater phytoplankton communities change when based on microscopy or high-throughput sequencing data?. Freshwater Biology, 2021, 66(4): 640-655. DOI:10.1111/fwb.13667 |
| [16] |
Groendahl S, Kahlert M, Fink P. The best of both worlds: A combined approach for analyzing microalgal diversity via metabarcoding and morphology-based methods. PLoS One, 2017, 12(2): e0172808. DOI:10.1371/journal.pone.0172808 |
| [17] |
Brown PD, Craine JM, Richards D et al. DNA metabarcoding of the phytoplankton of Great Salt Lake's Gilbert Bay: Spatiotemporal assemblage changes and comparisons to microscopy. Journal of Great Lakes Research, 2022, 48(1): 110-124. DOI:10.1016/j.jglr.2021.10.016 |
| [18] |
Kulaš A, Gligora Udovič M, Tapolczai K et al. Diatom eDNA metabarcoding and morphological methods for bioassessment of karstic river. Science of the Total Environment, 2022, 829: 154536. DOI:10.1016/j.scitotenv.2022.154536 |
| [19] |
Šimunović M, Kulaš A, Žutinić P et al. Phytoplankton diversity of a natural karst lake combining morphological and molecular approaches. Water, 2023, 15(7): 1379. DOI:10.3390/w15071379 |
| [20] |
Huo SL, Li XC, Xi BD et al. Combining morphological and metabarcoding approaches reveals the freshwater eukaryotic phytoplankton community. Environmental Sciences Europe, 2020, 32(1): 37. DOI:10.1186/s12302-020-00321-w |
| [21] |
He H, Ning XY, Chen KQ et al. Intraguild predation dampens trophic cascades in shallow aquatic mesocosms in the subtropics: Implications for lake restoration by biomanipulation. Freshwater Biology, 2021, 66(8): 1571-1580. DOI:10.1111/fwb.13739 |
| [22] |
章宗涉, 黄祥飞. 淡水浮游生物研究方法. 北京: 科学出版社, 1995: 333-344.
|
| [23] |
胡鸿钧, 魏印心. 中国淡水藻类: 系统、分类及生态. 北京: 科学出版社, 2006.
|
| [24] |
Zhou TX, Luo WL, Da J et al. Spatial distribution of bacterioplankton community composition and their diversity in Lake Fuxian during the thermal stratification period. J Lake Sci, 2022, 34(5): 1642-1655. [周天旭, 罗文磊, 笪俊等. 抚仙湖垂向分层期间水体细菌群落结构组成及多样性的空间分布. 湖泊科学, 2022, 34(5): 1642-1655. DOI:10.18307/2022.0518] |
| [25] |
Li HB, Xing P, Chen MJ et al. Short-term bacterial community composition dynamics in response to accumulation and breakdown of Microcystis blooms. Water Research, 2011, 45(4): 1702-1710. DOI:10.1016/j.watres.2010.11.011 |
| [26] |
Mangot JF, Domaizon I, Taib N et al. Short-term dynamics of small eukaryotes. Environ Microbiol, 2013, 15(6): 1745-1758. DOI:10.1111/1462-2920.12065 |
| [27] |
Caporaso JG, Lauber CL, Walters WA et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4516-4522. DOI:10.1073/pnas.1000080107 |
| [28] |
Tourlousse DM, Yoshiike S, Ohashi A et al. Synthetic spike-in standards for high-throughput 16S rRNA gene amplicon sequencing. Nucleic Acids Research, 2017, 45(4): e23. DOI:10.1093/nar/gkw984 |
| [29] |
Krueger F, James F, Ewels P et al. TrimGalore: v0.6.10 - add default decompression path (0.6.10). DOI: 10.5281/zenodo.7598955.
|
| [30] |
Magoč T, Salzberg SL. FLASH: Fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [31] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [32] |
Callahan BJ, McMurdie PJ, Rosen MJ et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods, 2016, 13: 581-583. DOI:10.1038/nmeth.3869 |
| [33] |
McNaughton SJ. Relationships among functional properties of Californian grassland. Nature, 1967, 216: 168-169. DOI:10.1038/216168b0 |
| [34] |
Tan HC, Wang YY, Cheng YL et al. Dynamic analysis of niche and interspecific association of dominant phytoplankton species in Xiashan Reservoir. J Lake Sci, 2023, 35(3): 844-853. [谭好臣, 王瑗媛, 陈阳亮等. 多水源水库浮游植物优势种生态位及种间联结性动态分析: 以山东峡山水库为例. 湖泊科学, 2023, 35(3): 844-853. DOI:10.18307/2023.0308] |
| [35] |
Ma KP. Measurement of biotic community diversity Ⅰ α diversity (Pat 1). Biodiversity Science, 1994, 2(3): 162-168. [马克平. 生物群落多样性的测度方法Ⅰ: α多样性的测度方法(上). 生物多样性, 1994, 2(3): 162-168. DOI:10.17520/biods,1994027] |
| [36] |
Ma KP, Liu MY. Measurement of biotic community diversity Ⅰ α diversity (Part 2). Biodiversity Science, 1994, 2(4): 231-239. [马克平, 刘玉明. 生物群落多样性的测度方法Ⅰ α多样性的测度方法(下). 生物多样性, 1994, 2(4): 231-239. DOI:10.17520/biods.1994038] |
| [37] |
Liu CR, Ma KP. Measurement method of biodiversity of biological community v. estimation method of species number of biological community. Acta Ecologica Sinica, 1997, 17(6): 39-48. [刘灿然, 马克平. 生物群落多样性的测度方法: 生物群落物种数目的估计方法. 生态学报, 1997, 17(6): 39-48.] |
| [38] |
Oksanen J, Blanchet FG, Frienfly M et al. Vegan community ecology package version 2.5-7 November 2020, 2020. http://cran.r-project.org, https://github.com/vegandevs/vegan.
|
| [39] |
Zhan AB, He S, Brown EA et al. Reproducibility of pyrosequencing data for biodiversity assessment in complex communities. Methods in Ecology and Evolution, 2014, 5(9): 881-890. DOI:10.1111/2041-210X.12230 |
| [40] |
Liu C, Cui YM, Li XZ et al. Microeco: An R package for data mining in microbial community ecology. FEMS Microbiology Ecology, 2021, 97(2): fiaa255. DOI:10.1093/femsec/fiaa255 |
| [41] |
Wen T, Niu GQ, Chen T et al. The best practice for microbiome analysis using R. Protein & Cell, 2023, 14(10): 713-725. DOI:10.1093/procel/pwad024 |
| [42] |
Arim M, Marquet PA. Intraguild predation: A widespread interaction related to species biology. Ecology Letters, 2004, 7(7): 557-564. DOI:10.1111/j.1461-0248.2004.00613.x |
| [43] |
Ritchie EG, Johnson CN. Predator interactions, mesopredator release and biodiversity conservation. Ecology Letters, 2009, 12(9): 982-998. DOI:10.1111/j.1461-0248.2009.01347.x |
| [44] |
Diniz AS, do Nascimento Moura A. Top-down and bottom-up effects of fish on a macrophyte-mediated trophic network: A mesocosm approach. Aquatic Ecology, 2022, 56(4): 1157-1175. DOI:10.1007/s10452-022-09976-4 |
| [45] |
Gran-Stadniczeñko S, Egge E, Hostyeva V et al. Protist diversity and seasonal dynamics in Skagerrak plankton communities as revealed by metabarcoding and microscopy. Journal of Eukaryotic Microbiology, 2019, 66(3): 494-513. DOI:10.1111/jeu.12700 |
| [46] |
Gao WL, Chen ZJ, Li YY et al. Bioassessment of a drinking water reservoir using plankton: High throughput sequencing vs. traditional morphological method. Water, 2018, 10(1): 82. DOI:10.3390/w10010082 |
| [47] |
Decelle J, Romac S, Stern RF et al. PhytoREF: A reference database of the plastidial 16S rRNA gene of photosynthetic eukaryotes with curated taxonomy. Molecular Ecology Resources, 2015, 15(6): 1435-1445. DOI:10.1111/1755-0998.12401 |
| [48] |
Lefler FW, Berthold DE, Laughinghouse HDI. Cyanoseq: A database of cyanobacterial 16S rRNA gene sequences with curated taxonomy. Journal of Phycology, 2023, 59(3): 470-480. DOI:10.1111/jpy.13335 |
| [49] |
Brantschen J, Blackman RC, Walser JC et al. Environmental DNA gives comparable results to morphology-based indices of macroinvertebrates in a large-scale ecological assessment. PLoS One, 2021, 16(9): e0257510. DOI:10.1371/journal.pone.0257510 |
| [50] |
Abad D, Albaina A, Aguirre M et al. Is metabarcoding suitable for estuarine plankton monitoring? A comparative study with microscopy. Marine Biology, 2016, 163(7): 149. DOI:10.1007/s00227-016-2920-0 |
| [51] |
Tang SQ, Wang Q, Liu L et al. Biodiversity of aquatic invertebrates based on environmental DNA metabarcoding technology: A case study of Lake Haizhu in Guangzhou. J Lake Sci, 2023, 35(4): 1443-1461. [唐诗琴, 王庆, 刘璐等. 基于环境DNA宏条形码技术的水体无脊椎动物多样性研究: 以广州海珠湖为例. 湖泊科学, 2023, 35(4): 1443-1461. DOI:10.18307/2023.0437] |
| [52] |
Banerji A, Bagley M, Elk M et al. Spatial and temporal dynamics of a freshwater eukaryotic plankton community revealed via 18S rRNA gene metabarcoding. Hydrobiologia, 2018, 818(1): 71-86. DOI:10.1007/s10750-018-3593-0 |
| [53] |
Hanžek N, Gligora Udovič M, Kajan K et al. Assessing ecological status in karstic lakes through the integration of phytoplankton functional groups, morphological approach and environmental DNA metabarcoding. Ecological Indicators, 2021, 131: 108-166. DOI:10.1016/j.ecolind.2021.108166 |
| [54] |
Elbrecht V, Vamos EE, Meissner K et al. Assessing strengths and weaknesses of DNA metabarcoding-based macroinvertebrate identification for routine stream monitoring. Methods in Ecology and Evolution, 2017, 8(10): 1265-1275. DOI:10.1111/2041-210X.12789 |
| [55] |
Zhan AB, Hulák M, Sylvester F et al. High sensitivity of 454 pyrosequencing for detection of rare species in aquatic communities. Methods in Ecology and Evolution, 2013, 4(6): 558-565. DOI:10.1111/2041-210X.12037 |
| [56] |
Comtet T, Sandionigi A, Viard F et al. DNA (meta) barcoding of biological invasions: A powerful tool to elucidate invasion processes and help managing aliens. Biological Invasions, 2015, 17(3): 905-922. DOI:10.1007/s10530-015-0854-y |
| [57] |
Vakati V, Eyun SI, Lee W. Unraveling the intricate biodiversity of the benthic harpacticoid genus Nannopus (Copepoda, Harpacticoida, Nannopodidae) in Korean waters. Molecular Phylogenetics and Evolution, 2019, 130: 366-379. DOI:10.1016/j.ympev.2018.10.004 |
| [58] |
Trobajo R, Mann DG, Clavero E et al. The use of partialcox1, rbcL and LSU rDNA sequences for phylogenetics and species identification within the Nitzschia paleaspecies complex (Bacillariophyceae). European Journal of Phycology, 2010, 45(4): 413-425. DOI:10.1080/09670262.2010.498586 |
| [59] |
Abarca N, Jahn R, Zimmermann J et al. Does the cosmopolitan diatom Gomphonema parvulum (Kützing) Kützing have a biogeography?. PLoS One, 2014, 9(1): e86885. DOI:10.1371/journal.pone.0086885 |
| [60] |
Zhang JY, Sun BL, Zhu BC et al. Research progress in the taxonomic identification of algae on the basis of molecular markers. J Lake Sci, 2021, 33(6): 1607-1625. [张军毅, 孙蓓丽, 朱冰川等. 基于分子标记的藻类鉴定研究进展. 湖泊科学, 2021, 33(6): 1607-1625. DOI:10.18307/2021.0601] |
| [61] |
Montecinos AE, Couceiro L, Peters AF et al. Species delimitation and phylogeographic analyses in the Ectocarpus subgroup siliculosi (Ectocarpales, Phaeophyceae). Journal of Phycology, 2017, 53(1): 17-31. DOI:10.1111/jpy.12452 |
| [62] |
Bruno de Sousa C, Cox CJ, Brito L et al. Improved phylogeny of brown algae Cystoseira (Fucales) from the Atlantic-Mediterranean Region based on mitochondrial sequences. PLoS One, 2019, 14(1): e0210143. DOI:10.1371/journal.pone.0210143 |
| [63] |
Zimmermann J, Glöckner G, Jahn R et al. Metabarcoding vs. morphological identification to assess diatom diversity in environmental studies. Molecular Ecology Resources, 2015, 15(3): 526-542. DOI:10.1111/1755-0998.12336 |
| [64] |
Tytgat B, Nguyen DT, Nguyen TXP et al. Monitoring of marine nematode communities through 18S rRNA metabarcoding as a sensitive alternative to morphology. Ecological Indicators, 2019, 107: 105554. DOI:10.1016/j.ecolind.2019.105554 |
| [65] |
Xiao X, Sogge H, Lagesen K et al. Use of high throughput sequencing and light microscopy show contrasting results in a study of phytoplankton occurrence in a freshwater environment. PLoS One, 2014, 9(8): e106510. DOI:10.1371/journal.pone.0106510 |
| [66] |
Yin TQ, Wang Q, Yang YF et al. Comparative study on zooplankton community structure in Pearl River Estuary based on morphological and DNA identification. Journal of Tropical Oceanography, 2022, 41(3): 172-185. [尹天齐, 王庆, 杨宇峰等. 基于形态学和DNA分子鉴定的珠江口浮游动物群落结构比较研究. 热带海洋学报, 2022, 41(3): 172-185. DOI:10.11978/2021151] |
| [67] |
Dou JT, Liu DH, Guo YJ et al. Relationship between species diversity of plant communities and heterogeneity of underground habitat. Henan Science, 2023, 41(5): 663-669. [豆靖涛, 刘东华, 郭玉娟等. 植物群落物种多样性与地境异质性的关系分析. 河南科学, 2023, 41(5): 663-669. DOI:10.3969/j.issn.1004-3918.2023.05.006] |
| [68] |
Zhao YW, Chen JQ, Dong L et al. Advances in the application of environmental DNA in aquatic ecosystems. Journal of Agro-Environment Science, 2021, 40(10): 2057-2065. [赵彦伟, 陈家琪, 董丽等. 环境DNA技术在水生态领域应用研究进展. 农业环境科学学报, 2021, 40(10): 2057-2065. DOI:10.11654/jaes.2021-0666] |
| [69] |
Wang M, Jin XW, Dong XL et al. Advances in the macrozoobenthos biodiversity monitoring and ecosystem assessment using environmental DNA metabarcoding. AES, 2021, 41(18): 7440-7453. [王萌, 金小伟, 林晓龙等. 基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展. 生态学报, 2021, 41(18): 7440-7453. DOI:10.5846/stxb202009162411] |
| [70] |
Yang QQ, Liu SW, Yu XP et al. Research progress on DNA barcoding analysis methods. Chinese Journal of Applied Ecology, 2018, 29(3): 1006-1014. [杨倩倩, 刘苏汶, 俞晓平. DNA条形码分析方法研究进展. 应用生态学报, 2018, 29(3): 1006-1014. DOI:10.13287/j.1001-9332.201803.032] |
| [71] |
Li CH, Ling LX, Tan J et al. Challenge, breakthrough and future perspectives of environmental DNA technology in monitoring aquatic organisms. Journal of Shanghai Ocean University, 2023, 32(3): 564-574. [李晨虹, 凌岚馨, 谭娟等. 环境DNA技术在水生生物监测中的挑战、突破和发展前景. 上海海洋大学学报, 2023, 32(3): 564-574. DOI:10.12024/jsou.20221104019] |
| [72] |
Li H, Kan X, Wei N et al. Establishment of a method for environmental DNA extraction from water samples. Anhui Agric Sci, 2019, 47(9): 108-110, 115. [黎慧, 阚霞, 魏宁等. 一种水环境eDNA提取方法的建立. 安徽农业科学, 2019, 47(9): 108-110, 115. DOI:10.3969/i.issn.0517-6611.2019.09.032] |
| [73] |
Takahara T, Minamoto T, Doi H. Effects of sample processing on the detection rate of environmental DNA from the Common Carp (Cyprinus carpio). Biological Conservation, 2015, 183: 64-69. DOI:10.1016/j.biocon.2014.11.014 |
| [74] |
Shan XJ, Li M, Wang WJ. Application of environmental DNA technology in aquatic ecosystem. Progress in Fishery Sciences, 2018, 39(3): 23-29. [单秀娟, 李苗, 王伟继. 环境DNA(eDNA)技术在水生生态系统中的应用研究进展. 渔业科学进展, 2018, 39(3): 23-29. DOI:10.19663/j.issn2095-9869.20171025001] |
| [75] |
Bradley IM, Pinto AJ, Guest JS. Design and evaluation of illumina MiSeq-compatible, 18S rRNA gene-specific primers for improved characterization of mixed phototrophic communities. Applied and Environmental Microbiology, 2016, 82(19): 5878-5891. DOI:10.1128/AEM.01630-16 |
| [76] |
Qin CG, Zuo T, Yu G et al. Advances in research of environmental DNA (eDNA) in biomass assessment of aquatic ecosystems. South China Fisheries Science, 2020, 16(5): 123-128. [秦传新, 左涛, 于刚等. 环境DNA在水生生态系统生物量评估中的研究进展. 南方水产科学, 2020, 16(5): 123-128. DOI:10.12131/20190256] |
| [77] |
Mao QD, Yang SW, Jin WD et al. Research status and prospects for DNA barcode technology in algal taxonomy. Journal of Hydroecology, 2020, 41(6): 9-18. [毛启迪, 杨苏文, 金位栋等. DNA条形码技术在藻类分类学中的研究现状与展望. 水生态学杂志, 2020, 41(6): 9-18. DOI:10.15928/j.1674-3075.2020.06.002] |
| [78] |
Li HX, Huang XN, Li SG et al. Environmental DNA (eDNA)-metabarcoding-based early monitoring and warning for invasive species in aquatic ecosystems. Biodiversity Science, 2019, 27(5): 491-504. [李晗溪, 黄雪娜, 李世国等. 基于环境DNA-宏条形码技术的水生生态系统入侵生物的早期监测与预警. 生物多样性, 2019, 27(5): 491-504. DOI:10.17520/biods.2018233] |
 2024, Vol. 36
2024, Vol. 36 


