(2: 南京农业大学无锡渔业学院, 无锡 214000)
(2: Wuxi Fisheries College, Nanjing Agricultural University, Wuxi 214000, P. R. China)
eDNA (environmental DNA)是指从环境样品(水体、土壤、沉积物、空气、混合物等)中提取的DNA,是各种生物的DNA混合物[1-3]。从环境样品中提取eDNA,用特定DNA宏条形码(meta-barcoding)引物对其进行扩增测序、分类学分析、相对丰度分析、功能预测等,可以监测环境中物种组成、群落结构、生态功能等相关信息[3-5]。近年来随着宏条形码技术的成熟、二代测序技术成本的下降[3],eDNA监测工作有向常态化发展的趋势[6],其中在禁捕水域针对鱼类物种组成及资源开展eDNA监测的需求最为迫切。
实现eDNA监测工作常态化的前提是实现eDNA监测的标准化[7-8]。eDNA监测技术链条中,eDNA测序结果的分析注释作为监测工作的出口控制环节,是整个监测工作后续结果判断和评估的基础[3],决定了监测结果判断和评估的精确度和准确度。参考数据库选择、阈值指标选择、目标数据准备是eDNA测序数据分析和注释中最为关键的3个技术环节,其重要性和影响已有整体性论述[3],截至目前的案例研究也都有各自的选择(参考数据库以NCBI的nt数据库为主[9-13],小部分进行自建本地参考数据库[14];OTU聚类的序列相似度以0.97、0.99为主[12, 14],也有0.95、0.98、1.00[10, 13];物种注释中的序列覆盖度取值有0.80、0.85、0.95,序列一致性取值有0.95、0.96、0.97、0.99、1.00[10, 12, 14];也有不少研究未标明相关技术环节参数[9, 11];我们随机抽取了一些案例研究的相关参数信息于附表Ⅰ中,其参数比较多样化,结果可信度、可比性存疑,亟需标准化。但若要满足具体的标准化,尚需进行具体定量比较分析。
针对这3个技术环节的标准化需求,本研究以长江中游2组eDNA监测COI基因序列数据为分析对象,针对鱼类的检出进行3组实验来分别检验:1)不同参考数据库及物种注释算法对注释结果的影响;2)OTU聚类序列相似度和物种注释分类置信度(序列一致性和序列覆盖度)对注释结果的影响;3)目标数据中各物种序列丰富度对注释结果的影响,并探讨影响机制。
1 材料方法 1.1 数据集来源2020年6月在长江中游段设置30个采样断面,相邻采样断面间的径流距离为30 km左右,乘船在江中间按照采样断面用无菌采样瓶采集1份上层水水样1.5 L。在正式采集水样并封装之前,用原水涮洗3次。水样在冰浴中暂存,在实验室中用0.2 μm孔径天津津腾TJMF微孔滤膜进行抽滤(采样后6 h内处理完毕),获得存留了eDNA的滤膜,放入50 mL无菌离心管,自封袋装好,-80 ℃冰箱保存,泡沫箱干冰浴运输。委托上海美吉生物医药科技有限公司用线粒体COI基因的扩增子(引物为mlCOIintF(5′-GGWACWGGWTGAACWGTWTAYCCYCC-3′)/jgHCO2198R(5′-TANACYTCNGGRTGNCCRAARAAYCA-3′),片段长度320 bp左右[15-16])在Illumina PE300平台以双端测序方式进行二代高通量测序,并进行双端序列拼接,获得长江中游段eDNA数据集[17]。2020年9月在长江武汉段的1个采样断面连续13天采集eDNA样品并送样测序,具体采样、测序方法同上,获得长江武汉段eDNA数据集[18]。相关序列原始数据已存于国家基因库生命大数据平台(China National GeneBank DataBase, CNGBdb, https://db.cngb.org/)的长江中游eDNA序列文件夹中(项目编号: CNP0002410,DOI: 10.26036/CNP0002410)。
1.2 本地参考数据库的构建根据“长江水生生物资源与环境本底状况调查(2017—2021年)”所整理出的长江鱼类名录[19],在NCBI数据库中搜集各物种的线粒体COI基因序列,并基于2021年在长江渔业资源与环境调查中所捕捞采集的各种鱼类的鳍条样品,通过DNA提取、用线粒体COI基因的宏条形码引物mlCOIintF/jgHCO2198R进行PCR扩增、送武汉天一辉远生物科技有限公司进行序列测定,获得相关鱼类物种的线粒体COI序列,整合构建本地针对长江鱼类的线粒体COI基因的宏条形码引物mlCOIintF/jgHCO2198R参考数据库。共收集到隶属于18目41科149属281种的2040条线粒体COI序列(附表Ⅱ),其中从NCBI搜集获得236个物种共1744条序列(截至2022年3月),自行扩增、测序获得115个物种共296条序列(【金山文档】长江鱼类COI基因条形码-持续更新https://kdocs.cn/l/cgi2zpUmWHaS)。用所收集到的所有参考序列(共281种2040条)构建本地多序列参考库,从本地多序列参考库中对每一个物种随机抽取一条参考序列(共281种281条)构建本地单序列参考库。
1.3 不同参考数据库及物种注释算法的注释结果比较分析利用美吉生物云平台(www.majorbio.com)的分析计算模块,对长江中游段eDNA监测COI数据、长江武汉段eDNA监测COI数据进行质控、拼接(用FLASH version 1.2.11 https://ccb.jhu.edu/software/FLASH/index.shtml)、OTU聚类(用USEARCH7-uparse http://drive5.com/uparse/,取99% 的序列相似度)、物种注释(用Blast(basic local alignment search tool)算法、RDP Classifier算法version 2.11 https://sourceforge.net/projects/rdp-classifier/)。以NCBI核酸序列数据库nt_v20200604、nt_v20210917库、nt_v20221012库、本地多序列参考库、本地单序列参考库为参考数据库,采用Blast算法和RDP Classifier算法取90% 的分类置信度(Blast算法中为序列一致性和序列覆盖度阈值均取90%)进行物种注释,获得注释结果,筛选出辐鳍鱼纲(Actinopteri)结果,最后进行基于不同参考数据库所获得注释结果(鱼类物种数、鱼类物种组成、鱼类OTU数)的比较分析。
1.4 不同OTU聚类序列相似度和物种注释分类置信度的注释结果比较分析基于OTU聚类序列相似度和物种注释分类置信度的取值规则及常用取值,对OTU聚类序列相似度和物种注释分类置信度(用Blast算法,即序列一致性和序列覆盖度,两者取同一个值)取值设定14个组合,unoise3 & 0.99、unoise3 & 0.9、unoise3 & 0.8、unoise3 & 0.7、0.999 & 0.99、0.999 & 0.9、0.999 & 0.8、0.999 & 0.7、0.99 & 0.97、0.99 & 0.9、0.99 & 0.8、0.99 & 0.7、0.97 & 0.8、0.9 & 0.8,对长江中游段eDNA监测COI数据、长江武汉段eDNA监测COI数据进行分析注释,参考数据库用NCBI核酸序列数据库的nt_v20221012库,获得注释结果,筛选出辐鳍鱼纲(Actinopteri)结果,进行不同阈值取值所获得注释结果(鱼类物种数、鱼类物种组成、鱼类OTU数、获得物种注释的OTU比例)的比较分析。分析计算平台、模块、步骤见1.3节。
1.5 本地参考数据库目标数据中各物种不同序列丰富度的注释结果比较分析基于本地单序列参考库、本地多序列参考库分别构建2个目标数据。考虑到在分析计算步骤中的OTU聚类环节默认去除无重复序列,所以在构建目标数据过程中对参考序列进行7倍重复。OTU聚类序列相似度和物种注释分类置信度(用Blast算法,即序列一致性和序列覆盖度,两者取同一个值)取值设定4个组合,0.999 & 0.99、0.999 & 0.9、0.99 & 0.9、0.99 & 0.8,对2个目标数据进行物种注释,参考数据库用NCBI核酸序列数据库的nt_v20221012库,获得注释结果,筛选出辐鳍鱼纲(Actinopteri)结果,进行不同目标数据所获得注释结果(鱼类物种数、鱼类物种组成、鱼类OTU数、物种正确注释比例)的比较分析。分析计算平台、模块、步骤见1.3节。
2 结果 2.1 参考数据库及物种注释算法对注释结果的影响针对长江中游段eDNA监测COI数据、长江武汉段eDNA监测COI数据这2个数据集进行的3个nt参考数据库Blast算法注释结果比较分析显示,3个nt参考数据库注释到的鱼类物种数、鱼类物种组成、鱼类OTU数差异不大(图 1,附图Ⅰa、b)。长江中游段注释到的鱼类物种数为27~29、鱼类OTU数为47~48(图 1),共涉及鱼类物种35个,其中共有物种21个,注释结果的物种组成一致性可达72% (附图Ⅰa)。长江武汉段注释到的鱼类物种数为32~34、鱼类OTU数为95(图 1),共涉及鱼类物种41个,其中共有物种25个,注释结果的物种组成一致性可达78% (附图Ⅰb)。
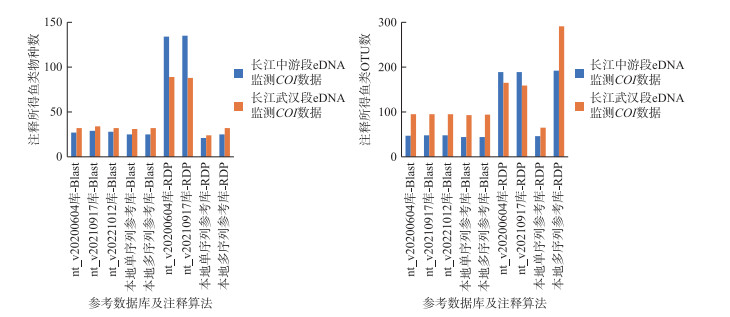
|
图 1 不同版本nt参考数据库、本地参考库及相应注释算法对注释结果的影响 Fig.1 The influence of different visions of nt reference database from NCBI and of local reference databases and of annotation algorithms on annotation results |
针对2个数据集进行的2个本地参考数据库Blast算法注释结果比较分析显示,多序列参考库和单序列参考库注释到的鱼类物种数、鱼类物种组成、鱼类OTU数基本一致(图 1,附图Ⅰa、b)。长江中游段注释到的鱼类物种数为25、鱼类OTU数为44(图 1),共涉及鱼类物种26个,其中共有物种24个,注释结果的物种组成一致性可达96% (附图Ⅰa)。长江武汉段注释到的鱼类物种数为31~32、鱼类OTU数为93~94(图 1),共涉及鱼类物种34个,其中共有物种29个,注释结果的物种组成一致性可达91% (附图Ⅰb)。
比较分析2个数据集的3个nt参考数据库和2个本地参考数据库Blast算法注释结果,nt参考数据库和本地参考数据库注释到的鱼类物种数、鱼类OTU数差异不大(图 1),但鱼类物种组成存在系统性差异(附图Ⅰa、b)。长江中游段用3个nt参考数据库和2个本地参考数据库注释到的鱼类共涉及42种,其中共有鱼类物种数为15,注释结果的物种组成一致性约为52% (附图Ⅰa)。长江中游段用3个nt参考数据库和2个本地参考数据库注释到的鱼类共涉及51种,其中共有鱼类物种数为21,注释结果的物种组成一致性约为68% (附图Ⅰb)。
2个数据集的2个nt参考数据库RDP Classifier算法注释结果,鱼类物种组成基本覆盖3个nt参考数据库Blast算法注释出的鱼类物种,比Blast算法注释出的鱼类物种有大量增加(151% ~343%,相应的OTU也有大量增加),多出的物种大都(99%)不是长江鱼类物种(图 1,附图Ⅰa、b)。长江中游段RDP Classifier算法注释到的鱼类物种数为134~135、鱼类OTU数为189(图 1),共涉及鱼类物种155个,覆盖Blast算法注释出的所有21个共有物种和35个所涉物种中的34个,覆盖率可达97% (附图Ⅰa)。长江武汉段RDP Classifier算法注释到的鱼类物种数为88~89、鱼类OTU数为159~165(图 1),共涉及鱼类物种103个,覆盖Blast算法注释出的所有25个共有物种和41个所涉物种中的39个,覆盖率可达95% (附图Ⅰb)。
2个数据集的2个本地参考数据库RDP Classifier算法注释结果,鱼类物种组成覆盖大部分2个本地参考数据库Blast算法注释出的鱼类物种,比Blast算法注释出的鱼类物种有所减少,尤其是单序列本地参考库注释出的鱼类物种数明显减少,OTU数有所增加,所增加的OTU多为只注释到科属的结果(s_unclassified_)(图 1,附图Ⅰa、b)。长江中游段RDP Classifier算法注释到的鱼类物种数为21~25、鱼类OTU数为46~192(图 1),共涉及鱼类物种28个,其中共有物种18个,覆盖Blast算法注释出的24个共有物种中的16个和26个所涉物种中的22个,RDP Classifier算法注释结果对Blast算法注释结果的覆盖率约为67% ~85% (附图Ⅰa)。长江武汉段RDP Classifier算法注释到的鱼类物种数为24~32、鱼类OTU数为65~291(图 1),共涉及鱼类物种35个,其中共有物种21个,覆盖Blast算法注释出的29个共有物种中的19个和34个所涉物种中的28个,RDP Classifier算法注释结果对Blast算法注释结果的覆盖率约为66% ~82% (附图Ⅰb)。RDP Classifier算法下,多序列参考库注释出的鱼类物种数、OTU数明显比单序列参考库注释出的多,但多出的物种和OTU主要是个别只注释到科或属水平的结果(附图Ⅰa、b)。
2.2 OTU聚类序列相似度和物种注释分类置信度对注释结果的影响对2个数据集的不同OTU聚类序列相似度和物种注释分类置信度(Blast算法,序列一致性和序列覆盖度,两者取同一个值)取值组合所获得的鱼类注释结果比较分析显示,OTU聚类序列相似度阈值越高,所获得的OTU数量越多,物种注释分类置信度阈值越高,匹配到参考序列上的OTU越少(获得物种注释的OTU比例越低),但在物种注释分类置信度0.8~0.99区间注释所得鱼类物种数和物种组成基本一致,OTU数也差异不大,而物种注释分类置信度0.7注释所得鱼类物种数、物种组成、OTU数则和0.8及以上的有明显差别(图 2,附图Ⅱa~d)。
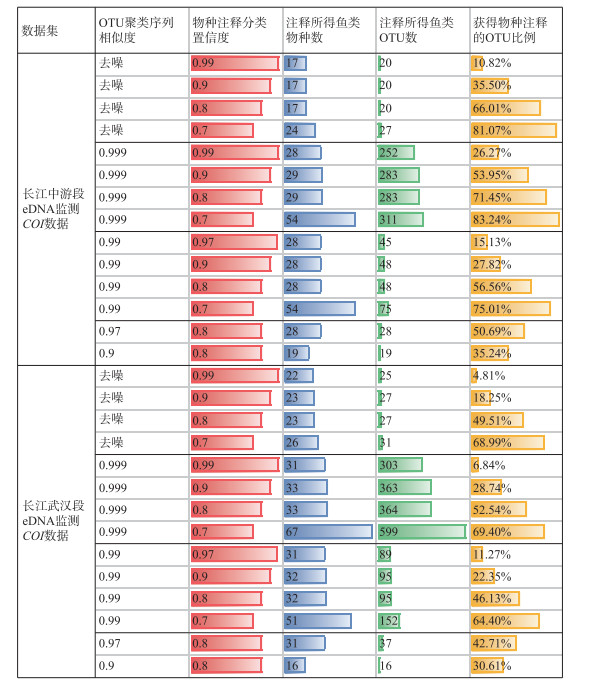
|
图 2 聚类序列相似度和物种注释分类置信度对注释结果的影响(物种注释分类置信度,在Blast算法中为序列一致性和序列覆盖度,两者取同一个值) Fig.2 The influence of different OTU cluster sequence similarity and different species annotation classification confidence on annotation results(In the Blast algorithm, the classification confidence of species annotation refers to the sequence similarity and sequence coverage, both of them take the same value) |
在OTU聚类分析中,分别用序列相似度0.999、0.99、0.97、0.9对长江中游段eDNA监测COI数据聚类,获得OTU数量为45637、14783、10698、6129,对长江武汉段eDNA监测COI数据聚类,获得OTU数量为38766、15235、7541、4120。去噪分析所获得的OTU数量比序列相似度0.9的更少,对长江中游段eDNA监测COI数据聚类获得OTU数量为4648,对长江武汉段eDNA监测COI数据聚类获得OTU数量为3573。
在物种注释分析中,分类置信度从0.7到0.8、0.9、0.99,获得物种注释的OTU占比逐步降低,在分类置信度大于0.9时,获得物种注释的OTU比例基本都在55% 以下。分类置信度在0.8~0.99区间注释所得的鱼类物种数差异不大于6%,OTU数差异不大于17% (图 2),物种组成差异不大于6% (附图Ⅱa、c)。与0.8~0.99分类置信度注释结果相比,0.7的分类置信度注释所得的鱼类物种数多47% ~116%,OTU数多23% ~98% (图 2),物种组成覆盖所有0.8~0.99分类置信度注释到的物种,并比0.8~0.99分类置信度注释到的结果多出一系列错误注释的物种(附图Ⅱa、c)。
在OTU聚类和物种注释的组合分析中,大于等于0.97的OTU聚类序列相似度和大于等于0.8的物种注释分类置信度的组合能够注释获得比较可靠的鱼类物种组成,组合间的物种组成一致性可达88% 以上(附图Ⅱ a、c)。大于等于0.99的OTU聚类序列相似度和大于等于0.8的物种注释分类置信度的组合能够注释获得可以反映鱼类种内遗传多样性的OTU数量(图 2,附图Ⅱa、c)。由2个数据集反映的在长江中游监测到具有较高遗传多样性的鱼类有黑尾近红鲌(Ancherythroculter nigrocauda)、草鱼(Ctenopharyngodon idella)、贝氏(Hemiculter bleekeri)、鲢(Hypophthalmichthys molitrix)、鳊(Parabramis pekinensis)、似鳊(Pseudobrama simoni)、寡鳞飘鱼(Pseudolaubuca engraulis)、赤眼鳟(Squaliobarbus curriculus)、银鲴(Xenocypris argentea)等(附图Ⅱa、c)。
在OTU聚类和物种注释的组合分析中,0.999的OTU聚类序列相似度和大于等于0.8的物种注释分类置信度的组合所注释获得的鱼类序列数,明显比0.9~0.99的OTU聚类序列相似度和大于等于0.8的物种注释分类置信度的组合所注释获得的鱼类序列数要少,总数量少76% ~81%,导致差距的主要原因是几个物种的序列差异,比如长江中游段数据集中的贝氏(Hemiculter bleekeri)、鲢(Hypophthalmichthys molitrix)、寡鳞飘鱼(Pseudolaubuca engraulis),长江武汉段数据集中的黑尾近红鲌(Ancherythroculter nigrocauda)、草鱼(Ctenopharyngodon idella)、银鲴(Xenocypris argentea)(附图Ⅱb、d),均属数据中遗传多样性最高的几个物种,序列数少81% ~96%,而其他大多数物种的序列数少10% ~30%。
2.3 目标数据中各物种序列丰富度对注释结果的影响基于本地单序列参考库、本地多序列参考库所构建的2个目标数据开展的OTU聚类和物种注释分析对比结果显示,在不同分析注释参数条件下均呈现出,目标数据中各物种的序列丰富度越高,注释所得的物种数、OTU数越多,物种正确注释比例越高(图 3)。同时,结果也验证了在对不同物种丰富度下的目标数据注释过程中,OTU聚类序列相似度越高,所获得的OTU越精细,OTU数量也越多,注释所得的物种也越多,物种正确注释比例越高(图 3)。

|
图 3 目标数据中各物种序列丰富度对注释结果的影响(物种注释分类置信度,在Blast算法中的序列一致性和序列覆盖度,两者取同一个值) Fig.3 The influence of different sequence richness of each species in target sequence data on annotation results(In the Blast algorithm, the classification confidence of species annotation refers to the sequence similarity and sequence coverage, both of them take the same value) |
在对2个不同物种丰富度的目标数据注释过程中,0.999的OTU聚类序列相似度、0.9的物种注释分类置信度(Blast算法,序列一致性和序列覆盖度,两者取同一个值)均获得了最高的注释所得鱼类物种数、注释所得鱼类OTU数、物种正确注释比例(图 3)。在最优分析注释参数(0.999 & 0.9)条件下,多序列数据比单序列数据注释所得鱼类物种数和OTU数分别多7% 和215%,物种正确注释比例高约4% (图 3)。
单序列数据用4组参数注释到的鱼类物种(4组结果)共涉及258种,其中一致的有237种;多序列数据用4组参数注释到的鱼类物种(4组结果)共涉及296种,其中一致的有235种;单序列数据和多序列数据用4组参数注释到的鱼类物种(8组结果)共涉及299种,其中一致的有216种(附图Ⅲa)。高OTU聚类序列相似度和物种注释分类置信度的结果并不完全对低OTU聚类序列相似度和物种注释分类置信度的结果构成覆盖,多序列数据注释所得结果也不完全覆盖单序列数据注释所得结果(附图Ⅲa)。
单序列数据用4组参数一共正确注释到鱼类220种,其中4组参数下均正确注释到的有201种;多序列数据用4组参数一共正确注释到鱼类235种,其中4组参数下均正确注释到的有206种;单序列数据和多序列数据用4组参数一共正确注释到鱼类236种,其中2组数据4组参数下均正确注释到的有189种(附图Ⅲb)。单序列数据和多序列数据用4组参数注释到的鱼类(8组结果)中有15% ~17% 的鱼类物种是错误的(附图Ⅲa、b)。单序列数据和多序列数据都未被正确注释的鱼类物种均为本研究扩增获得但nt数据库中没有参考序列的物种(附表Ⅱ,附图Ⅲb)。
3 讨论物种注释的Blast算法和RDP Classifier算法因为计算逻辑不同而获得不同的物种注释结果,但因为指向相同的目标,所以最符合目标的物种注释结果两种算法均可获得,并且RDP Classifier算法的结果可以覆盖Blast算法的结果,还会多出一些错误匹配的物种。Blast算法是基于局部相似性然后拓展延伸到全局的比对算法,并不确保能够找到全局最优解,但所找到的绝大多数解都近乎全局最优解,本研究中参考不同版本nt库注释获得的结果一致性为72% ~78% (附图Ⅰa、b)。RDP Classifier基于贝叶斯原理进行分类,通过计算后先验概率和条件概率并用Bootstrap策略计算其置信度获得最终注释,基本上能够覆盖目标结果,但同时也会获得一些非目标结果的错误注释,本研究中RDP Classifier算法nt库注释获得的结果可覆盖95% 以上的Blast算法注释获得的结果,同时也多151% ~343% 的物种,多出的大都(99%)是错误注释的物种(附图Ⅰa、b),类似结果在先前的研究中已有提及和探讨[18]。因此在基于COI基因的鱼类eDNA监测应用中,结果的注释如果选择Blast算法,对物种注释结果要保持谨慎,物种组成中可能仅有70% ~80% 的物种注释正确,其他物种可能是注释错误,也可能是注释遗漏。如果选择RDP Classifier算法,对物种注释结果更要保持谨慎,可能大部分物种(60% ~80%)都是错误注释。注释过程中如果参考数据库中缺少相关物种的参考序列,在Blast算法下,相关物种的目标序列会归为s_unclassified,在RDP Classifier算法下,相关物种的目标序列会被注释到近源物种或者相关科属下。
在Blast算法下,参考数据库对注释结果的影响主要取决于参考数据库内的物种覆盖度和种内变异覆盖度,参考库对目标物种的覆盖越高、每个物种的参考序列越丰富、对物种内序列变异覆盖越全,OTU比对脱靶的概率越低,能够比对得上的OTU数量越多,所得注释结果也越全面,但如果参考库未能有效覆盖相关物种或相关物种的核苷酸序列变异,就容易导致在比对注释过程中部分OTU的脱靶、部分物种的注释遗漏。在本研究中,不同版本nt库注释获得的物种组成和本地参考数据库注释获得的物种组成在部分物种上具有系统性差异(附图Ⅰa、b),这种系统性差异来自本地参考库和不同版本nt库内参考序列物种组成和序列变异覆盖度的系统性差异。长江记录有鱼类458种(包含外来种)[19],其中222种鱼类在nt库中缺少线粒体COI基因的扩增子(引物为mlCOIintF/jgHCO2198R)对应的参考序列,部分有参考序列的物种序列数量还比较少,对物种内的核苷酸变异覆盖有限(截至2022年3月),这导致了在实践应用中物种数正确检出的偏少,以及各物种序列相对丰度和实际群落结构的不匹配[18, 20]。为了克服nt库中参考序列的不足,可以构建本地参考数据库,快速便捷地更新补充相关物种的参考序列,尽可能全面地覆盖各物种的种内变异。在本地数据库构建方面,已有中国淡水大型底栖无脊椎动物条形码数据库在先[21]。针对长江鱼类本地数据库构建,建议根据长江鱼类名录[19],整合NCBI的nt数据库中已有的相关宏条形码参考序列和本地扩增获得的相关宏条形码参考序列,建立动态补充的本地参考数据库,以尽可能地全面覆盖本地物种及各物种内的核苷酸序列变异。本研究初步构建的长江鱼类条形码本地参考数据库以所有人可访问、可下载、可编辑的在线文档形式作为共享本地数据库供各相关研究者使用,执行CC-BY-4.0协议,各长江鱼类的参考序列有待持续补充。
在Blast算法下,OTU聚类序列相似度和物种注释分类置信度取值建议综合考虑物种注释结果的精准度和注释结果的覆盖度。OTU聚类时序列相似度设置越高,聚类形成的OTU就越精细、数目也就越多[3]。序列比对注释的分类置信度设置得偏低,往往会出现把某一物种的序列错误匹配到序列相似的另一物种上;序列比对注释的分类置信度设置得偏高,往往会出现一些通过比对无法与参考数据库中的任一序列形成匹配的OTU序列[3]。不同类群物种间的参考序列差异程度有差异,不同物种内的变异程度也有差异,所以在OTU聚类和物种注释过程中所适宜的参数设置也会有差异[22],比如针对细菌的线粒体16S rRNA基因的参数取值(比如0.97 & 0.8[23])通常低于针对真核生物的COI基因的参数取值(比如0.99 & 0.97[18])。本研究针对鱼类(COI基因上的320 bp左右大小的片段)的2个数据集分析中,在OTU聚类序列相似度大于等于0.99、物种注释分类置信度(序列一致性和序列覆盖度,两者取同一个值)大于等于0.8时,注释所得OTU数和物种数的比值大于1.5(图 2),OTU能够反映一定的种内核苷酸序列变异(附图Ⅱa、c),注释所得OTU数和物种数均相对稳定(图 2),物种注释相对一致(物种组成一致性可达88% 以上,附图Ⅱa、c)。在OTU聚类序列相似度为0.999时,注释到鱼类的序列数比序列相似度为0.99及以下时少76% ~81% (附图Ⅱb、d);在OTU聚类序列相似度一定时,注释到鱼类的OTU数随着物种注释分类置信度的升高而降低,0.97~0.99的物种注释分类置信度可获得6% ~26% 的OTU注释率,0.9的物种注释分类置信度可获得22% ~54% 的OTU注释率,0.8的物种注释分类置信度可获得46% ~71% 的OTU注释率(图 2)。因此在针对鱼类线粒体COI基因宏条形码的eDNA监测数据分析中,结果的注释如果选择Blast算法,建议OTU聚类序列相似度在0.99~0.999间取值(或者一大一小取两个值进行计算),物种注释分类置信度在0.9~0.99间取值(或者一大一小取两个值进行计算,然后对结果进行综合对比来确定)。
在提高参考数据库中各物种参考序列丰富度、选择合适的OTU聚类序列相似度和物种注释分类置信度的同时,增加目标数据的丰富度(比如在监测采样中进行有一定时空差异的重复采样),将可获得更全面的物种组成检出结果,并有望获得更准确的群落结构状况。对于鱼类种类组成的eDNA监测层面来讲,核心内容在于物种检出,无论是通过提升参考序列的丰富度覆盖尽可能多的种内变异,使eDNA所监测到的各物种特定变异序列都能够获得可匹配的参考序列,还是通过增加eDNA所监测到的各物种的种内核苷酸序列变异种类,使eDNA所监测到的各物种的种内核苷酸序列变异中总有一个或者几个能够匹配到参考序列上,都能够实现物种检出的目的[3]。本研究中,多序列数据比单序列数据注释出的物种数多5% ~7%,注释出的OTU数多78% ~215%,物种注释正确占比高4% 左右(达81.49%,图 3)。因此对于目标区域内的目标物种存在一定种内核苷酸序列变异,同时参考数据库里的参考序列对种内核苷酸序列变异的覆盖度不高的情况下,可以通过增加目标数据的丰富度(比如增加目标区域的时空差异性重复采样)以获得更全面的物种组成检出结果,并且还能获得更准确的群落结构组成状况。
基于宏条形码的eDNA监测技术路径中,受制于物种注释算法,eDNA序列物种注释可能存在并且长期存在注释错误和注释遗漏的问题。本研究对2个目标数据的分析注释结果显示,单序列数据和多序列数据中均未被正确注释的鱼类物种均为本研究扩增获得但nt数据库中没有参考序列的物种(附表Ⅱ,附图Ⅲb),说明在参考数据库中的参考序列和目标数据中的序列一致时,在物种注释过程中不存在某个物种被其他物种遮蔽的必然性。在0.999、0.99的OTU聚类序列相似度,0.99、0.9、0.8的物种注释分类置信度(Blast算法,序列一致性和序列覆盖度,两者取同一个值)水平,单个注释计算结果均有15% 以上的错误率、3% 以上的遗漏率(附表Ⅱ,附图Ⅲa、b),说明因为算法的内在局限性,单次计算存在一定的将一个物种的序列错误匹配到其他物种的参考序列上的概率。多序列数据在0.999的OTU聚类序列相似度和0.99、0.9的物种注释分类置信度下的物种正确注释比例为81% 左右,多序列数据在0.99的OTU聚类序列相似度和0.9、0.8的物种注释分类置信度下和单序列数据在0.999的OTU聚类序列相似度和0.99、0.9的物种注释分类置信度下的物种正确注释比例为77% 左右,单序列数据在0.99的OTU聚类序列相似度和0.9、0.8的物种注释分类置信度下的物种正确注释比例为72% 左右(附表Ⅱ,附图Ⅲa、b),说明在单次计算中将一个物种的序列错误匹配到其他物种的参考序列上的概率还受目标数据、计算参数影响。
4 结论针对长江中游鱼类的eDNA监测中,测序数据(线粒体COI基因的扩增子,引物为mlCOIintF/jgHCO2198R,序列片段320 bp左右)的OTU聚类,建议聚类序列相似度取值范围为0.99~0.999,对所获OTU用Blast算法进行参考nt库的物种注释,建议注释分类置信度取值范围为0.9~0.99(序列一致性和序列覆盖度,两者取同一个值),所获物种组成中可能仅有70% ~80% 是正确的。为了提高注释结果的正确率,可以提高参考数据库中的物种覆盖度和各物种种内变异覆盖度(建立完善的本地参考数据库),增加目标区域的时空差异性重复采样进而增加目标数据的丰富度。但受制于物种注释算法,单次计算存在将一个物种的序列错误匹配到其他物种参考序列上的概率,物种注释错误和注释遗漏的问题可能将长期存在。针对基于COI基因的鱼类eDNA监测中,在参考数据库、计算指标阈值、目标数据合适的情况下,物种注释错误比例力求控制在15% 以内,物种注释遗漏比例力求控制在3% 以内。随着参考数据库的完善、计算指标阈值的优化、目标数据的丰富,鱼类eDNA监测结果的物种组成有望更为准确地反映监测江段实际鱼类物种组成,各物种序列相对丰度有望更为贴近监测江段实际鱼类群落结构。
5 附录附表Ⅰ、Ⅱ和附图Ⅰ~Ⅲ见电子版(DOI: 10.18307/2024.0631)。
| 附表Ⅰ 各相关研究对参考数据库、OTU聚类相似度、物种注释分类置信度(序列一致性、序列覆盖度)的选择 Appendix Ⅰ The selection of reference databases, OTU clustering sequence similarity and species annotation classification confidence (sequence similarity and sequence coverage) |
| 附表Ⅱ 本地参考数据库的物种组成及其相应的线粒体COI序列数 Appendix Ⅱ The species composition and its COI sequence number in the local reference database |
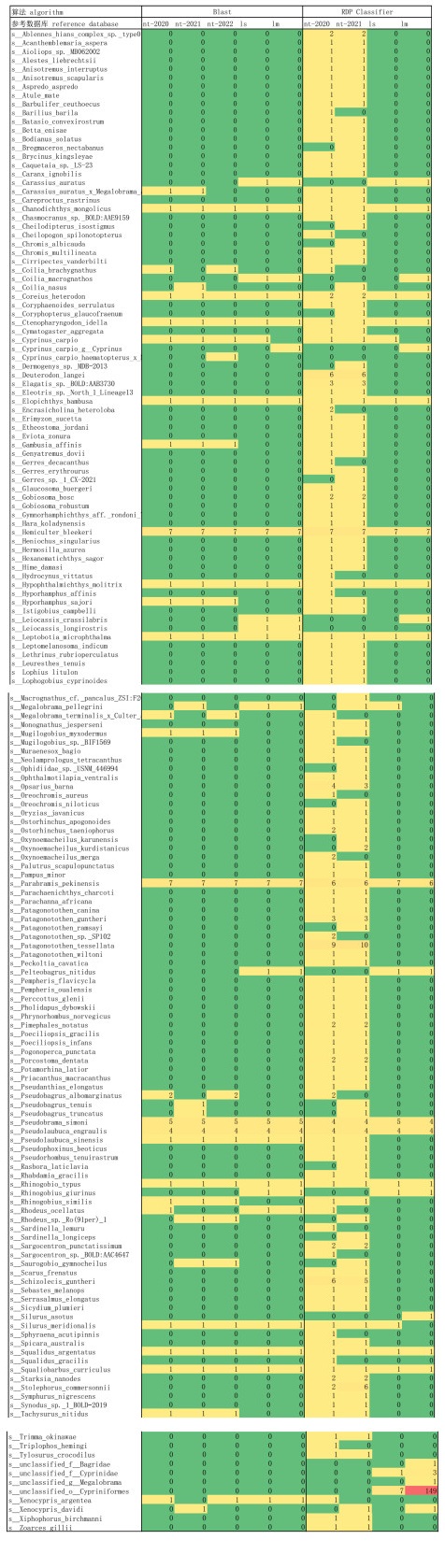
|
附图Ⅰa 长江中游段各物种OTU数(不同算法和参考数据库) (注:nt-2020、nt-2021、nt-2022分别指2020、2021、2022年基于NCBI核酸数据库建立的参考数据库;ls指本地单序列参考数据库,lm指本地多序列参考数据库。OTU聚类序列相似度取值0.99,物种注释分类置信度(序列一致性&序列覆盖度)取值0.9。) FigureAttachedIa OTU number of each species in the middle reach of Yangtze River based on different algorithms and reference databases |
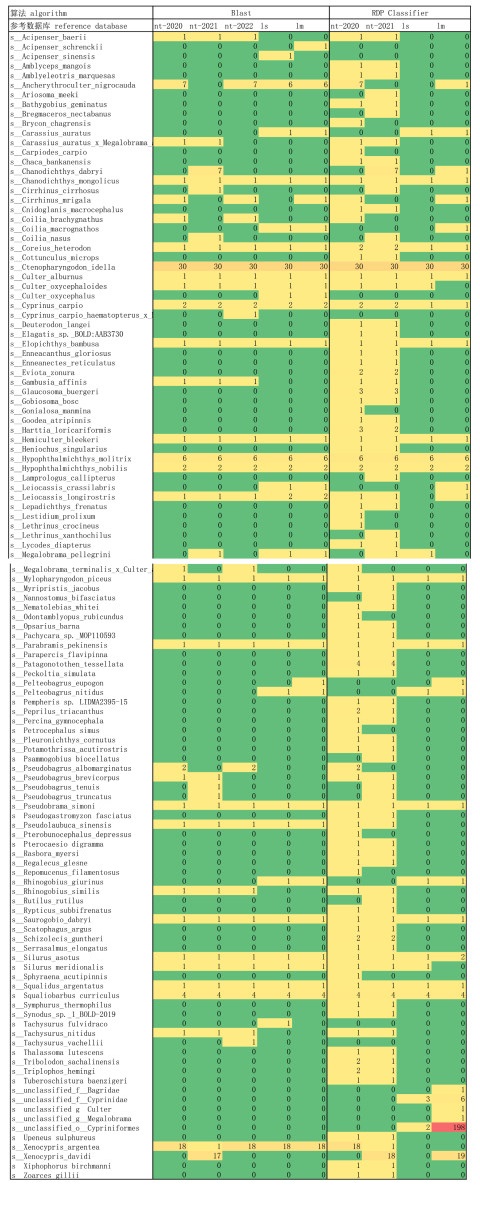
|
附图Ⅰb 长江武汉段各物种OTU数(不同算法和参考数据库)(注:nt-2020,nt-2021,nt-2022指2020、2021、2022年基于NCBI核酸数据库建立的参考数据库;ls指本地单序列参考数据库,lm指本地多序列参考数据库。OTU聚类序列相似度取值0.99,物种注释分类置信度(序列一致性&序列覆盖度)取值0.9。) FigureAttachedIb OTU number of each species in the Wuhan section of Yangtze River based on different algorithms and reference databases |
|
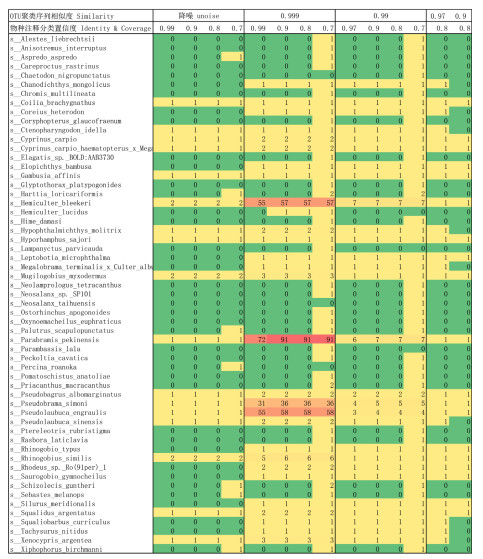
附图Ⅱa 长江中游段各物种OTU数(不同OTU聚类序列相似度和物种注释分类置信度) FigureAttachedIIa OTU number of each species in the middle reach of Yangtze River based on different OTU clustering sequence similarity and species annotation classification confidence (sequence similarity and sequence coverage) |

|
附图Ⅱb 长江中游段各物种序列数(不同OTU聚类序列相似度和物种注释分类置信度) FigureAttachedIIb Sequence number of each species in the middle reach of Yangtze River based on different OTU clustering sequence similarity and species annotation classification confidence (sequence similarity and sequence coverage) |
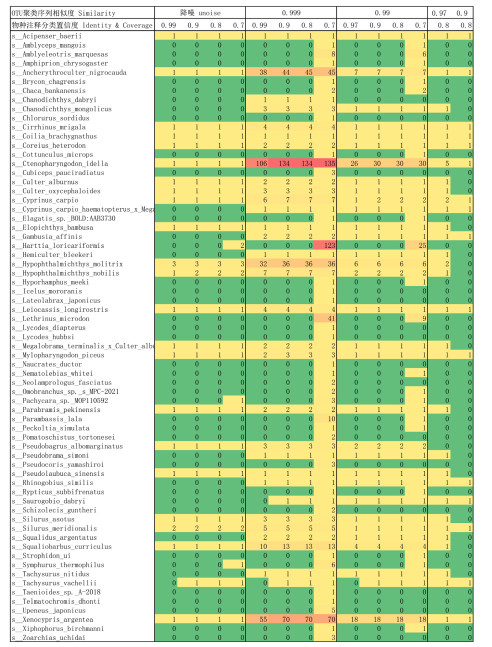
|
附图Ⅱc 长江武汉段各物种OTU数(不同OTU聚类序列相似度和物种注释分类置信度) FigureAttachedIIc OTU number of each species in the Wuhan section of Yangtze River based on different OTU clustering sequence similarity and species annotation classification confidence (sequence similarity and sequence coverage) |
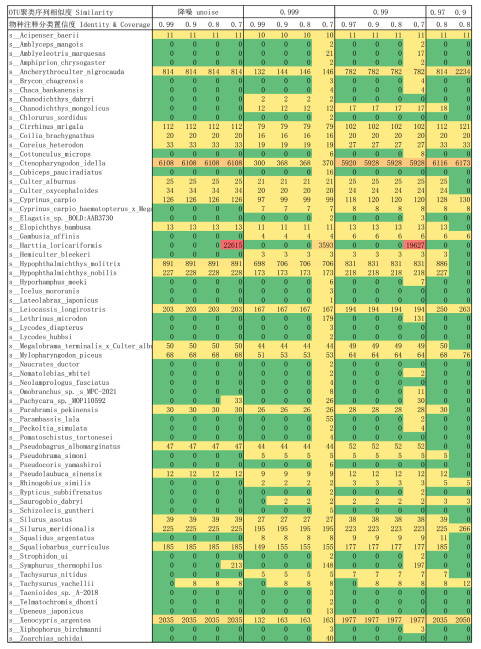
|
附图Ⅱd 长江武汉段各物种序列数(不同OTU聚类序列相似度和物种注释分类置信度) FigureAttachedIId Sequence number of each species in the Wuhan section of Yangtze River based on different OTU clustering sequence similarity and species annotation classification confidence (sequence similarity and sequence coverage) |

|
附图Ⅲa 本地参考数据集注释到的物种 FigureAttachedIIIa Species list of being annotated from local reference dataset |
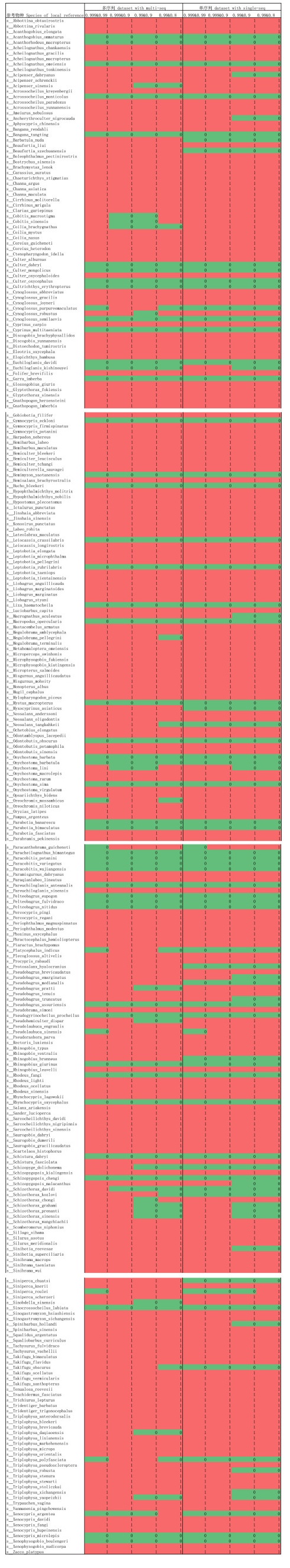
|
附图Ⅲb 本地参考数据集正确注释到的物种 FigureAttachedIIIb Species list of being correctlyannotated from local reference dataset |
附录参考文献
[1] Stackebrandt E, Goebel BM. Taxonomic note: A place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology. International Journal of Systematic and Evolutionary Microbiology, 1994, 44(4): 846-849. DOI: 10.1099/00207713-44-4-846.
[2] 薛棋文, 杨江华, 张丽娟等. eDNA宏条形码监测沉积物原生生物群落多样性. 生态毒理学报, 2022, 17(4): 175-186.
[3] Gao YC, Li HT, Wang XC et al. Research on zooplankton diversity using DNA-based metabarcoding technique: A case study in the Yalvjiang Estuary. Acta Ecologica Sinica, 2020, 40(11): 3822-3832. DOI: 10.5846/stxb201903100451. [高养春, 李海涛, 王孝程等. 利用宏DNA条形码研究浮游动物多样性——以鸭绿江口为例. 生态学报, 2020, 40(11): 3822-3832.]
[4] Feng YZ, Sun D, Shao QW et al. Applications of DNA metabarcoding in biodiversity and ecology of marine zooplankton. Acta Ecologica Sinica, 2022, 42(21): 8544-8554. DOI: 10.5846/stxb202111223288. [冯芸芝, 孙栋, 邵倩文等. DNA宏条形码技术在海洋浮游动物多样性和生态学研究中的应用. 生态学报, 2022, 42(21): 8544-8554.]
[5] Stoeckle BC, Kuehn R, Geist J. Environmental DNA as a monitoring tool for the endangered freshwater pearl mussel (Margaritifera margaritifera L.): A substitute for classical monitoring approaches? Aquatic Conservation: Marine and Freshwater Ecosystems, 2016, 26(6): 1120-1129. DOI: 10.1002/aqc.2611.
[6] Miralles L, Parrondo M, Hernández de Rojas A et al. Development and validation of eDNA markers for the detection of Crepidula fornicata in environmental samples. Marine Pollution Bulletin, 2019, 146: 827-830. DOI: 10.1016/j.marpolbul.2019.07.050.
[7] Li Wenhao, Qin Mingshuo, Hou Xianglei et al. Application of eDNA metabarcoding for detecting anura in north china. Asian Herpetological Research, 2022, 13(4): 224-232.
[8] Hänfling B, Lawson Handley L, Read DS et al. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods. Molecular Ecology, 2016, 25(13): 3101-3119. DOI: 10.1111/mec.13660.
[9] Xu N, Chang JB. Preliminary study on fish species detection in the middle and Lower Yangtze River using environmental DNA. Journal of Hydroecology, 2016, 37(5): 49-55. DOI: 10.15928/j.1674-3075.2016.05.007. [徐念, 常剑波. 长江中下游干流环境DNA样本鱼类物种检测的初步研究. 水生态学杂志, 2016, 37(5): 49-55.]
[10] Yamamoto S, Masuda R, Sato Y et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea. Scientific Reports, 2017, 7: 40368. DOI: 10.1038/srep40368.
[11] Balasingham KD, Walter RP, Mandrak NE et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries. Molecular Ecology, 2018, 27(1): 112-127. DOI: 10.1111/mec.14395.
[12] Xu N, Zhu B, Shi F et al. Monitoring seasonal distribution of an endangered anadromous sturgeon in a large river using environmental DNA. The Science of Nature, 2018, 105(11): 62. DOI: 10.1007/s00114-018-1587-4.
[13] Itakura H, Wakiya R, Yamamoto S et al. Environmental DNA analysis reveals the spatial distribution, abundance, and biomass of Japanese eels at the river-basin scale. Aquatic Conservation: Marine and Freshwater Ecosystems, 2019, 29(3): 361-373. DOI: 10.1002/aqc.3058.
[14] Lawson Handley L, Read DS, Winfield IJ et al. Temporal and spatial variation in distribution of fish environmental DNA in England’s largest lake. Environmental DNA, 2019, 1(1): 26-39. DOI: 10.1002/edn3.5.
[15] Nardi CF, Fernández DA, Vanella FA et al. The expansion of exotic Chinook salmon (Oncorhynchus tshawytscha) in the extreme south of Patagonia: An environmental DNA approach. Biological Invasions, 2019, 21(4): 1415-1425. DOI: 10.1007/s10530-018-1908-8.
[16] 舒璐, 林佳艳, 徐源等. 基于环境DNA宏条形码的洱海鱼类多样性研究. 水生生物学报, 2020, 44(5): 1080-1086.
[17] Chen JW, Chen Z, Liu SS et al. Revealing an invasion risk of fish species in Qingdao underwater world by environmental DNA metabarcoding. Journal of Ocean University of China, 2021, 20(1): 124-136. DOI: 10.1007/s11802-021-4448-2.
[18] Gao XC, Jiang W. The construction and application of BLAST database of DNA barcode for common fish in the Three Gorges Reservoir. Genomics and Applied Biology, 2021, 40(5): 1952-1960. DOI: 10.13417/j.gab.040.001952. [郜星晨, 姜伟. 三峡库区常见鱼类DNA条形码本地BLAST数据库的构建和应用. 基因组学与应用生物学, 2021, 40(5): 1952-1960.]
[19] 杨海乐, 吴金明, 张辉等. 大型河流中鱼类组成的eDNA监测效率:以长江武汉江段为例. 中国水产科学, 2021, 28(6): 796-807.
[20] Ruan HT, Wang RL, Li HT et al. Effects of sampling strategies and DNA extraction methods on eDNA metabarcoding: A case study of estuarine fish diversity monitoring. Zoological Research, 2022, 43(2): 192-204. DOI: 10.24272/j.issn.2095-8137.2021.331.
[21] Li H, Yang F, Zhang R et al. Environmental DNA metabarcoding of fish communities in a small hydropower dam reservoir: A comparison between the eDNA approach and established fishing methods. Journal of Freshwater Ecology, 2022, 37(1): 341-362. DOI: 10.1080/02705060.2022.2086181.
[22] Ma XH, Yang HW, Zhong X et al. eDNA metabarcoding analysis of the composition and spatial patterns of fish communities in the sanbanxi reservoir, China. Sustainability, 2022, 14(20): 12966. DOI: 10.3390/su142012966.
[23] Zainal Abidin DH, Mohd Nor SA, Lavoué S et al. Assessing a megadiverse but poorly known community of fishes in a tropical mangrove estuary through environmental DNA (eDNA) metabarcoding. Scientific Reports, 2022, 12: 16346. DOI: 10.1038/s41598-022-19954-3.
[24] Shen M, Guo NN, Luo ZL et al. Explore the distribution and influencing factors of fish in major rivers in Beijing with eDNA metabarcoding technology. Biodiversity Science, 2022, 30(7): 130-141. DOI: 10.17520/biods.2022240. [沈梅, 郭宁宁, 罗遵兰等. 基于eDNA metabarcoding探究北京市主要河流鱼类分布及影响因素. 生物多样性, 2022, 30(7): 130-141.]
[25] 陈治, 蔡杏伟, 张清凤等. 海南岛淡水鱼类环境DNA宏条形码参考数据库的初步构建及比较分析. 南方水产科学, 2022, 18(3): 1-12.
[26] 蒋佩文, 李敏, 张帅等. 基于线粒体COI和12S rDNA基因构建珠江河口鱼类DNA宏条形码数据库. 南方水产科学, 2022, 18(3): 13-21.
[27] Wang M, Yang X, Wang W et al. Fish diversity in Chongqing section of the national nature reserve for rare and endemic fish in the Upper Yangtze River based on edna technology. Acta Hydrobiologica Sinica, 2022, 46(1): 2-16. DOI: 10.7541/2021.2021.132. [王梦, 杨鑫, 王维等. 基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究. 水生生物学报, 2022, 46(1): 2-16.]
[28] Xu LF, Yao DD, Yang YW et al. Fish diversity analysis of artificial lakes in Karst Plateau based on eDNA metabarcoding technology. Journal of Southern Agriculture, 2022, 53(10): 2725-2735. [许龙飞, 姚邓雕, 杨原伟等. 基于eDNA宏条形码技术的喀斯特高原人工湖泊鱼类多样性分析. 南方农业学报, 2022, 53(10): 2725-2735.]
[29] Ito G, Yamauchi H, Shigeyoshi M et al. Using eDNA metabarcoding to establish targets for freshwater fish composition following river restoration. Global Ecology and Conservation, 2023, 43: e02448. DOI: 10.1016/j.gecco.2023.e02448.
[30] 吴昀晟, 唐永凯, 李建林等. 环境DNA在长江江豚监测中的应用. 中国水产科学, 2019, 26(1): 124-132.
[31] van der Heyde M, Bunce M, Wardell-Johnson G et al. Testing multiple substrates for terrestrial biodiversity monitoring using environmental DNA metabarcoding. Molecular Ecology Resources, 2020, 20(3): 10.1111/1755-10.1111/0998.13148. DOI: 10.1111/1755-0998.13148.
[32] Wang C, Tao M, Li AM et al. Research on the biodiversity of Qinhuai River based on environmental DNA metabacroding. Acta Ecologica Sinica, 2022, 42(2): 611-624. DOI: 10.5846/stxb202007221920. [王晨, 陶孟, 李爱民等. 基于环境DNA宏条形码技术的秦淮河生物多样性研究. 生态学报, 2022, 42(2): 611-624.]
| [1] |
Taberlet P, Coissac E, Hajibabaei M et al. Environmental DNA. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [2] |
Pawlowski J, Apothéloz-Perret-Gentil L, Altermatt F. Environmental DNA: What's behind the term? Clarifying the terminology and recommendations for its future use in biomonitoring. Molecular Ecology, 2020, 29(22): 4258-4264. DOI:10.1111/mec.15643 |
| [3] |
Yang HL, Zhang H, Du H. A framework for standardizing the processes of eDNA monitoring and an accessible vision of the future. J Lake Sci, 2023, 35(1): 12-31. [杨海乐, 张辉, 杜浩. eDNA监测方法标准化框架及未来图景. 湖泊科学, 2023, 35(1): 12-31. DOI:10.18307/2023.0100] |
| [4] |
Deiner K, Bik HM, Mächler E et al. Environmental DNA metabarcoding: Transforming how we survey animal and plant communities. Molecular Ecology, 2017, 26(21): 5872-5895. DOI:10.1111/mec.14350 |
| [5] |
Coble AA, Flinders CA, Homyack JA et al. eDNA as a tool for identifying freshwater species in sustainable forestry: A critical review and potential future applications. Science of the Total Environment, 2019, 649: 1157-1170. DOI:10.1016/j.scitotenv.2018.08.370 |
| [6] |
Deiner K, Yamanaka H, Bernatchez L. The future of biodiversity monitoring and conservation utilizing environmental DNA. Environmental DNA, 2021, 3(1): 3-7. DOI:10.1002/edn3.178 |
| [7] |
Dickie IA, Boyer S, Buckley HL et al. Towards robust and repeatable sampling methods in eDNA-based studies. Molecular Ecology Resources, 2018, 18(5): 940-952. DOI:10.1111/1755-0998.12907 |
| [8] |
Nicholson A, McIsaac D, MacDonald C et al. An analysis of metadata reporting in freshwater environmental DNA research calls for the development of best practice guidelines. Environmental DNA, 2020, 2(3): 343-349. DOI:10.1002/edn3.81 |
| [9] |
Itakura H, Wakiya R, Yamamoto S et al. Environmental DNA analysis reveals the spatial distribution, abundance, and biomass of Japanese eels at the river-basin scale. Aquatic Conservation: Marine and Freshwater Ecosystems, 2019, 29(3): 361-373. DOI:10.1002/aqc.3058 |
| [10] |
Hänfling B, Lawson HL, Read DS et al. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods. Molecular Ecology, 2016, 25(13): 3101-3119. DOI:10.1111/mec.13660 |
| [11] |
Nardi CF, Fernández DA, Vanella FA et al. The expansion of exotic Chinook salmon (Oncorhynchus tshawytscha) in the extreme south of Patagonia: An environmental DNA approach. Biological Invasions, 2019, 21(4): 1415-1425. DOI:10.1007/s10530-018-1908-8 |
| [12] |
Chen JW, Chen Z, Liu SS et al. Revealing an invasion risk of fish species in Qingdao underwater world by environmental DNA metabarcoding. Journal of Ocean University of China, 2021, 20(1): 124-136. DOI:10.1007/s11802-021-4448-2 |
| [13] |
Ito G, Yamauchi H, Shigeyoshi M et al. Using eDNA metabarcoding to establish targets for freshwater fish composition following river restoration. Global Ecology and Conservation, 2023, 43: e02448. DOI:10.1016/j.gecco.2023.e02448 |
| [14] |
Balasingham KD, Walter RP, Mandrak NE et al. Environmental DNA detection of rare and invasive fish species in two Great Lakes tributaries. Molecular Ecology, 2018, 27(1): 112-127. DOI:10.1111/mec.14395 |
| [15] |
Giebner H, Langen K, Bourlat SJ et al. Comparing diversity levels in environmental samples: DNA sequence capture and metabarcoding approaches using 18S and COI genes. Molecular Ecology Resources, 2020, 20(5): 1333-1345. DOI:10.1111/1755-0998.13201 |
| [16] |
Collins RA, Bakker J, Wangensteen OS et al. Non-specific amplification compromises environmental DNA metabarcoding with COI. Methods in Ecology and Evolution, 2019, 10(11): 1985-2001. DOI:10.1111/2041-210X.13276 |
| [17] |
Yang HL, Xu LX, Zhou Q et al. Identifying the method of quantifying the spatial resolution of eDNA monitoring: A case study in middle Yangtze River in mean-flow period. Resources and Environment in the Yangtze Basin, 2024, 33(4): 855-869. [杨海乐, 许兰馨, 周琼等. eDNA监测空间分辨率量化方法研究——以长江中游干流平水期为案例. 长江流域资源与环境, 2024, 33(4): 855-869.] |
| [18] |
Yang HL, Wu JM, Zhang H et al. Environmental DNA metabarcoding utilization efficiency in monitoring large river fish species composition: A case study in the Wuhan transect of the Yangtze River. Journal of Fishery Sciences of China, 2021, 28(6): 796-807. [杨海乐, 吴金明, 张辉等. 大型河流中鱼类组成的eDNA监测效率: 以长江武汉江段为例. 中国水产科学, 2021, 28(6): 796-807.] |
| [19] |
Yang HL, Shen L, He YF et al. Status of aquatic organisms resources and their environments in the Yangtze River system(2017-2021). Journal of Fisheries of China, 2023, 47(2): 3-30. [杨海乐, 沈丽, 何勇凤等. 长江水生生物资源与环境本底状况调查(2017—2021). 水产学报, 2023, 47(2): 3-30. DOI:10.11964/jfc.20220913677] |
| [20] |
Euclide PT, Lor Y, Spear MJ et al. Environmental DNA metabarcoding as a tool for biodiversity assessment and monitoring: Reconstructing established fish communities of north-temperate lakes and rivers. Diversity and Distributions, 2021, 27(10): 1966-1980. DOI:10.1111/ddi.13253 |
| [21] |
Wang M, Yuan Y, Yu HY et al. Construction of barcode library of freshwater macroinvertebrate in China. Environmental Monitoring in China, 2022, 38(1): 36-44. [王萌, 苑艺, 于海燕等. 中国淡水大型底栖无脊椎动物条形码数据库构建. 中国环境监测, 2022, 38(1): 36-44. DOI:10.19316/j.issn.1002-6002.2022.01.04] |
| [22] |
Yang HL, Du H, Qi HF et al. Effectiveness assessment of using riverine water eDNA to simultaneously monitor the riverine and riparian biodiversity information. Scientific Reports, 2021, 11: 24241. DOI:10.1038/s41598-021-03733-7 |
| [23] |
Ren Z, Wang F, Qu XD et al. Taxonomic and functional differences between microbial communities in Qinghai Lake and its input streams. Frontiers in Microbiology, 2017, 8: 2319. DOI:10.3389/fmicb.2017.02319 |
 2024, Vol. 36
2024, Vol. 36 


