(2: 南京大学环境学院, 污染控制与资源化研究国家重点实验室, 南京 210023)
(3: 陕西省环境监测中心, 西安 710054)
(2: State Key Laboratory of Pollution Control & Resource Reuse, School of the Environment Nanjing University, Nanjing 210023, P. R. China)
(3: Shaanxi Province Environmental Monitoring Center, Xian 710054, P. R. China)
由于过度捕捞和栖息地破坏,淡水生态系统的物种多样性急剧减少[1-2]。物种多样性保护依赖于有效的监测数据,包括对物种组成、丰度和种群结构的全面认知[3]。传统方法主要依靠网具等捕捞后的专家鉴定,耗时耗力,且鱼卵、仔稚鱼和近缘物种的形态学鉴定难度较大[4-5]。环境DNA(environmental DNA,eDNA)主要来源为脱落的皮肤细胞、尿液、粪便、唾液或分泌物等[3],可以在不捕捉生物体的基础上,通过定量PCR(quantitative polymerase chain reaction,qPCR)、宏条形码(eDNA metabarcoding)等技术快速获得物种组成和多样性信息。其中,定量PCR通过特异性引物对特定物种进行定量评估[6-8];环境DNA宏条形码利用通用引物同时检测多个物种,是生物监测的有力工具[9-12],具有高准确性、高灵敏度、环境友好等优势[13-15],逐渐被应用于物种多样性监测、生态健康评估、入侵生物学和保护生物学等领域[16-21]。
高质量环境DNA是精准监测的基础,了解环境DNA降解对于评估环境DNA监测结果至关重要。研究表明,从生物体释放的环境DNA通常会快速降解[22-25],并在一定时间内遵循指数衰减函数模型[26-28]。环境DNA的降解由生物和非生物因素共同驱动[29-31],生物因素包括微生物和胞外酶等[32-34],非生物因素包括紫外线辐射、pH、盐度和温度等[35-39]。在各种因素中,温度和微生物活性被认为是环境DNA降解的主要因素[23, 25, 32, 37]。已有对海洋的研究报道了环境DNA在5 d后就无法检测到[22];而在低于10 ℃的低温海洋环境下,环境DNA能够持续存在2周以上[40]。目前DNA衰变的研究多通过定量PCR检测环境DNA中目标物种DNA的降解情况[32, 40-41],但是单一物种定量PCR检测结果是否同样适用于宏条形码检测,水样延时过滤和保存温度是否会影响淡水鱼类环境DNA的降解甚至影响宏条形码检测的准确性尚不清楚。
为此,本研究针对我国常见的7种淡水鱼类,通过定量PCR和环境DNA宏条形码分别检测实验室水箱条件下鱼类环境DNA在不同温度下的降解规律,并对野外真实环境样品进一步检测分析,探究:1)温度对鱼类环境DNA降解的影响;2)是否需要选择不同的前处理时间,以确保监测结果的可靠性;3)环境DNA降解对定量PCR、宏条形码监测结果的影响。借此优化环境DNA生物监测的采样策略,提高宏条形码监测精准性和可靠性,为环境DNA生物监测提供科学依据。
1 材料与方法 1.1 实验室水箱实验在25 ℃恒温培养室内以1个300 L水族箱为载体,加入250 L自来水,曝气5 d,投放7种驯养后的鱼类,分别为乌鳢(Channa argus)、草鱼(Ctenopharyngodon idella)、鲢鱼(Hypophthalmichthys molitrix)、鳙鱼(Hypophthalmichthys nobilis)、青鱼(Mylopharyngodon piceus)、鲫鱼(Carassius auratus)和黄尾鯝(Xenocypris davidi),每种鱼类10尾。水族箱上覆盖纱网防止鱼跃行为,实验过程中持续曝气并不添加任何饵料。3 d后,将鱼类取出,用1 L采样瓶采集水样,共50瓶。在采样过程中,曝气装置持续运行,保持水体均匀流动。随机选取其中一瓶作为0 h样品,用无菌聚碳酸酯滤膜(直径47 mm、孔径0.45 μm)过滤,过滤体积为150 mL,将滤膜放入5 mL冻存管中,保存于-20 ℃。剩余样品分别置于35、25、15和4 ℃恒温箱中(图 1),随着时间推移分别取样过滤。空白对照为未投放鱼类的曝气水。
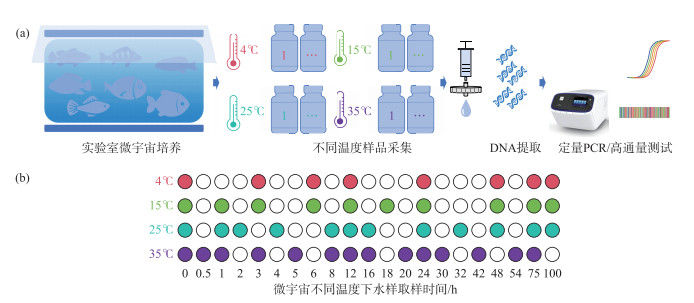
|
图 1 实验室水箱实验流程 (a:实验室水箱实验流程;b:取样时间节点。不同颜色表示不同温度,标记颜色的圆圈表示取样间隔时间) Fig.1 The flow chart of laboratory tank experiments (a: laboratory tank experiment flow chart; b: sampling time node. the diagram marked color circles represent sampling interval time, and different colors represent different temperatures) |
野外环境样品采自羊山湖(32°6′34″N,118°57′6″E),采样时间为2021年4月26日。使用采水器在水面下15 cm处采集20 L水样,并分装到1 L无菌的采样瓶中,共15瓶。采样完成后,随机选择一瓶水样现场抽滤后,作为0 h样品,过滤体积为330 mL,并设置3个平行。剩余样品置于25 ℃恒温箱中,分别于1、2、4、8、16、24、48、75、96、120、312、408 h后过滤,滤膜保存于-20 ℃。
1.3 DNA提取采用Water DNA Kit(易基诺,中国)试剂盒磁珠法提取环境DNA。对于过滤水样,将滤膜放入5 mL离心管中,加入1 mL eDNA Later、10 μL蛋白酶K和100 μL磁珠,放在振荡器上振荡20 min,并按照说明书进行后续操作。对于鱼类组织样品,采用Tissue DNA Kit(OMEGA,美国)试剂盒提取DNA。先用无菌剪刀剪取30 g背鳍肌肉组织样品,加入1 mL裂解液,在震荡仪上震荡15 min后,在70 ℃水浴充分裂解,并按说明书进行后续操作。提取的鱼类组织DNA样品用于定量PCR实验标准曲线的建立。获取的DNA溶液使用QubitTM dsDNA HS Assay Kits(Invitrogen,美国)测定浓度后,保存于-20 ℃。
1.4 定量PCR检测以提取的各种鱼类组织DNA为模板,采用Taq酶(诺唯赞,中国)进行PCR扩增,PCR反应终体积为25 μL。反应体系包括浓度为10 μmol/L的上下游引物各0.5 μL(表 1),1 μL模板DNA,19.9 μL无酶水,2.5 μL 10 × PCR高保真反应溶剂,0.5 μL dNTP mix(10 mmol/L),0.1 μL Taq DNA聚合酶。每个PCR反应设置3个重复。PCR程序为:95 ℃预变性2 min;95 ℃变性15 s,55 ℃退火20 s,72 ℃延伸30 s(35个循环);72 ℃延伸5 min,4 ℃保存。将同一样本PCR产物混合,使用VAHTS DNA Clean Beads(诺唯赞,中国)纯化,并用QubitTM dsDNA HS Assay Kits进行DNA浓度测定后,用于定量PCR实验标准曲线的建立。
| 表 1 定量PCR引物信息 Tab. 1 The primers information for qPCR |
对实验室水箱实验和野外实验中的目标物种进行定量PCR检测,所用引物及退火温度见表 1。定量PCR反应体系为20 μL,包含10 μL的2 × POWRUP SYBR MASTER MIX buffer(Applied Biosystems,美国),上下游引物各1 μL,6 μL无酶水,2 μL DNA。定量PCR反应程序为:50 ℃ 2 min,95 ℃ 2 min;95 ℃ 15 s,退火15 s,72 ℃ 1 min(40个循环[42-43]);95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s。定量PCR结束后,为了验证扩增的有效性及扩增产物的特异性,使用质量分数2%的琼脂糖凝胶电泳对扩增产物进行检测。
1.5 环境DNA半衰期计算对于环境DNA随时间降解情况的分析使用单相衰减模型(图 2),使用Graphpad Prism软件“非线性回归”下“指数方程”中的“One phase decay”计算DNA半衰期以及最快降解至无检出的时间,评估不同温度条件下鱼类环境DNA降解情况。
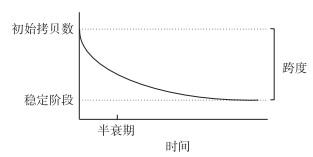
|
图 2 单相衰减模型 Fig.2 The model of one phase decay |
使用12S通用引物扩增鱼类12S rDNA基因区域(表 1)。扩增反应体系为30 μL,包括15 μL 2 × Taq Master Mix,上下游引物各1 μL,11 μL无酶水,2 μL DNA。PCR反应程序为:95 ℃预变性3 min;95 ℃变性15 s、62 ℃退火30 s、72 ℃延伸15 s(35个循环);72 ℃延伸5 min;4 ℃低温保存。
1.6.2 高通量测序将PCR产物等摩尔混合后取100 ng构建测序文库。使用VAHTS Universal DNA Library Prep Kit for Ion Torrent试剂盒(诺唯赞,中国)进行文库构建,经过样本末端补齐、添加测序接头以及文库扩增后,使用Agilent 2100 Bioanalyzer(Agilent Technologies,美国)检验质量以及浓度测定,并用Ion Torrent测序仪(Life Technologies,美国)进行测序。
1.6.3 生物信息学分析将测序得到的FASTA文件在QIIME环境下,去除重复序列,只保留unique序列;使用usearch7以注释阈值为97%对扩增子序列变体(amplicon sequence variants, ASV)进行聚类,利用uchime去除嵌合子,获得的ASV利用NCBI数据库进行BLAST比对,根据相似性注释为不同的分类等级(种:100%,属:98%,科:96%,目:93%,纲:90%)。后续的数据分析及统计绘图均在R中进行。基于鱼类的相对序列丰度将鱼类分为高丰度物种(相对丰度>1%)和低丰度物种(相对丰度<1%)。采用Ordinary one-way ANOVA检验分析物种相对丰度与温度之间的关系。
2 结果与分析高通量测序共获得高质量(质量值>20,正确率>99%)序列5573340条,其中41个水箱实验样品的平均序列为114642条,12个野外样品的平均序列为24327条。共获得378个扩增子序列变体,其中310个ASV被注释为鱼类,占总ASV的82.01%。除了鱼类,野外样品中还有少量序列注释为鸟类(4.50%)、哺乳动物(11.64%)、两栖类(0.53%)以及爬行类(1.32%)。
2.1 室内水箱实验定量PCR和宏条形码检测结果水样环境DNA随保存时间的延长而降低,温度越高,环境DNA衰减的速率越快(衰减速率排序为4 ℃<15 ℃<25 ℃<35 ℃),超过90%的环境DNA降解发生在取水后的24 h。4 ℃条件下,环境DNA浓度在0~100 h内基本维持稳定,未见明显衰减。在15、25、35 ℃温度条件下,环境DNA浓度随着样品保存时间的推移逐渐降低(图 3a)。在4 ℃下,定量PCR检出的草鱼、鲢鱼/鳙鱼的拷贝数显著高于15、25和35 ℃,且在72 h时仍有部分检出(图 3b)。乌鳢环境DNA衰减速度最快,在24 h后基本无法检出。在25 ℃下,定量PCR检出的总鱼类环境DNA的计算半衰期为2.63 h,不同温度下各种鱼类环境DNA的计算半衰期不超过6 h,最短为0.91 h(图 2,表 2)。
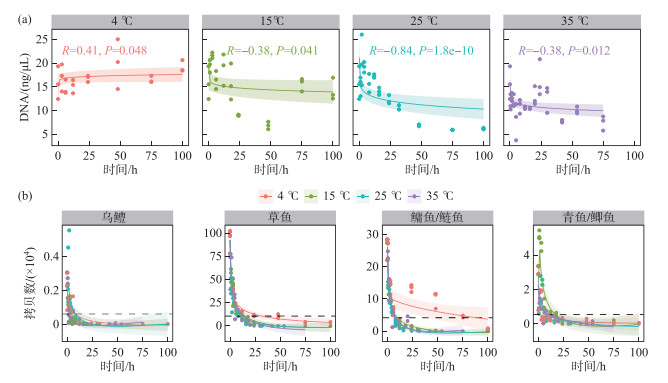
|
图 3 水箱实验中不同温度条件下鱼类环境DNA测定浓度(a)与定量PCR拷贝数(b)的变化趋势(黑色虚线代表90%环境DNA降解) Fig.3 The trends of fish eDNA concentration (a) and eDNA copies detected by qPCR (b) at different temperatures in the laboratory tank experiments (the black dashed line represents 90% eDNA degradation) |
| 表 2 水箱实验及野外实验定量PCR检测鱼类环境DNA降解分析结果 Tab. 2 Results of fish DNA degradation in the laboratory tank and field experiments detected by qPCR |
在室温环境下,实验室水箱实验和野外环境水体中的鱼类环境DNA均呈现快速下降的趋势,在24 h后几乎无法被检测到,但实验室水箱实验中的鱼类环境DNA要远高于野外环境水体。野外环境水体中的微生物环境DNA拷贝数显著低于水箱实验。与鱼类环境DNA的快速降解不同,实验室水箱实验中微生物环境DNA拷贝数整体呈现先上升后下降的趋势,约10 h时达到最大值(图 4)。
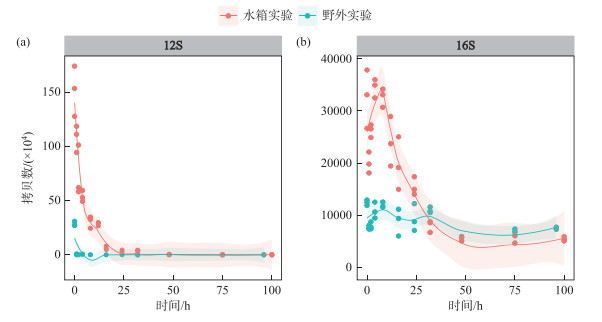
|
图 4 鱼类(a)与微生物(b)环境DNA定量PCR拷贝数随时间的变化趋势 (a:鱼类环境DNA拷贝数的变化趋势(12S);b:微生物环境DNA拷贝数的变化趋势(16S)) Fig.4 Variation trend of fish (a) and microbial (b) eDNA copies over time detected by qPCR (a: variation trend of fish eDNA (12S) copies; b: variation trend of microbial eDNA (16S) copies) |
在实验室水箱实验中,宏条形码检出的鱼类物种及序列随着水样保存时间的延长无显著变化,且不同温度之间鱼类环境DNA序列的相对丰度基本一致。不同样本的环境DNA宏条形码测序深度均达到1万以上,能有效检出乌鳢、鲫鱼、草鱼、鲢鱼、鳙鱼、青鱼和黄尾鲴7种鱼类,且相对丰度保持稳定。其中青鱼占比最多(约35%),其次是草鱼、鳙鱼、乌鳢和鲫鱼,鲢鱼和黄尾鲴占比最低(图 5)。
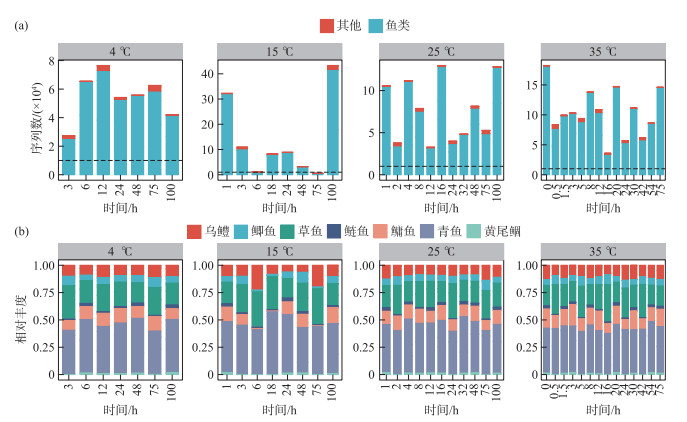
|
图 5 水箱实验中不同温度条件下环境DNA宏条形码检测结果 (a:不同温度下测序获得的序列总数;b:鱼类相对丰度。其中黑色虚线代表测序有效序列数达到10000条) Fig.5 The results of eDNA metabarcoding detection at different temperatures in the laboratory tank experiments (a: the total number of sequences was obtained by sequencing at different temperatures; b: relative abundance of fish. the black dashed line indicated that the number of valid sequences reached 10000) |
宏条形码检出的各鱼类环境DNA相对丰度随着保存时间的延长虽存在一定波动,但基本保持稳定。相关性分析显示,温度条件对鱼类环境DNA相对丰度的影响与鱼类物种有关。其中,青鱼和鳙鱼环境DNA相对丰度与温度条件的相关性P值分别为0.0243和0.0027,均小于0.05,存在显著性差异;其他鱼类环境DNA相对丰度与温度无显著相关性(表 3)。
| 表 3 水箱实验环境DNA宏条形码鱼类相对丰度与温度的相关性分析 Tab. 3 Correlation analysis of relative abundance of fish detected by eDNA metabarcoding and temperatures in the laboratory tank experiments |
宏条形码检出羊山湖的主要鱼类为鲫鱼、似鱎、鲤鱼、青鱼等(图 6a)。定量PCR检测显示,野外环境中鱼类DNA的衰减趋势与实验室水箱实验基本一致,均在24 h内快速降解。野外环境水体中目标鱼类(乌鳢、青鱼/鲫鱼)的拷贝数显著低于水箱实验,且降解速度更快。野外水体中,总鱼类环境DNA计算半衰期为0.15 h,在16 h后定量PCR检测无法有效检出(表 2)。
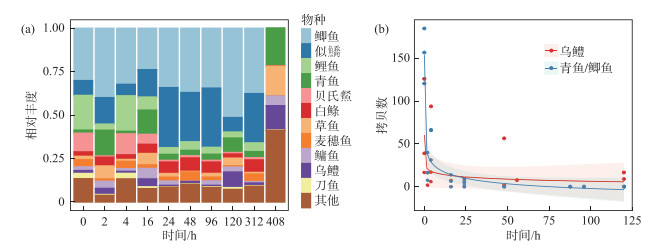
|
图 6 野外实验中不同保存时长下物种检出水平及变化趋势(a:宏条形码检测不同保存时间下鱼类相对丰度;b:定量PCR检测乌鳢、青鱼/鲫鱼拷贝数随时间的衰减趋势) Fig.6 The detection level and variation trend of species at different storage time in the field experiments (a: the fish relative abundance detected by metabarcoding at different storage time; b: the attenuation trend of Carassius auratus, Mylopharyngodon piceus / Carassius auratus copies detected by qPCR over time) |
野外实验中,随着水样保存时间的延长,宏条形码检出鱼类物种总数降低,难以被检出的鱼类主要是低丰度物种,高丰度物种的检出水平随时间的延长并未显著下降。保存时间为0~4 h时检出鱼类42种,随后物种检出水平逐步下降,408 h时仅能检出约20种鱼类。检出的鱼类中,有11种高丰度鱼类(相对丰度均大于1%),其相对丰度之和大于90%。除了第48 h外,高丰度鱼类(相对丰度>1%)的检出种类数在0~300 h基本保持稳定(均为11种),并在408 h时下降为4种(图 7)。
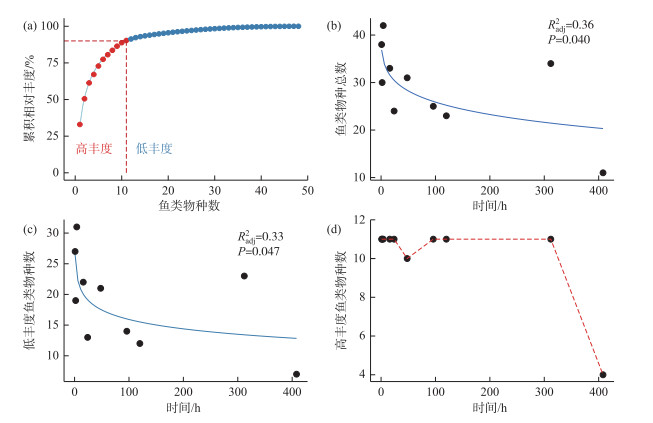
|
图 7 环境DNA宏条形码检测鱼类物种检出随时间的变化趋势 (a:鱼类累积相对丰度;b:鱼类物种总数;c:低丰度鱼类物种数;d:高丰度鱼类物种数) Fig.7 The trends of fish species detected by eDNA metabarcoding over time (a: the cumulative relative abundance of fish; b: the total number of fish species; c: low abundance fish species; d: high abundance fish species) |
环境DNA衰减对单一物种定量PCR检测结果的影响要大于对多物种宏条形码检测结果的影响。野外实验和实验室水箱实验均表明,当温度超过15 ℃时,定量PCR检测的鱼类DNA拷贝数在24 h内降解超过90%,在48 h后已经基本无法通过定量PCR进行有效检测。但是宏条形码仍能在水样放置100 h后检出实验室水箱模拟实验中投放的所有鱼类,虽物种组成存在一定波动,但各物种的相对丰度基本保持稳定。甚至在野外水体中,水样采集13 d后仍能用宏条形码技术检出大量鱼类物种信息,这与之前的大量研究结果一致[22, 40]。这说明宏条形码能更灵敏地捕捉环境中痕量环境DNA信号。尽管有研究表明在特定的条件下,定量PCR的检出限能低至几个拷贝[46]。但是,在宏条形码测序前也需要利用PCR扩增放大DNA信号,只要测序深度足够大,其检测的灵敏度就更高。因此,在定量PCR已经无信号检出时,宏条形码仍能检出大量物种信息。尽管宏条形码的监测更加稳健,但由于其采用的通用引物存在物种扩增的偏好性,且测序无法检测PCR对信号的放大过程,这导致宏条形码在物种定量上并不精准,在实际应用时要结合目的选择合适的监测方法。
低温保存能够在一定程度上延缓环境DNA的衰减,且温度对DNA衰减的延缓与物种种类有关。在4 ℃条件下,随着保存时间的延长,样本总环境DNA浓度无明显变化;但是当温度超过15 ℃时,随着时间的推移,环境DNA浓度呈现下降趋势(图 3a)。实验室水箱实验中,鱼类环境DNA拷贝数在各温度下(除4 ℃外)24 h内降解均超过90%,在48 h时基本无法检出,这说明鱼类环境DNA自离开生物体之后在环境中呈现快速降解趋势,而温度的提升加速了这一过程[38]。与35 ℃条件下相比,在4 ℃条件下乌鳢的半衰期延长了4.5倍,草鱼延长了1.5倍,鲢鱼/鳙鱼延长了近3倍(表 2)。不同鱼类物种对温度有不同的响应,分析可能原因是:(1)不同鱼类的生物学特性存在差异,进而导致鱼类环境DNA释放类型和衰减速率存在差异;(2)不同物种单位生物量释放的环境DNA总量不同,定量PCR在检测高浓度时存在遮蔽性,导致高浓度时DNA定量存在一定误差[32];(3)物种扩增引物的偏好性和效率存在差异。此外,环境DNA的形态(如组织结构、细胞结构等)也可能是影响DNA降解率和持久性的潜在因素[33]。
在野外实验与实验室水箱实验中,鱼类环境DNA降解趋势呈现一致性,但野外样品鱼类环境DNA浓度更低,衰减速度更快,定量PCR检测到的环境DNA信号的时间范围更短,在16 h基本降解至无检出。在野外实验中,定量PCR检测仅能识别出乌鳢、青鱼/鲫鱼,且目标鱼类的相对丰度较低。与实验室水箱模拟实验相比,实际野外环境中微生物多样性更复杂,生物酶更丰富,环境DNA的半衰期更短。环境理化因素和微生物活性是影响鱼类环境DNA衰减的重要因素。野外水体中氮、磷等营养盐浓度高于实验室曝气水,一定程度上促进了微生物和生物酶的分泌[37],进而影响环境DNA降解。已有文献报道,在以温度为驱动因素的核酸动力学降解中,微生物产生的胞外核酸酶活性是造成环境DNA降解的主要因素之一[23, 30, 37, 39]。整体上,温度对环境DNA衰减的影响与物种种类有关,进而导致不同温度下宏条形码检出的部分物种相对丰度存在显著差异。因此,在实际监测中,对于鱼类相对丰度的数据,除了考虑物种自身生物量的多少外,还需要考虑环境温度对环境DNA衰减的影响。
4 结论1) 环境DNA衰减对单一物种定量PCR检测结果的影响要大于对多物种宏条形码检测结果的影响。
2) 低温保存能够一定程度上延缓环境DNA的衰减,且温度对环境DNA衰减的延缓与物种种类有关。
3) 野外环境样品鱼类环境DNA浓度更低、衰减速度更快,定量PCR检测到的环境DNA信号的时间范围更短。
4) 采用定量PCR方法监测时,建议采样后4 h内完成过滤;采用宏条形码方法时,建议24 h内完成样品过滤,以最大程度降低环境DNA降解对监测结果的影响。
| [1] |
Santos R, Joyeux A, Besnard A et al. An integrative approach to assess ecological risks of surface water contamination for fish populations. Environmental Pollution, 2017, 220(Pt A): 588-596. DOI:10.1016/j.envpol.2016.10.007 |
| [2] |
Reid AJ, Carlson AK, Creed IF et al. Emerging threats and persistent conservation challenges for freshwater biodiversity. Biological Reviews of the Cambridge Philosophical Society, 2019, 94(3): 849-873. DOI:10.1111/brv.12480 |
| [3] |
Rees HC, Maddison BC, Middleditch DJ et al. REVIEW: The detection of aquatic animal species using environmental DNA-a review of eDNA as a survey tool in ecology. Journal of Applied Ecology, 2014, 51(5): 1450-1459. DOI:10.1111/1365-2664.12306 |
| [4] |
Piggott MP, Banks SC, Broadhurst BT et al. Comparison of traditional and environmental DNA survey methods for detecting rare and abundant freshwater fish. Aquatic Conservation: Marine and Freshwater Ecosystems, 2021, 31(1): 173-184. DOI:10.1002/aqc.3474 |
| [5] |
Lyal CHC, Weitzman AL. Taxonomy: Exploring the impediment. Science, 2004, 305(5687): 1106. DOI:10.1126/science.305.5687.1106a |
| [6] |
Yusishen ME, Eichorn FC, Anderson WG et al. Development of quantitative PCR assays for the detection and quantification of lake sturgeon (Acipenser fulvescens) environmental DNA. Conservation Genetics Resources, 2020, 12(1): 17-19. DOI:10.1007/s12686-018-1054-8 |
| [7] |
Takahashi S, Sakata MK, Minamoto T et al. Comparing the efficiency of open and enclosed filtration systems in environmental DNA quantification for fish and jellyfish. PLoS One, 2020, 15(4): e0231718. DOI:10.1371/journal.pone.0231718 |
| [8] |
Hoy MS, Ostberg CO. Development of a quantitative PCR assay for detection of redside shiner (Richardsonius balteatus) from environmental DNA. BMC Research Notes, 2019, 12(1): 782. DOI:10.1186/s13104-019-4819-6 |
| [9] |
Shu L, Ludwig A, Peng ZG. Environmental DNA metabarcoding primers for freshwater fish detection and quantification: In silico and in tanks. Ecology and Evolution, 2021, 11(12): 8281-8294. DOI:10.1002/ece3.7658 |
| [10] |
Tsuji S, Shibata N, Inui R et al. Environmental DNA phylogeography: Successful reconstruction of phylogeographic patterns of multiple fish species from cups of water. Molecular Ecology Resources, 2023, 23(5): 1050-1065. DOI:10.1111/1755-0998.13772 |
| [11] |
Zhang JW, Yang JH, Zhang XW. Study on methods of zooplankton sampling for DNA metabarcoding monitoring. Environmental Monitoring and Forewarning, 2022, 14(1): 35-40. [张靖雯, 杨江华, 张效伟. 浮游动物DNA宏条形码多样性监测采样方法研究. 环境监控与预警, 2022, 14(1): 35-40.] |
| [12] |
Cai K, Yang YN, Li XW et al. Preliminary study on application of environmental DNA technology on fish community monitoring in Jiangsu Province. Environmental Monitoring and Forewarning, 2022, 14(2): 43-48. [蔡琨, 杨雅楠, 李旭文等. 基于环境DNA技术的江苏省鱼类群落监测应用初探. 环境监控与预警, 2022, 14(2): 43-48. DOI:10.3969/j.issn.1674-6732.2022.02.007] |
| [13] |
Bylemans J, Gleeson DM, Duncan RP et al. A performance evaluation of targeted eDNA and eDNA metabarcoding analyses for freshwater fishes. Environmental DNA, 2019, 1(4): 402-414. DOI:10.1002/edn3.41 |
| [14] |
Harper LR, Lawson Handley L, Hahn C et al. Needle in a haystack? A comparison of eDNA metabarcoding and targeted qPCR for detection of the great crested newt (Triturus cristatus). Ecology and Evolution, 2018, 8(12): 6330-6341. DOI:10.1002/ece3.4013 |
| [15] |
Zhang LJ, Yang JH, Zhang Y et al. eDNA biomonitoring revealed the ecological effects of water diversion projects between Yangtze River and Tai Lake. Water Research, 2022, 210: 117994. DOI:10.1016/j.watres.2021.117994 |
| [16] |
Wu YS, Tang YK, Li JL et al. The application of environmental DNA in the monitoring of the Yangtze finless porpoise, Neophocaena phocaenoides asaeorientalis. Journal of Fishery Sciences of China, 2019, 26(1): 124-132. [吴昀晟, 唐永凯, 李建林等. 环境DNA在长江江豚监测中的应用. 中国水产科学, 2019, 26(1): 124-132. DOI:10.3724/SP.J.1118.2019.18133] |
| [17] |
Li FL, Yang JH, Yang YN et al. Using environmental DNA metabarcoding to monitor the changes and health status of aquatic ecosystems. Environmental Monitoring in China, 2018, 34(6): 37-46. [李飞龙, 杨江华, 杨雅楠等. 环境DNA宏条形码监测水生态系统变化与健康状态. 中国环境监测, 2018, 34(6): 37-46.] |
| [18] |
Zhang WW, Xie YW, Yang JH et al. Applications and prospects of metabarcoding in environmental monitoring of phytoplankton community. Asian Journal of Ecotoxicology, 2017, 12(1): 15-24. [张宛宛, 谢玉为, 杨江华等. DNA宏条形码(metabarcoding)技术在浮游植物群落监测研究中的应用. 生态毒理学报, 2017, 12(1): 15-24. DOI:10.7524/AJE.1673-5897.20160729002] |
| [19] |
Sun JY, Yang JH, Zhang XW. Identification and biomass monitoring of zooplankton cladocera species with eDNA metabarcoding technology. Asian Journal of Ecotoxicology, 2018, 13(5): 76-86. [孙晶莹, 杨江华, 张效伟. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究. 生态毒理学报, 2018, 13(5): 76-86. DOI:10.7524/AJE.1673-5897.20180108001] |
| [20] |
Zhang LJ, Xu S, Zhao Z et al. Precision of eDNA metabarcoding technology for biodiversity monitoring of eukaryotic phytoplankton in lakes. Environmental Science, 2021, 42(2): 796-807. [张丽娟, 徐杉, 赵峥等. 环境DNA宏条形码监测湖泊真核浮游植物的精准性. 环境科学, 2021, 42(2): 796-807. DOI:10.13227/j.hjkx.202007236] |
| [21] |
Yang JH, Zhang LJ, Mu YW et al. Unsupervised biological integrity assessment by eDNA biomonitoring of multi-trophic aquatic taxa. Environment International, 2023, 175: 107950. DOI:10.1016/j.envint.2023.107950 |
| [22] |
Collins RA, Wangensteen OS, O'Gorman EJ et al. Persistence of environmental DNA in marine systems. Communications Biology, 2018(1): 185. DOI:10.1038/s42003-018-0192-6 |
| [23] |
Lance RF, Klymus KE, Richter CA et al. Experimental observations on the decay of environmental DNA from bighead and silver carps. Management of Biological Invasions, 2017, 8(3): 343-359. DOI:10.3391/mbi.2017.8.3.08 |
| [24] |
Thomsen PF, Kielgast J, Iversen LL et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 2012, 7(8): e41732. DOI:10.1371/journal.pone.0041732 |
| [25] |
Tsuji S, Ushio M, Sakurai S et al. Water temperature-dependent degradation of environmental DNA and its relation to bacterial abundance. PLoS One, 2017, 12(4): e0176608. DOI:10.1371/journal.pone.0176608 |
| [26] |
Sassoubre LM, Yamahara KM, Gardner LD et al. Quantification of environmental DNA (eDNA) shedding and decay rates for three marine fish. Environmental Science & Technology, 2016, 50(19): 10456-10464. DOI:10.1021/acs.est.6b03114 |
| [27] |
Deiner K, Altermatt F. Transport distance of invertebrate environmental DNA in a natural river. PLoS One, 2014, 9(2): e88786. DOI:10.1371/journal.pone.0088786 |
| [28] |
Willerslev E, Hansen AJ, Rønn R et al. Long-term persistence of bacterial DNA. Current Biology, 2004, 14(1): R9-R10. DOI:10.1016/j.cub.2003.12.012 |
| [29] |
Sansom BJ, Sassoubre LM. Environmental DNA (eDNA) shedding and decay rates to model freshwater mussel eDNA transport in a river. Environmental Science & Technology, 2017, 51(24): 14244-14253. DOI:10.1021/acs.est.7b05199 |
| [30] |
Jo T, Murakami H, Yamamoto S et al. Effect of water temperature and fish biomass on environmental DNA shedding, degradation, and size distribution. Ecology and Evolution, 2019, 9(3): 1135-1146. DOI:10.1002/ece3.4802 |
| [31] |
Andruszkiewicz AE, Zhang WG, Lavery AC et al. Environmental DNA shedding and decay rates from diverse animal forms and thermal regimes. Environmental DNA, 2021, 3(2): 492-514. DOI:10.1002/edn3.141 |
| [32] |
Barnes MA, Turner CR, Jerde CL et al. Environmental conditions influence eDNA persistence in aquatic systems. Environmental Science & Technology, 2014, 48(3): 1819-1827. DOI:10.1021/es404734p |
| [33] |
Mauvisseau Q, Harper LR, Sander M et al. The multiple states of environmental DNA and what is known about their persistence in aquatic environments. Environmental Science & Technology, 2022, 56(9): 5322-5333. DOI:10.1021/acs.est.1c07638 |
| [34] |
Jo T, Murakami H, Masuda R et al. Rapid degradation of longer DNA fragments enables the improved estimation of distribution and biomass using environmental DNA. Molecular Ecology Resources, 2017, 17(6): e25-e33. DOI:10.1111/1755-0998.12685 |
| [35] |
Pilliod DS, Goldberg CS, Arkle RS et al. Factors influencing detection of eDNA from a stream-dwelling amphibian. Molecular Ecology Resources, 2014, 14(1): 109-116. DOI:10.1111/1755-0998.12159 |
| [36] |
Seymour M, Durance I, Cosby BJ et al. Acidity promotes degradation of multi-species environmental DNA in lotic mesocosms. Communications Biology, 2018, 1(4): 1-8. DOI:10.1038/s42003-017-0005-3 |
| [37] |
Strickler KM, Fremier AK, Goldberg CS. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation, 2015, 183: 85-92. DOI:10.1016/j.biocon.2014.11.038 |
| [38] |
Hinlo R, Gleeson D, Lintermans M et al. Methods to maximise recovery of environmental DNA from water samples. PLoS One, 2017, 12(6): e0179251. DOI:10.1371/journal.pone.0179251 |
| [39] |
Kasai A, Takada S, Yamazaki A et al. The effect of temperature on environmental DNA degradation of Japanese eel. Fisheries Science, 2020, 86(3): 465-471. DOI:10.1007/s12562-020-01409-1 |
| [40] |
McCartin LJ, Vohsen SA, Ambrose SW et al. Temperature controls eDNA persistence across physicochemical conditions in seawater. Environmental Science & Technology, 2022, 56(12): 8629-8639. DOI:10.1021/acs.est.2c01672 |
| [41] |
Zulkefli NS, Kim KH, Hwang SJ. Effects of microbial activity and environmental parameters on the degradation of extracellular environmental DNA from a eutrophic lake. International Journal of Environmental Research and Public Health, 2019, 16(18): 3339. DOI:10.3390/ijerph16183339 |
| [42] |
Charvoz L, Apothéloz-Perret-Gentil L, Reo E et al. Monitoring newt communities in urban area using eDNA metabarcoding. PeerJ, 2021, 9: e12357. DOI:10.7717/peerj.12357 |
| [43] |
Hernandez C, Bougas B, Perreault-Payette A et al. 60 specific eDNA qPCR assays to detect invasive, threatened, and exploited freshwater vertebrates and invertebrates in Eastern Canada. Environmental DNA, 2020, 2(3): 373-386. DOI:10.1002/edn3.89 |
| [44] |
Yang JH, Zhang LJ, Mu YW et al. Small changes make big progress: A more efficient eDNA monitoring method for freshwater fish. Environmental DNA, 2023, 5(2): 363-374. DOI:10.1002/edn3.387 |
| [45] |
Yang JH, Jeppe K, Pettigrove V et al. Environmental DNA metabarcoding supporting community assessment of environmental stressors in a field-based sediment microcosm study. Environmental Science & Technology, 2018, 52(24): 14469-14479. DOI:10.1021/acs.est.8b04903 |
| [46] |
Klymus KE, Merkes CM, Allison MJ et al. Reporting the limits of detection and quantification for environmental DNA assays. Environmental DNA, 2020, 2(3): 271-282. DOI:10.1002/edn3.29 |
 2024, Vol. 36
2024, Vol. 36 


