(2: 浙江省水生生物资源养护与开发技术研究重点实验室, 湖州 313000)
(3: 中国水产科学研究院水生动物繁育与营养重点实验室, 湖州 313000)
(2: Zhejiang Provincial Key Laboratory of Aquatic Resource Conservation and Development, Huzhou 313000, P. R. China)
(3: Key Laboratory of Aquatic Animal Genetic Breeding and Nutrition, Chinese Academy of Fishery Science, Huzhou 313000, P. R. China)
微囊藻毒素(Microcystin,MC)是一些产毒微囊藻产生的单环七肽,共有100多种异构体.它是一种肝毒素,长期饮用有微囊藻毒素的水,会引发肝损伤甚至肝癌[1-3].微囊藻毒素主要由微囊藻属(Microcystis)、鱼腥藻属(Anabaena)、束丝藻属(Aphanizomenon)、念珠藻属(Nostoc)、席藻属(Phormidium)和浮丝藻属(Planktothrix)等一些藻种产生[4].产毒微囊藻的暴发,对于人类的公共安全、水生动物及牲畜造成极大的危害[5].在我国大部分富营养化水体中,铜绿微囊藻(Microcystis aeruginosa)在数量和发生频率上均占优势.
太湖是我国第三大淡水湖泊,水域面积约2428 km2.近年来,由于太湖流域经济的飞速发展,向水体中排放了大量污染物,造成水体污染和富营养化日益严重, 蓝藻水华暴发频繁[6].南太湖位于太湖南岸,一般指浙江省内沿岸部分湖面,管辖水域面积约300 km2, 湖岸线长64 km.苕溪水系和长兴水系是南太湖主要的入湖水系,这2条水系共有大钱港、新港口、小梅口、杨家浦、合溪、新塘和夹浦7个入湖口.南太湖入湖水系与太湖水域季节性水交换频繁,洪水期由入湖水系向太湖蓄水,河道枯水期,太湖水体倒灌入入湖河道[7-8].南太湖附近几个县市饮用水取水口位于这几条入湖溪流内.因太湖水倒灌,太湖蓝藻对南太湖入湖河道水质的影响巨大,严重威胁该区域供水安全[8].因此,及时监测该区域的蓝藻发生情况,对保证该区域的供水安全具有重要意义.
蓝藻水华监测,传统方法主要通过显微镜镜检法,对水体中的藻细胞计数及分类,后来又有通过藻类的16S rDNA序列分析进行藻种分类,然而这些方法不能区分产毒微囊藻及非产毒微囊藻,并在蓝藻暴发之前对其进行准确预测[9-10].实时荧光定量PCR(Real-time fluorescence quantitative PCR,qPCR)方法是一种特异性强、敏感性高、准确性好的分子检测方法,已被用于产毒微囊藻的监测[9].微囊藻毒素由一些藻毒素合成酶基因簇控制,该基因簇由mcyA~mcyJ等10个基因构成.通过检测微囊藻毒素的产毒基因mcyA、mcyB、mcyD可以用于监测水体中的产毒微囊藻细胞[11-14].mcyE基因可以用于设计种特异性引物,用于监测水体中的产生微囊藻毒素的不同产毒藻种[15-16].Ouahid等利用mcyE等藻毒素合成酶基因监测水体中的产毒微囊藻[17].Al-Tebrineh等利用mcyE和ndaF基因设计的实时荧光定量PCR方法,同时监测水体中的不同微囊藻毒素产毒藻种[15].
由于不同水域环境中水体的产毒微囊藻的种类及分布不同,本研究利用mcyE/ndaF基因为目标模版,建立实时荧光定量PCR方法,对南太湖入湖口7个水质监测点水体中的产毒微囊藻进行检测,为该区域的蓝藻水华监测、预警和水质安全提供依据.
1 材料与方法 1.1 藻种来源和培养条件实验用藻种购买于中国科学院典型培养物保藏委员会淡水藻种库,藻种编号为鱼腥藻(Anabaena sp.)FACHB-82、水华束丝藻(Aphanizomenon flos-aque)FACHB-1039、铜绿微囊藻FACHB-315、柱胞鱼腥藻(Anabaena cylindrica)FACHB-170、席藻(Phormidium sp.)FACHB-1099、水华束丝藻FACHB-245、颤藻(Oscillatoria sp.) FACHB-1053,于BG-11培养基中扩大培养,颤藻FACHB-528在SE培养基中扩大培养.培养温度为25℃,光照强度为30 μE/(m2·s),光暗比12 h :12 h.
1.2 藻细胞基因组DNA的提取 1.2.1 基因组DNA提取分别采用2种方法提取藻细胞基因组DNA:方法1按植物基因组DNA提取试剂盒(SK8203,上海生工)的操作程序,提取藻细胞基因组DNA.取藻液10 ml,12000转/min离心15 min,弃上清,取沉淀用于基因组DNA提取;方法2基因组DNA的快速提取,参考文献[18].
1.2.2 DNA浓度与纯度检测分别取藻类基因组的DNA各5 μl,加入495 μl TE缓冲液稀释,用微量核酸蛋白测定仪(Beckman coulter DU-530,美国)测定DNA浓度,测定A260和A280,DNA浓度计算公式为:[DNA]=A260×稀释倍数100×40 μg/ml.
1.3 PCR引物设计与PCR扩增根据mcyE和ndaF基因设计普通PCR和荧光定量PCR引物,具体参考文献[15],引物序列见表 1.
| 表 1 普通PCR和荧光定量PCR扩增引物 Tab.1 Primers used in conventional PCR and qPCR |
16S rDNA和mcyE/ndaF基因的PCR反应体系:10 μmol/L引物-F 1 μl,10 μmol/L引物-R 1 μl,10×PCR buffer 2 μl,10 μmol/L dNTP 2 μl,25 mmol/L MgCl2 1.5 μl,模版DNA 1 μl,Tag酶(5U/μl)0.5 μl,dd H2O 17 μl,总体积25 μl,PCR反应条件如下:94℃预变性4 min,94℃变性30 s,退火温度分别为50℃和56℃,30 s,72℃延伸30 s,30个循环,72℃,5 min.将PCR产物在3 %琼脂糖凝胶进行电泳.
1.4 荧光定量PCR在Bio-rad CFX3700仪器上进行荧光定量PCR扩增,PCR反应体系:Hotstart fluo-PCR mix(SK2956A,上海生工)10 μl,10 μmol/L cyano-real mcyE-F 0.5 μl,10 μmol/L cyano-real mcyE-R 0.5 μl,0.1 % (w/v) BSA 2 μl,10 % (w/v)PVP-30 2 μl,模版DNA 1 μl,dd H2O 4 μl,总体积20 μl,PCR反应条件如下:95℃预变性10 min,95℃变性15 s,56℃退火30 s,72℃延伸30 s,40个循环,退火过程中收集荧光.每个样品重复3次.熔解曲线的过程: 95℃ 15 s,从60℃开始升高至95℃,每30 s温度升高0.5℃,进行熔解曲线分析.
1.5 特异性检测提取不同藻种基因组DNA,进行普通PCR扩增和荧光定量PCR扩增,并以16S rDNA为对照,检测引物的特异性.
1.6 荧光定量PCR标准曲线建立分别以铜绿微囊藻FACHB-315基因组DNA进行10倍稀释,测定DNA浓度,以基因拷贝数的对数值与反应循环数(Ct)值之间建立标准曲线.基因拷贝数的计算公式为:DNA的拷贝数=6.02×1023(copies/mol)× DNA浓度(g/μl)/基因组DNA分子量,微囊藻基因组为4.70 Mb,1 bp碱基分子量为660 g/mol.
扩增效率E=10-1/S-1,S为标准曲线线性方程的斜率.
1.7 环境样品检测在南太湖入湖口的7个监测点进行采样,采样时间为2014年8月3日,水温26℃.用采水器采集表层20 cm水样.每升水样加15 ml左右鲁哥试剂.1000 ml水样直接静置沉淀24 h,用虹吸管小心抽掉上清液,余下20~25 ml沉淀物转入50 ml离心管中,4℃保存,带回实验室后利用显微镜镜检法进行藻细胞检测.用0.45 μm滤膜过滤收集藻细胞,置于-20℃条件下保存水样和藻细胞.按基因组DNA快速提取法提取DNA,并进行荧光定量PCR,检测样品中产毒微囊藻.每个样品重复3次.
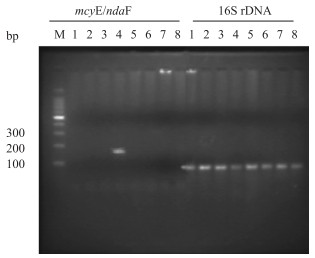
2 结果与分析 2.1 引物特异性检测提取不同产毒藻种的基因组DNA进行微囊藻毒素产毒基因mcyE/ndaF和16S rDNA基因PCR扩增,其中铜绿微囊藻FACHB-315为微囊藻毒素产毒藻种,鱼腥藻FACHB-82、颤藻FACHB-528、水华束丝藻FACHB-245、颤藻FACHB-1053、席藻FACHB-1099为鱼腥藻毒素产毒藻种,柱胞鱼腥藻FACHB-1039为柱胞藻毒素产毒藻种.仅在微囊藻毒素产毒藻种铜绿微囊藻FACHB-315扩增出125 bp左右目标条带,其他产毒藻种未扩增出目标条带,说明引物特异性较好;所有藻种的16S rDNA基因均扩增出80 bp左右的目标条带,表明藻种基因组DNA提取及PCR扩增体系无问题(图 1).对这些产毒藻种的荧光定量PCR结果表明,只有其中铜绿微囊藻FACHB-315有荧光检测信号,其余藻种均未能扩增出信号.
|
图 1 蓝藻毒素基因PCR扩增 Fig.1 PCR amplification of microcystin-producing gene (M: DNA ladder (100 bp Takara); 1鱼腥藻FACHB-82; 2颤藻FACHB-528; 3水华束丝藻FACHB-1039; 4铜绿微囊藻FACHB-3155; 5柱胞鱼腥藻FACHB-170; 6席藻FACHB-1099; 7水华束丝藻FACHB-245; 8颤藻FACHB-1053;琼脂糖凝胶含量为3 %) |
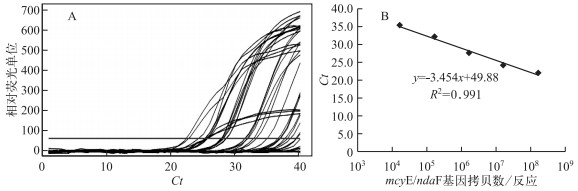
以铜绿微囊藻FACHB-315基因组DNA 10倍梯度稀释后作为荧光定量PCR模版DNA,进行qPCR测定.以标准品稀释后浓度的对数值为横坐标、Ct为纵坐标建立实时定量PCR的标准曲线,结果见图 2.利用该qPCR反应条件检测微囊藻毒素产毒藻种,标准曲线斜率为-3.454,R2 =0.991,扩增效率E=101/3.454-1= 94.6 %,定量检测区间为1.689×104~1.689×108拷贝数/μl.
|
图 2 mcyE/ndaF基因实时荧光定量PCR标准曲线 Fig.2 Standards curves of qPCR targeted with mcyE/ndaF gene (A:实时荧光定量PCR曲线;B:标准曲线) |
以铜绿微囊藻FACHB-315基因组DNA为模板进行荧光定量PCR,并进行熔解曲线分析,由图 3可见,熔解曲线平稳,峰尖且窄,熔解温度为80±0.5℃,表明该PCR扩增产物特异,无非特异性扩增.

|
图 3 熔解曲线分析 Fig.3 Melt curves analysis |
将不同浓度的铜绿微囊藻FACHB-315基因组DNA进行荧光定量PCR测定,结果如表 2所示.基因组标准品的循环数变异系数分别为0~5.62 %,表明基因组DNA的标准曲线稳定性良好,符合制备实时荧光定量PCR标准曲线的要求.
| 表 2 荧光定量PCR重复性检测 Tab.2 Repetitiveness test of qPCR |
将铜绿微囊藻FACHB-315藻细胞进行10倍系列稀释,分别取1 ml藻样,提取基因组DNA,进行qPCR,结果如表 3,可检测的藻细胞浓度最低为3.26×104,mcyE/ndaF基因拷贝数为5.415×105,藻细胞浓度与mcyE/ndaF基因的相关性良好(图 2),可以根据样品中的mcyE/ndaF基因拷贝数,利用y=-3.454x+49.88方程推测藻细胞浓度.
| 表 3 全细胞荧光定量PCR检测 Tab.3 qPCR detection with whole cell |
南太湖入湖口水质对于周边县市的饮用水安全及太湖水质有着重要影响,本研究选取南太湖入湖口7个监测点,在太湖蓝藻容易暴发的8月进行取样,利用荧光定量PCR方法检测产毒微囊藻丰度,结果见表 4,夹浦和合溪监测点的产毒微囊藻丰度最高.通过荧光定量PCR溶解曲线分析表明,这7个监测点的产毒微囊藻的熔解温度为80.5℃,表明产毒微囊藻的种类较为一致,均为铜绿微囊藻.
| 表 4 荧光定量PCR方法检测南太湖入湖口产毒微囊藻 Tab.4 Detection of microsystin-producing algae with qPCR methods |
在蓝藻水华暴发影响水质安全之前,快速准确地对产毒微囊藻进行监控是非常重要的.传统监测方法不能区分蓝藻水华中的产毒微囊藻与非产毒微囊藻.以微囊藻毒素产毒和节球藻毒素基因mcyE/ndaF为靶基因,应用实时荧光定量PCR方法可以快速准确的检测水体中的产毒微囊藻,可以同时检测多种产微囊藻毒素和节球藻素的不同藻种细胞,特异性强,灵敏度高、准确性好[15-16].本研究以微囊藻属、鱼腥藻属、柱孢藻属等8种不同藻种,以mcyE/ndaF为靶基因进行常规PCR和荧光定量PCR检测,表明引物特异性非常高.该结果与Al-Tebrineh等[15]的研究结果一致.
通过对产毒微囊藻铜绿微囊藻FACHB-315进行灵敏度和重复性检测,发现利用qPCR对铜绿微囊藻的范围为1.689×104~1.689×108拷贝数/μl,灵敏度低于Vaitomaa等的报道[19],其对于铜绿微囊藻的检测范围为6.6×102~6.6×106mcyE基因拷贝/反应体系,可能与藻基因组提取质量有关.本研究采用了一种DNA快速提取方法, 该方法可以高效快速地提取藻细胞DNA,整个过程仅需20 min,DNA提取质量经检测,纯度未达到A260/A280大于1.8,但可以用于高通量的普通PCR反应和荧光定量PCR反应.在荧光定量PCR反应中需加入2种试剂,即聚烙吡咯烷酮(PVP-30)和牛血清蛋白BSA,该试剂可以显著增强反应的灵敏度,降低反应对于模版质量的要求,与Xin等[18]的研究结果一致,对于高通量的荧光定量PCR反应十分重要.
全细胞荧光定量PCR表明,藻细胞浓度与mcyE/ndaF基因的相关性良好.因此应用该方法不仅可以用于水华发生时对铜绿微囊藻定量检测,而且可以用于低密度时对该藻进行实时监控,并及时做出预警.
本研究通过实时荧光定量PCR方法检测太湖蓝藻水华高发季节,南太湖入湖口产毒微囊藻的数量,研究结果表明,南太湖入湖口产毒微囊藻主要由铜绿微囊藻构成,在夹浦和合溪2个监测点铜绿微囊藻丰度较高,分别为(1.99±0.35)×105和(1.47±0.23)×105 cells/ml,这与杨晓红等[8]的研究结果一致.该方法可以快速准确地对产毒微囊藻进行监测、预警,对于保证该区域的饮用水安全及了解南太湖入湖水系对太湖水质的影响具有十分重要的意义.
4 结论建立了以mcyE/ndaF基因为靶基因的产毒微囊藻实时荧光定量PCR检测方法,其标准曲线方程为:y=-3.454x+49.88,斜率为-3.454,R2=0.991,扩增效率E=101/3.454-1=94.6 %,定量检测区间为1.689×104~1.689×108拷贝数/μl.对南太湖入湖口7个监测点的产毒微囊藻进行了检测,其中夹浦和合溪2个监测点的产毒微囊藻丰度最高,分别为(1.99±0.35)×105和(1.47±0.23)×105 cells/ml.7个监测点的产毒微囊藻种类较为一致,均为铜绿微囊藻.
| [1] |
张萍, 钟儒刚, 崔海信等. 微囊藻毒素诱发癌症的作用机制研究进展. 癌变·畸变·突变, 2009, 21(1): 70-73. |
| [2] |
许川, 舒为群, 曹佳. 我国水环境微囊藻毒素污染及其健康危害研究. 癌变·畸变·突变, 2007, 19(3): 202-205. |
| [3] |
陈刚, 俞顺章. 微囊藻毒素LR和黄曲霉毒素B1对肝脏促癌作用的实验研究. 癌变·畸变·突变, 1996, 8(3): 129-132. |
| [4] |
张敬平, 肖付刚, 赵晓联等. 微囊藻毒素分析检测技术. 北京: 化学工业出版社, 2009.
|
| [5] |
Meneely JP, Elliott CT. Microcystins:measuring human exposure and the impact on human health. Biomarkers, 2013, 18(8): 639-649. DOI:10.3109/1354750X.2013.841756 |
| [6] |
李大命, 孔繁翔, 于洋等. 太湖蓝藻水华期间水体和底泥中产毒微囊藻与非产毒微囊藻种群丰度研究. 环境科学学报, 2011, 31(2): 292-298. |
| [7] |
韩志萍, 邵朝纲, 张忠山等. 南太湖入湖口蓝藻生物量与TN、TP的年变化特征及相关性研究. 海洋与湖沼, 2012, 43(5): 911-918. DOI:10.11693/hyhz201205006006 |
| [8] |
杨晓红, 陈江, 周李等. 南太湖入湖口蓝藻水华时空分布规律及相关响应因子分析. 中国环境监测, 2011, 27(2): 92-96. |
| [9] |
Pearson LA, Neilan BA. The molecular genetics of cyanobacterial toxicity as a basis for monitoring water quality and public health risk. Current Opinion in Biotechnology, 2008, 19(3): 281-288. DOI:10.1016/j.copbio.2008.03.002 |
| [10] |
Sivonen K. Emerging high throughput analyses of cyanobacterial toxins and toxic cyanobacteria. Advances in Experimental Medicine and Biology, 2008, 619: 539-557. DOI:10.1007/978-0-387-75865-7 |
| [11] |
何恩奇, 钮伟民, 吴庆刚等. 产毒微囊藻mcyA基因荧光定量PCR方法的建立. 环境科学与技术, 2011, 34(12): 66-70. DOI:10.3969/j.issn.1003-6504.2011.12.014 |
| [12] |
Fortin N, Aranda-Rodriguez R, Jing H et al. Detection of microcystin-producing cyanobacteria in Missisquoi bay, Quebec, Canada, using quantitative PCR. Applied and Environmental Microbiology, 2010, 76(15): 5105-5112. DOI:10.1128/AEM.00183-10 |
| [13] |
Hautala H, Lamminmaki U, Spoof L. Quantitative PCR detection and improved sample preparation of microcystin-producing Anabaena, Microcystis and Planktothrix. Ecotoxicology and Environmental Safety, 2012, 87(1): 49-56. |
| [14] |
Via-ordorika L, Fastner J, Kurmayer R et al. Distribution of Microcystin-producing and non-microcystin-producing Microcystis sp. in European freshwater bodies:Detection of microcystins and microcystin genes in individual colonies. Systematic and Applied Microbiology, 2004, 27(5): 592-602. DOI:10.1078/0723202041748163 |
| [15] |
Al-tebrineh J, Gehringer M, Akcaalan R et al. A new quantitative PCR assay for the detection of hepatotoxigenic cyanobacteria. Toxicon, 2011, 57(4): 546-554. DOI:10.1016/j.toxicon.2010.12.018 |
| [16] |
Rantala A, Rizzi E, Castiglioni B et al. Identification of hepatotoxin-producing cyanobacteria by DNA-Chip. Environmental Microbiology, 2008, 10(3): 653-664. DOI:10.1111/emi.2008.10.issue-3 |
| [17] |
Ouahid Y, del Campo FFD. Typing of toxinogenic Microcystis from environmental samples by multiplex PCR. Applied Microbiology and Biotechnology, 2009, 85(2): 405-412. DOI:10.1007/s00253-009-2249-4 |
| [18] |
Xin ZG, Jeff PV, Melvin Q et al. High-throughput DNA extraction method suitable for PCR. Biotechniques, 2003, 34(4): 820-824. |
| [19] |
Vaitomaa J, Rantala A, Halinen K et al. Quantitative Real-time PCR for determination of microcystin synthetase E copy numbers for Microcystis and Anabaena in lakes. Applied and Environmental Microbiology, 2003, 69(12): 7289-7297. DOI:10.1128/AEM.69.12.7289-7297.2003 |
 2016, Vol. 28
2016, Vol. 28 

